TCGA助力乳头状肾细胞癌发表高分文章 ---免疫相关lncRNA预后模型分析
肾细胞癌(RCC)是全球泌尿系统肿瘤中的第三大常见恶性肿瘤,也是36种癌症中的第十六大常见癌症。乳头状肾细胞癌(pRCC)是仅次于肾透明细胞癌(ccRCC)的RCC第二常见组织学亚型,占RCC的15%-20%。据报道,pRCC与世界上广泛存在的ccRCC在临床病理特征和预后上存在较大差异。然而,相对更多的研究集中于ccRCC而不是pRCC, pRCC往往参考ccRCC的治疗策略,因此pRCC患者因缺乏特异性研究而不能达到良好的疗效。一旦患者宣称晚期pRCC,靶向药物的治疗效果明显低于ccRCC,意味着晚期pRCC的治疗效果有限且预后不良。因此,迫切需要确定pRCC的新型预后生物标志物。近期,福建医科大学附属第一医院许宁教授及其团队下载pRCC的转录组测序数据,鉴定了4个lncRNA构建预后模型,该模型与生存时间和生存状态显著相关,为治疗提供了更多的参考。相关研究以“An immune-related lncRNA prognostic model in papillary renal cell carcinoma: A lncRNA expression analysis”为题发表在genomics杂志上,杂志影响因子为6.205。
技术路线:
结果:
1、共表达网络鉴定pRCC中免疫相关lncRNA
作者从TCGA数据库中提取pRCC的原始表达数据,包括289份肿瘤样本和32份匹配的正常组织,根据分子签名数据库v4.0中的两个免疫基因集共选择了331个免疫相关mRNA,以∣cor∣>0.6和P<0.0001为临界值,通过共表达网络在pRCC中鉴定出453个免疫上调的lncRNA。
2、lncRNAs预后模型的构建
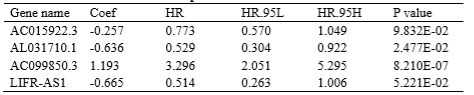
作者使用Perl脚本获得生存分析数据,将临床表达文件(lncRNA名称、患者生存时间、生存状态)与生存数据相结合,进行单因素Cox分析,以森林图的形式呈现前8位预后相关lncRNA各自的HR、95%CI及其P值,其中,7个为低风险,包括AL109936.2、AC135050.6、AC015922.3、FGF14-AS2、AL031710.1、AC107464.3和LIFR-AS1,而AC099850.3处于高风险。

通过多因素生存分析构建四个lncRNA(AC015922.3,AL031710.1,AC099850.3和LIFR-AS1)预后模型,具体信息如下表,并对274例患者进行风险评分计算:
Risk score=(0.257*AC015922.3exp)+(0.636*AL031710.1exp)+(1.193*AC099850.3exp)+(-0.665*LIFR-AS1exp)。
3、预后模型预测pRCC患者预后
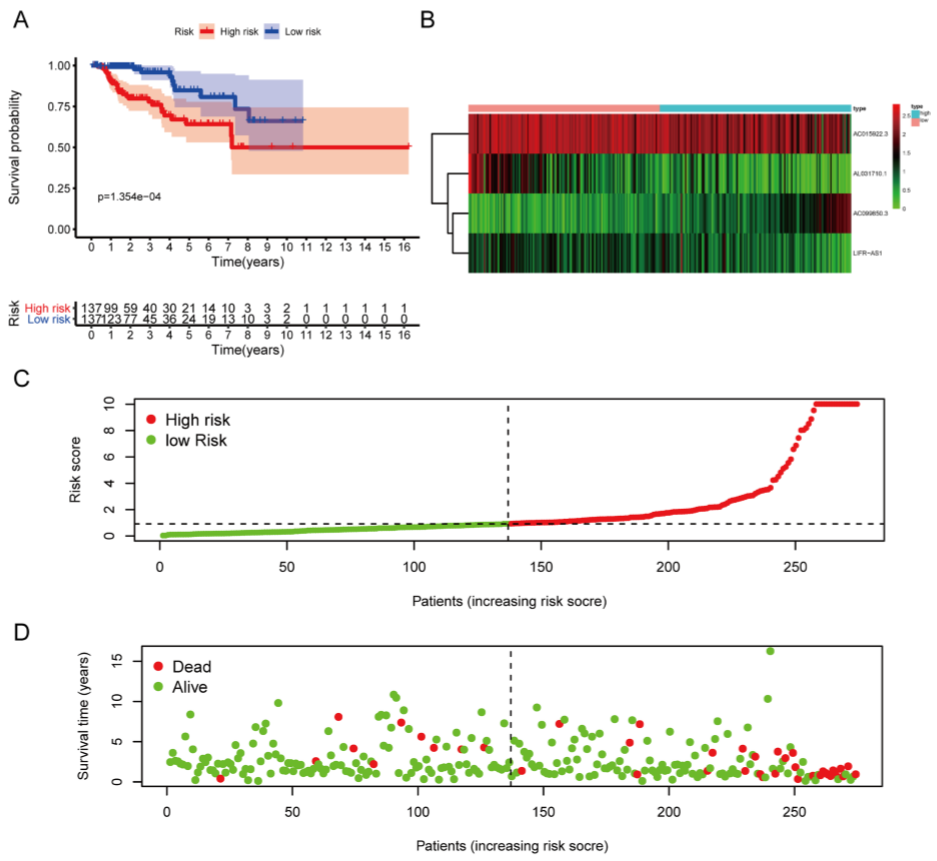
生存分析表明,这4种lncRNA的表达与pRCC的OS显著相关,KM生存分析表示低危组和高危组生存时间的差异有统计学意义(A-D)。

在274例患者中,越来越多的患者在随访期间死于pRCC,为高危组提供了64.3%的五年生存率,为低危组提供了84.9%的五年生存率。热图颜色反映了4种lncRNA在高危组和低危组中的表达水平,通过风险评分进行排序并将患者分为高危组和低危组,以风险评分0.9169为截断值,证明了OS的分布(A-D)。
4、预后模型的评估
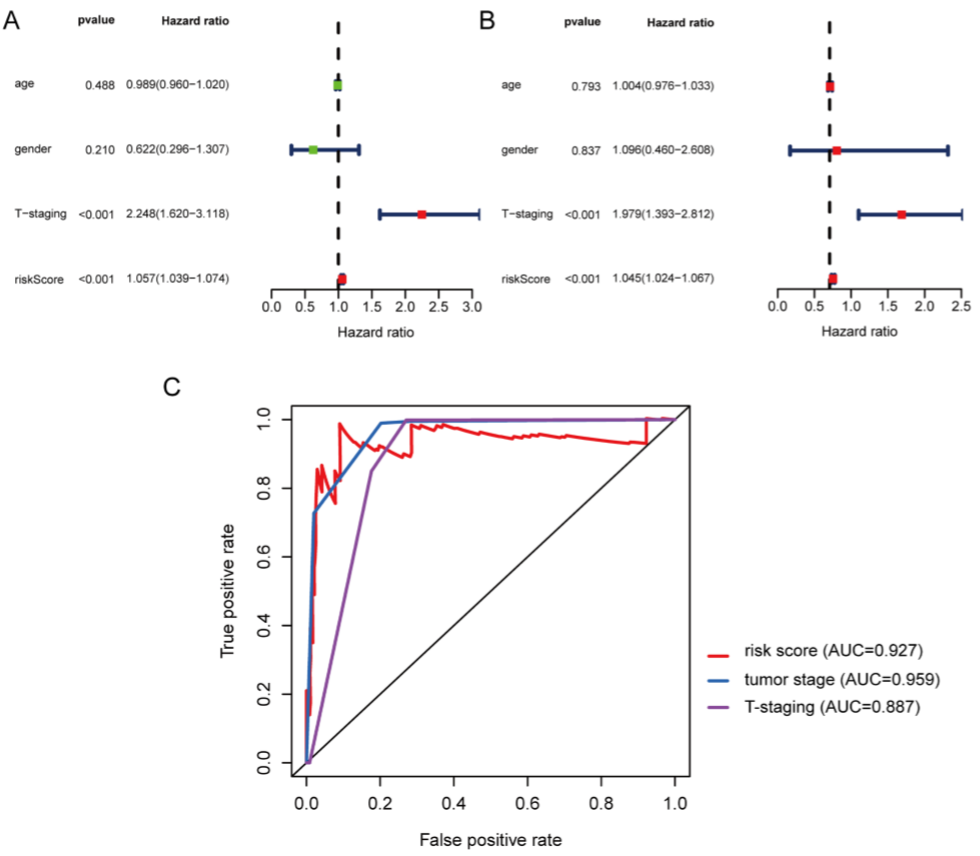
作者为了进一步验证预后模型的准确性和特异性,绘制了两个基于独立预后分析的森林图,以证明该模型是否可以在不借助其他临床特征(包括年龄、性别、生存时间、生存状态、TNM分期和肿瘤分期)的情况下成为可用的独立预后指标。单变量Cox回归分析显示,肿瘤分期、T分期和模型是pRCC的良好预后预测因素(A)。多变量Cox回归分析显示分期和预后模型可预测pRCC患者的OS(B)。同时,作者绘制了时间依赖性ROC曲线,计算了预后模型的曲线下面积(AUC)为0.927,分期的曲线下面积(AUC)为0.959,证明了lncRNA预后模型用于生存预测的竞争性能(C)。总之,四个lncRNA签名是pRCC患者极好的预后模型。
5、临床特征与4种lncRNA的相关性分析
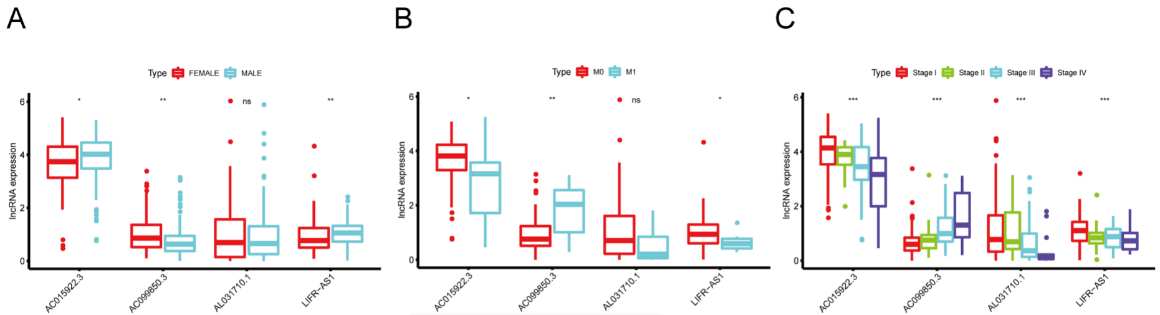
作者通过箱形图评价4种免疫相关lncRNA在3种临床特征中的表达水平,例如性别、M分期和肿瘤分期,与女性相比,AC015922.3和LIFR-AS1在男人中表达上调,在远处转移患者中表达下调,而AC099850.3在性别和远处转移患者中的表达结果相反,AL031710.1的表达与性别和远处转移无相关性(A,B)。AC015922.3、AL031710.1和LIFR-AS1的表达水平随着肿瘤分期的增加而降低,而AC099850.3随着肿瘤分期的增加而增加(C)。
6、不同基因组中高危和低危人群的差异
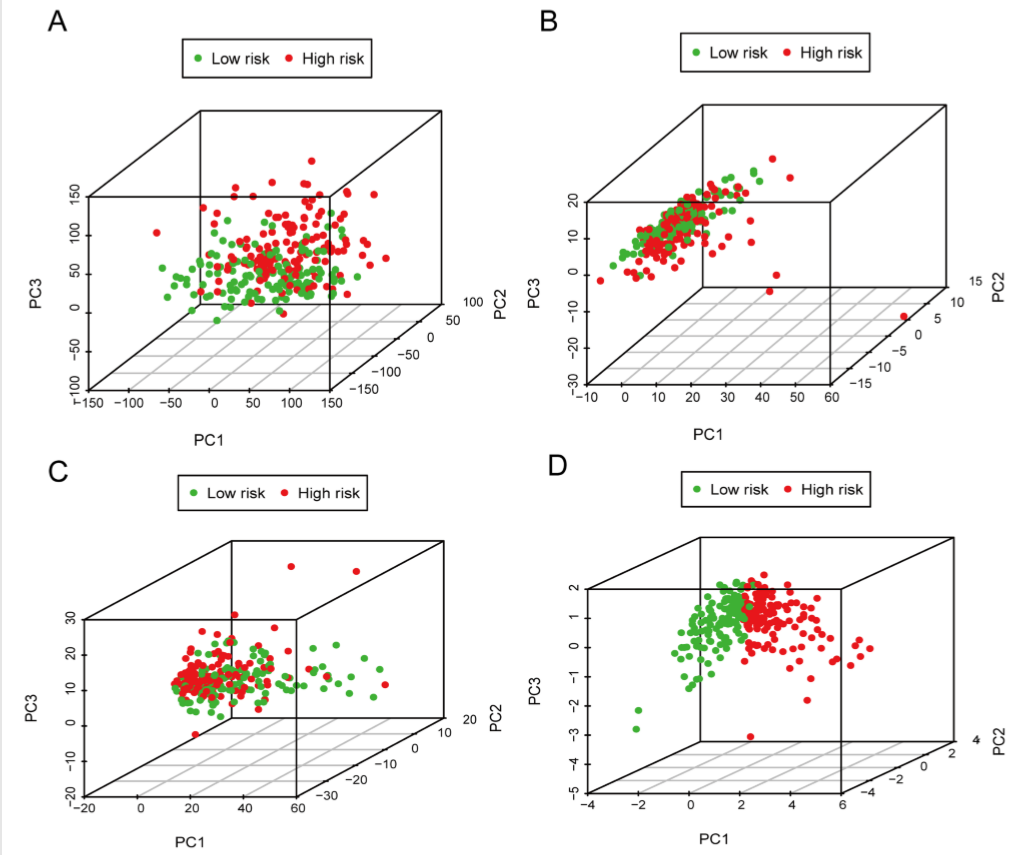
作者为了研究高危组和低危组的潜在差异使用降维技术可视化了基因表达谱,通过主成分分析(PCA)分析显示高危组和低危组在用于预后模型的所有基因(A),免疫基因(B),免疫lncRNA(C)以及4个lncRNA(D)上的差异, 红点和绿点的分布在所有基因中是混合的,而在4个lncRNA中红点和绿点之间有明显的分割线,表明构建的预后模型对识别高危和低危患者有很大帮助,也表明两组在免疫力上有显著差异。
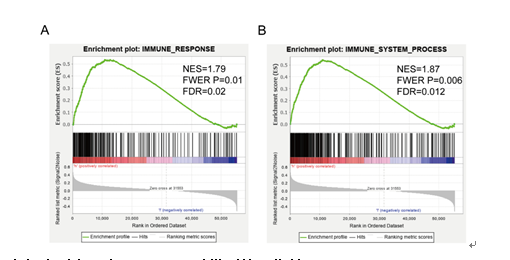
同时,通过基因集富集分析(GSEA)发现两个免疫基因集(免疫系统过程M13664,免疫反应M19817)在pRCC高危患者组中具有活跃的表现,∣FDR∣<0.25和P>0.05在两个GSEA分析中意味着高危组具有显著的免疫反应。
7、独立队列中四个lncRNA预后模型的可靠性
作者通过qRT-PCR检测66例pRCC患者中4种lncRNA的表达水平,用相同的方法构建66例pRCC患者的新风险评分公式:风险评分=(-0.545*AC015922.3exp)+(-0.452*AL031710.1exp)+(0.803*AC099850.3exp)+(-0.868*LIFR-AS1exp)。生存分析显示,在独立队列中,高危患者的预后明显差于低危患者,提示四个lncRNA预后模型是pRCC患者可靠且可重复的生存预测因子。

1、 独立队列中四个lncRNA预后模型的可靠性
作者通过qRT-PCR检测66例pRCC患者中4种lncRNA的表达水平,用相同的方法构建66例pRCC患者的新风险评分公式:风险评分=(-0.545*AC015922.3exp)+(-0.452*AL031710.1exp)+(0.803*AC099850.3exp)+(-0.868*LIFR-AS1exp)。生存分析显示,在独立队列中,高危患者的预后明显差于低危患者,提示四个lncRNA预后模型是pRCC患者可靠且可重复的生存预测因子。

结论:
作者鉴定了4个与免疫相关的lncRNA构建预后模型,该模型与生存时间和生存状态显著相关,为治疗提供了更多的参考。
参考文献:
Shao-Hao Chen, Fei Lin, Jun-Ming Zhu, et al. An immune-related lncRNA prognostic model in papillary renal cell carcinoma: a lncRNA expression analysis. Genomics. 2020.






