m6A研究又有新手段了?赶紧来了解一下吧!
RNA甲基化m6A是如今的研究热点之一,目前主要的研究思路包括差异表达的write,reader和eraser基因分析;m6A抗体检测全转录组甲基化水平;分析m6A甲基化水平变化的靶基因和下游机制研究。而在7月25日的Cell上新发表了一篇介绍不使用m6A抗体的检测mRNA m6A水平的Resource文章,小编第一时间和大家分享一下。

作者开发这一方法的前提是有研究报道了MazF能特异性的识别无修饰的ACA序列并发挥RNA酶活性在ACA之前剪切底物。但ACA序列的第一个A发生m6A甲基化之后将无法被MazF所识别,如图一所示。
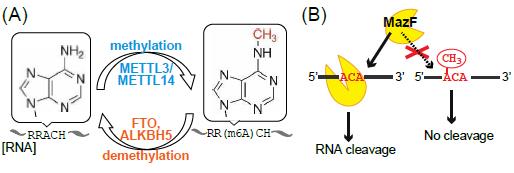
图1 MazF RNA酶作用示意图
根据这一原理,作者将纯化后的mRNA进行MazF酶处理,然后再对打断的RNA进行逆转建库测序。对于无修饰的位点,ACA处被完全剪切,测序reads正好分布于该位点上下游而且比对至该处的上下游reads数是相同的.如图2所示。而甲基化位点的reads会包含上下游的序列。作者开发了MAZTER-MINE软件包专门进行分析(图3)。
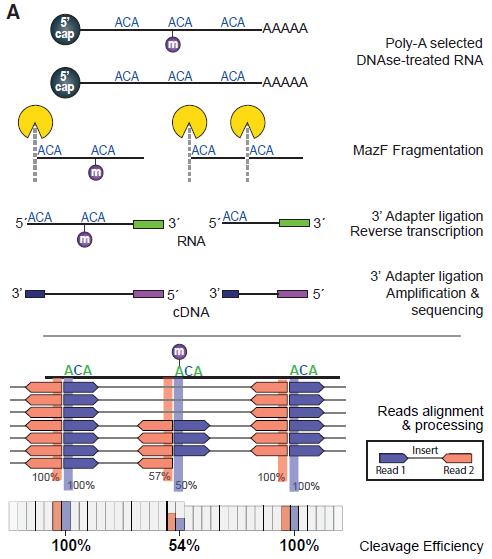
图2 MAZTER-Seq实验流程图

图3 MAZTER-MINE分析m6A示意图
接下来作者便是要验证这一新方法的可行性了。在酵母中敲除IME4的情况下,检测到的剪切效率显著高于野生型(剪切效率高代表低m6A水平),m6A抗体富集后的样品剪切效率也显著低于未富集的Input组。整体水平可靠,那检测的特异性位点是否准确呢?作者也将该方法检测到的新甲基化位点使用放射标记层析检测,发现预测的位点准确存在而且与剪切效率相符合。如图5所示。而图6中,作者则是与m6A抗体IP的方法进行了比较,也证实了这一方法的可行性。
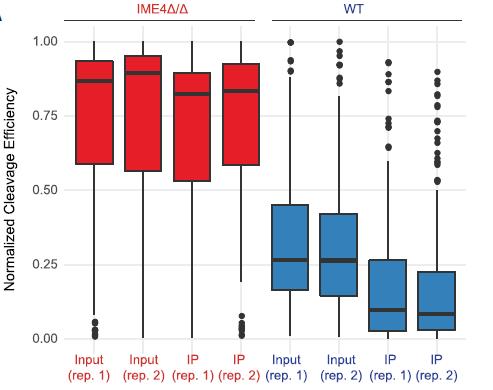

图5 MAZTER-Seq检测结果验证
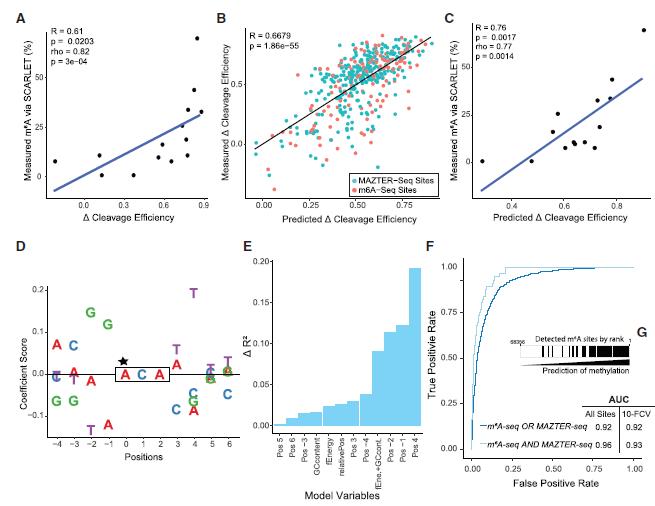
图6 MAZTER-Seq与m6A-Seq比较分析
此外,后文中作者也在大规模的CRISPR-Cas9改变m6A状态和酵母减数分裂模型中检测了MAZTER-Seq这一系统;并进一步通过这一方法检测了哺乳动物不同细胞间m6A水平的保守性;也探究了去甲基化酶FTO对整体m6A甲基化水平的影响等。这里小编主要给大家分享这一新技术,其他部分暂不过多分析了。新的技术能大大拓展我们的研究内容;对于这一技术,不知大家怎么看呢?






