研究热点m6A数据库——M6A2Target介绍
之前的文章写过m6A是近年的研究热点,虽然2020年国自然评选结果还没出,但是可以预测m6A相关中标项目数仍会上涨。如果您苦于没有相关资源,那么今天推荐的M6A2Target数据库一定不要错过。
M6A2Target数据库(http://m6a2target.canceromics.org)由中国研究团队合作完成,该数据库的文章于2020年5月11日发表在生物信息期刊Briefings in Bioinformatics(IF=8.99),通讯作者为中山大学肿瘤防治中心郑健研究员和中肿生物信息平台左志向副研究员。M6A2Target是第一个包含三类m6A甲基化酶(writers、erasers和readers,WER)对应靶基因的数据库,对目前人类和小鼠中已发表的m6A WER靶基因数据进行收集整理以及对潜在靶基因进行分析预测,从而为m6A相关研究提供候选基因和研究思路。

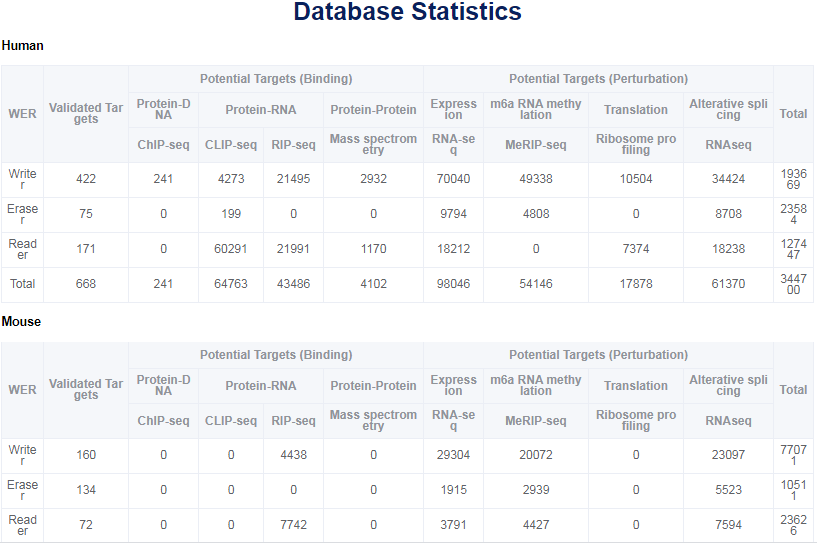
该数据库不但收集了人类与小鼠两个物种1034个已有文章报道的实验验证的靶基因,还通过高通量测序数据分析获得了大量的潜在靶基因。

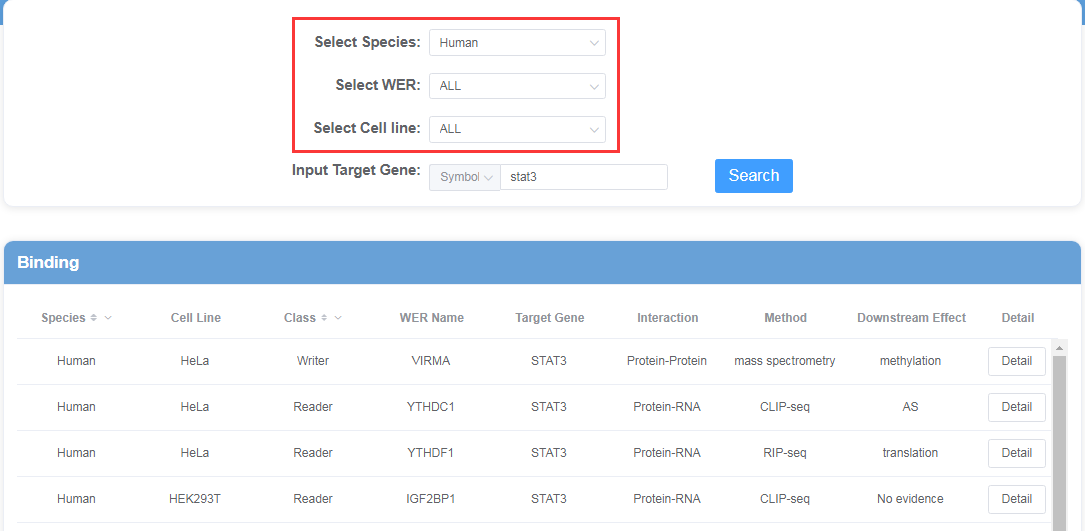
首页搜索栏可以快速查询目的基因,我们以STAT3为例进行检索。得到下图的结果,搜索结果可以选择物种(人和小鼠)、m6A的修饰酶和多种细胞系。
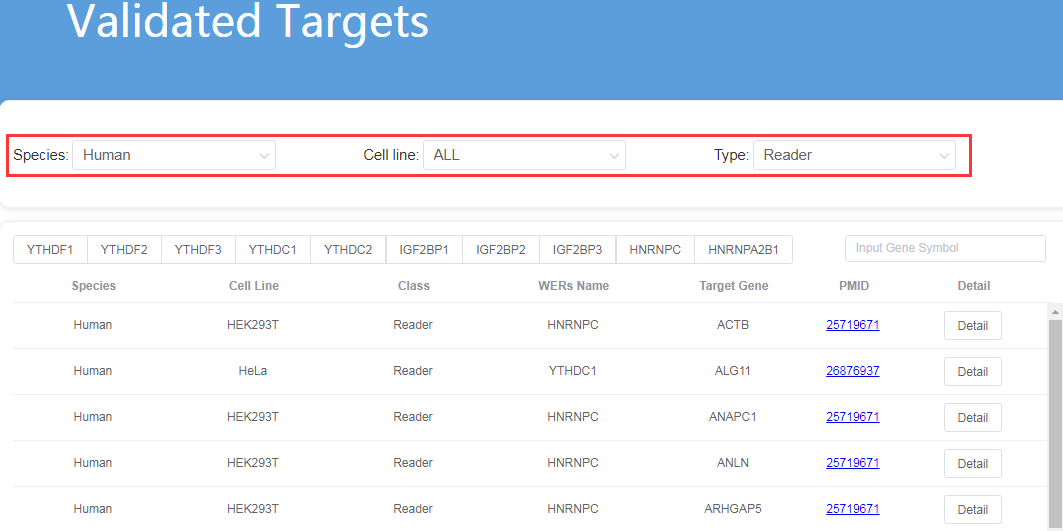
Validated Targets模块,包含了实验验证过的的m6A WER靶基因,包括免疫共沉淀、RNA pulldown、荧光素酶报告基因系统、敲低或过表达并得到qPCR的验证等,用户可以通过此模块查询是否存在已经实验验证的m6A靶基因。通过筛选物种,细胞系,WER类型或者选定不同的m6A WER则可以展示对应的m6A Tartgets,下方结果表格包含了物种、细胞系、WER类型、WER名称、对应的靶基因名以及文章PMID,同时还可以在表格右上方通过基因名筛选与此基因相关的结果。
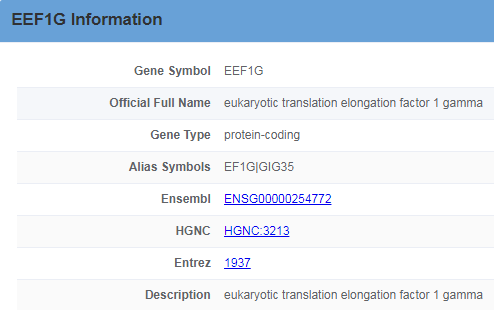
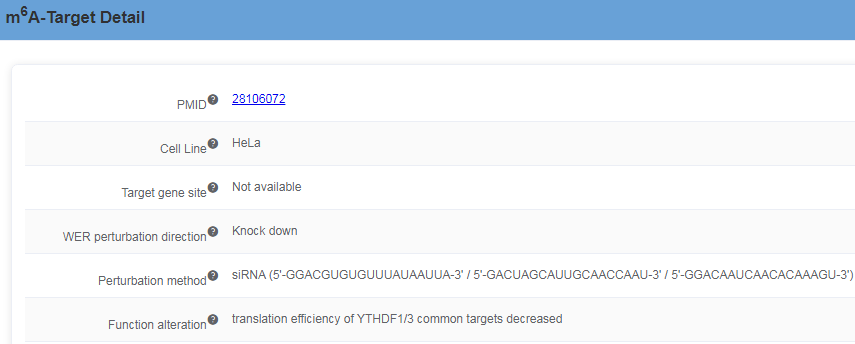
点击对应表格中对应行的Detail按钮,则可以进入详情页面。如图包含HNRNP 这个m6A及其靶基因ACTB的具体信息:如Ensembl ID,HGNC ID,Entrz ID,基因类型与基因的描述。同时还提供了各个类型基因ID对应数据库的跳转链接。同时下方还有HNRNPC与靶基因ACTB之间具体的相互作用信息,如对应文章的PMID,具体的细胞系,预测的靶基因位点,实验方法,测序方法以及对功能的影响。

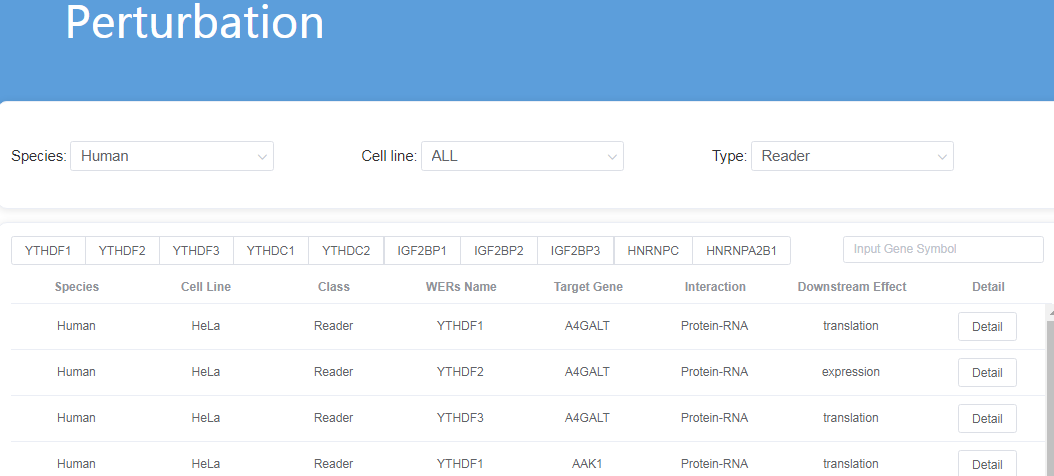
Potential Targets模块。储存了通过高通量测序数据分析预测的靶基因,包括Binding与Perturbation两部分。用于查询是否存在潜在靶基因。首先是与m6A WER有相互作用的测序数据分析整理为Binding,其中包括RIP-seq和CLIP-seq数据研究的蛋白与RNA的互作,ChIP-seq研究的蛋白与DNA互作以及通过Co-IP的方式打质谱鉴定蛋白与蛋白互作;而第二类则是收集了Ribo-seq, RNA-seq, Merip-seq等数据中由于WER绕动(比如敲除、敲降、过表达和突变等)导致下游m6A水平、基因表达水平、可变剪接或者翻译效率发生改变的WER,作为潜在靶基因,整理为Perturbation。其中Binding是直接证据,Perturbation是间接证据。结果表格除了m6A WER以及靶基因,还有对应的测序方法相互作用类型或者对下游的影响类型。

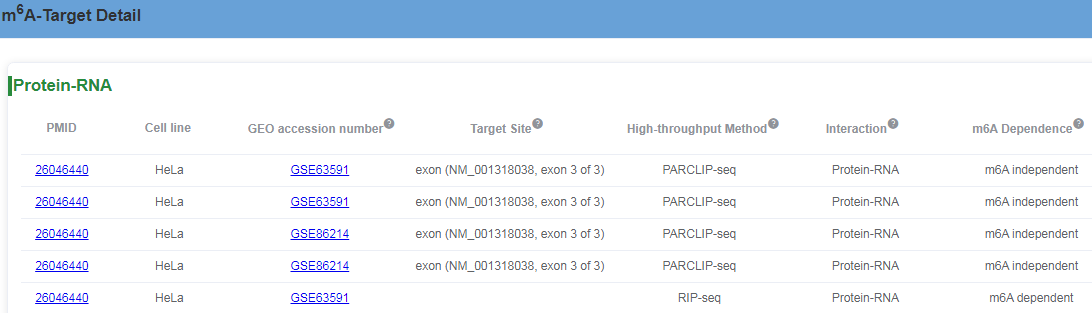
点击详情选项后,可以具体查看不同的相互作用(Protein-DNA,Protein-RNA和Protein-Protein) Target位点的具体信息,如细胞系,GEO accession number,Target位点,高通量测序方法,相互作用类型等。
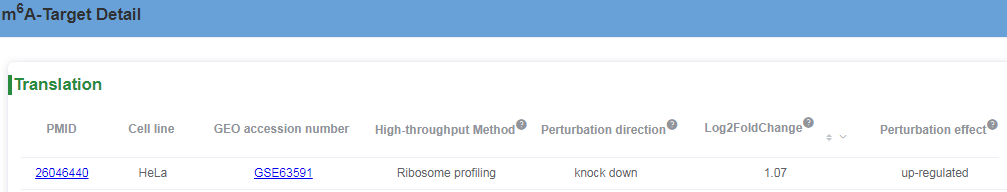
而在Perturbation的详情页面中,则可以分别查看导致下游基因表达水平、m6A甲基化水平、可变剪接和翻译效率中的靶基因具体改变情况,如具体的细胞系和样本、差异表达、差异甲基化或者差异翻译效率的具体差异结果等。
Genome Browser模块中,输入基因即可展示靶基因的各个测序数据在基因组中的位置情况。如图,输入基因SOX2,不但展示了实验验证结果在基因组中的位置情况,还展示了在RIP-seq、CLIP-seq等实验结果中SOX2与WER与SOX2相互作用的区域。同时还提供了SOX2基因上的SNP位点。

数据下载模块。最后数据库还按照物种以及分类提供了所有数据下载,方便用户进一步的分析与挖掘。