癌症相关的lncRNA/circRNA的web工具
癌症是一个主要的公共卫生问题,也是全球发病和死亡的主要原因。在过去的几十年里,RNA分子的新细胞作用被发现,已经确定癌症病理学中基因表达的动力学非常复杂。长链非编码RNA(lncRNA)作为早期筛查、诊断、预后判断和分析治疗反应的癌症生物标志物越来越受到关注。环状RNA(circRNA)是非编码癌症基因组的新成员,表现出截然不同的特性和多样的细胞功能。人们对人类lncRNA和circRNA越来越感兴趣,高通量和单细胞技术的可获得性导致了癌症相关lncRNA和circRNA数量的迅速增加。值得注意的是,癌症相关的lncRNA和circRNA可以根据其调控机制、生物学功能或临床应用分为不同的组。lncRNA和circRNA在涉及增强子、遗传变异、microRNA相互作用、转录因子(TF)和甲基化修饰的癌症相关调控机制中的作用也得到了广泛的研究。此外,癌症相关lncRNA和circRNA的新生物学功能已经出现。近年来,越来越多的证据表明lncRNA和circRNA在细胞生长、凋亡、自噬、上皮间质转化、免疫和编码能力中发挥重要功能。然而,没有专门的资源用于收集、存储和分发这些数据。大量的scRNA-seq和RNA-seq数据为数据挖掘和更深入地理解lncRNA功能创造了新的机会。开发快速、可定制的lncRNA分析和可视化方法是癌症研究的关键问题。方便高效的网络工具可以填补癌症相关lncRNA数据与向终端用户传递整合信息之间的空白,从而利用目前的lncRNA数据资源来研究人类癌症。近期,哈尔滨医科大学生物信息科学与技术学院张云鹏博士及其团队将Lnc2Cancer数据库更新为3.0版(http://www.bio-bigdata.net/lnc2cancer或者http://bio-bigdata.hrbmu.edu.cn/lnc2cancer), 提供了实验支持的lncRNA在人类癌症中的调控机制(miRNA、TF、遗传变异、甲基化和增强子)、生物学功能(细胞生长、凋亡、自噬、EMT、免疫和编码能力)以及临床应用(转移、复发、循环、耐药和预后)的信息,并开发了包括RNA和scRNA-seq表达数据在内的两个交互式网络工具平台,允许使用标准处理流水线探索lncRNA在癌症中的参与,相关研究以“Lnc2Cancer 3.0: an updated resource forexperimentally supported lncRNA/circRNA cancer associations and web tools based on RNA-seq and scRNA-seq data”为题发表在Nucleic Acids Research杂志上,杂志影响因子为11.501。
技术路线:

结果:
1. 数据扩展和预处理
作者在Pubmed数据库中筛选了>8000项研究,2018年至2020年的6500份报告涉及lncRNA,2017年至2020年的1570篇出版物涉及circRNA,并重新筛选了6500项研究(主要是2015年之前发表的报告),这些研究已被纳入Lnc2Cancer 2.0。随后,提取了实验支持的lncRNA或circRNA癌症关联信息,这些关联得到RNA干扰、体外敲低、western blot、实时定量聚合酶链反应或荧光素酶报告基因检测等相关实验的强烈支持,并提取了一些lncRNA在癌症中的高质量scRNA-seq表达数据,包括不同的癌症亚型、细胞数量、lncRNA数量、细胞系和组织,同时记录lncRNA、circRNA和癌症的详细信息。采用国际肿瘤学疾病分类第3版标准化分类注释每种癌症类型。Lnc2Cancer 3.0涉及了对15000篇已发表论文的系统综述,包括2775个人类lncRNA和220个人类癌症亚型之间关联的8297个条目,并包含了743个人类circRNA和64个人类癌症亚型之间关联的1049个条目。
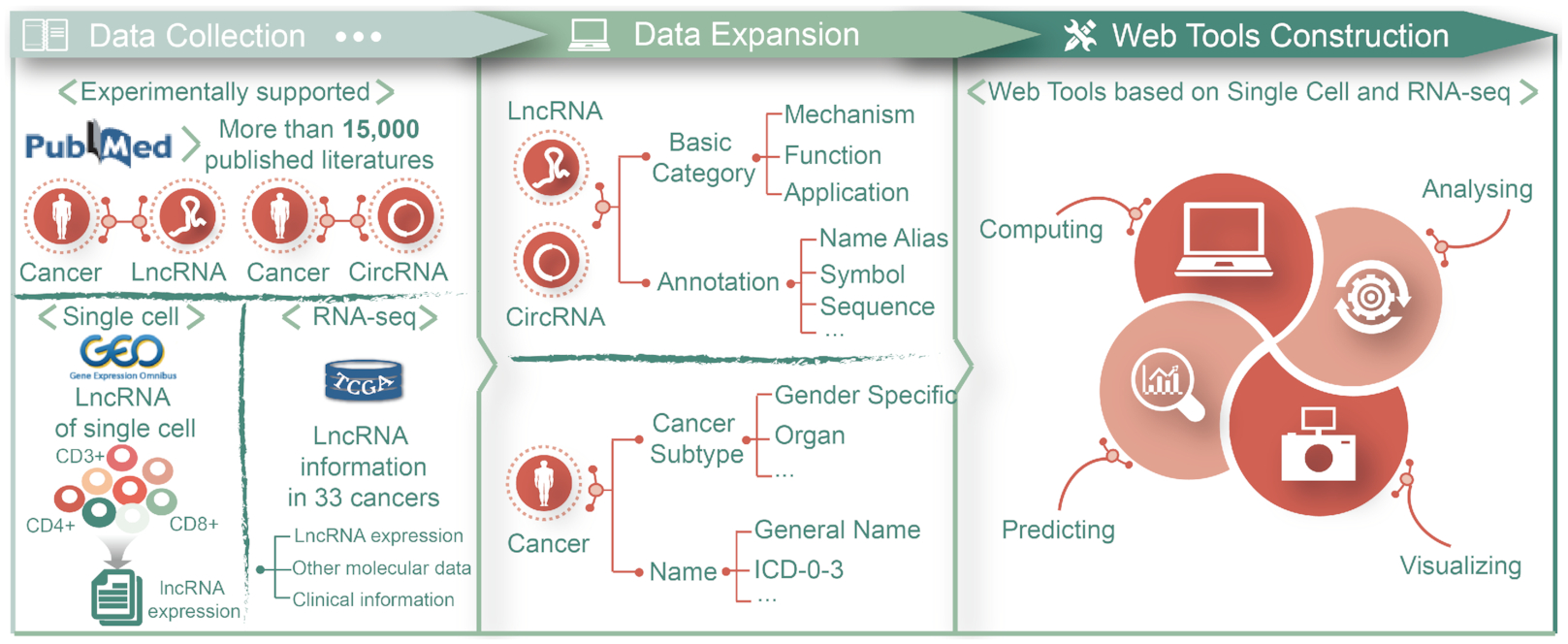
2. Lnc2Cancer 3.0的实用性工具
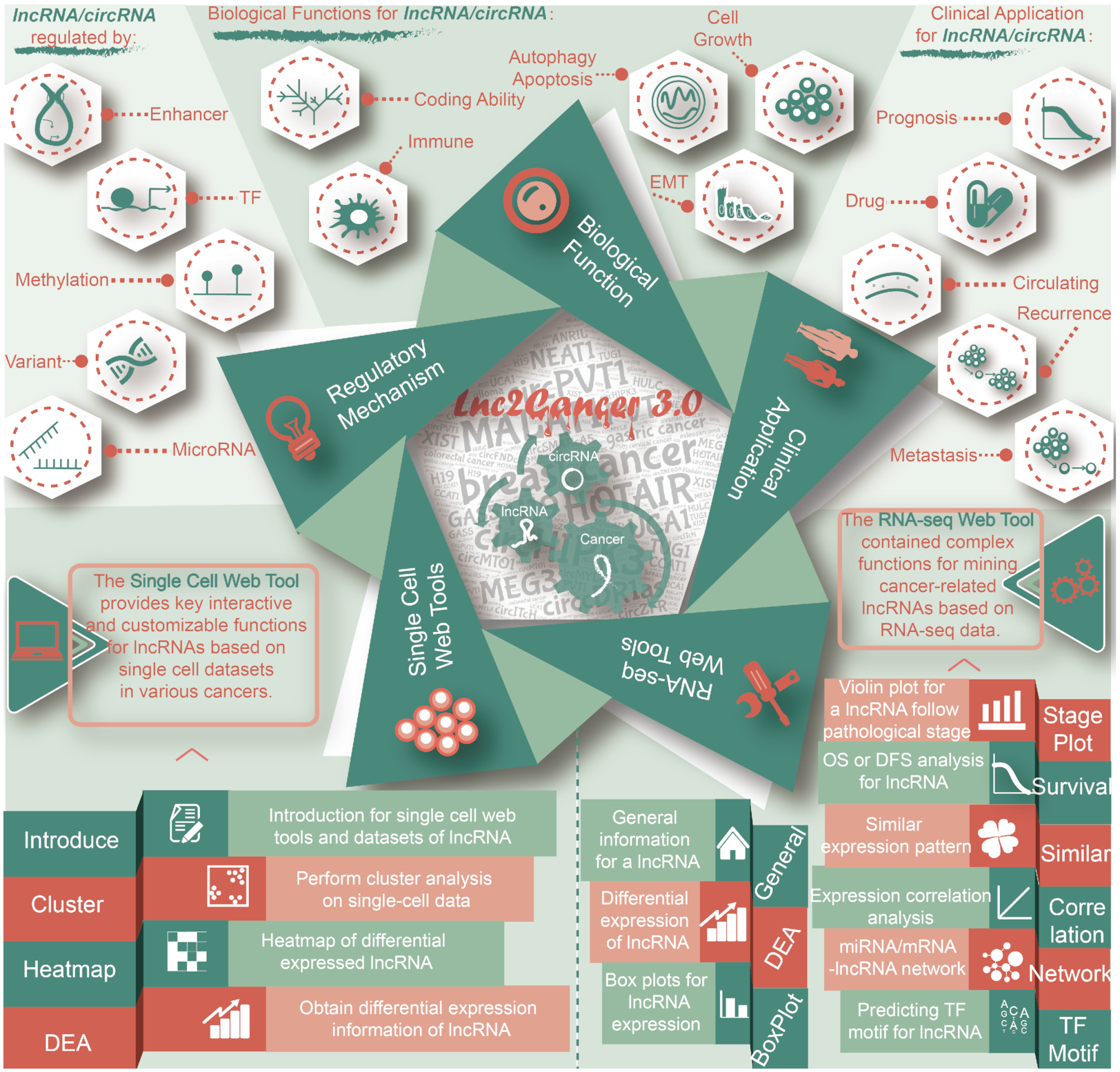
为了全面表征lncRNA和circRNA在癌症中的作用,作者手动绘制了它们的调控机制、生物学功能和临床应用,这些信息已经通过高质量的实验进行验证,分析了N6-甲基腺苷和肽的保守性,部分涉及了与免疫相关lncRNA和circRNA的免疫细胞类型,纳入了循环RNA(主要是外泌体检测到的lncRNA和circRNA)。总的来说,Lnc2Cancer 3.0提供了包括lncRNA和circRNA在癌症中的调控机制、生物学功能以及临床应用的系统流水线。在Lnc2Cancer 3.0中,作者设计了一个单细胞web工具,根据提供的单细胞数据集,可以被用来鉴定新的癌症相关的lncRNA。9个关于lncRNA表达的单细胞数据集,包括20种癌症类型和22100个细胞,收集自Gene expression Omnibus(GEO:https://www.ncbi.nlm.nih.gov/geo/)。单细胞web工具配备了三个关键功能:Cluster函数允许用户基于UMAP和t-SNE降维方法对单细胞lncRNA表达数据进行聚类分析;Heatmap功能提供了不同簇间差异表达lncRNA的热图;差异表达分析(DEA)功能使用户能够获得lncRNA的差异表达信息和小提琴图;上述所有功能均可使用R包Seurat(版本3.1.5)执行。作者从The cancer Genome Atlas(TCGA:https://portal.gdc.cancer.gov) 获得了包含lncRNA表达信息的RNA-seq数据集,建立了RNA-seq工具的9个功能:(1)一般功能允许用户构建涉及癌症相关lncRNA的低通量和高通量实验之间的串扰,提供了亚细胞定位(来自lncATLAS)、功能(来自LncBook)、基因本体注释(来自LncBook)、癌症和正常组织中的平均表达的一般信息,以及各种癌症中特定lncRNA的箱形图;(2)DEA功能使用户能够利用不同的自定义统计方法和特定癌症的阈值获得差异lncRNA表达分析和热图;(3)Boxplot功能生成带有自定义颜色的箱形图,用于比较癌症和正常样本中特定lncRNA的表达;(4)Stage Plot函数根据主要和详细的病理分期对特定lncRNA生成表达小提琴图;(5)Survival函数根据特定lncRNA的中位和分位数表达值进行总生存期或无病生存期(也称为无复发生存期)分析;(6)Similar功能使用一个输入的lncRNA和一个选择的癌症类型来识别一系列具有相似表达模式的lncRNA;(7)Correlation功能为两个癌症相关的lncRNA提供基于定制方法的lncRNA表达相关性分析,包括Pearson、Spearman和Kendall;(8)Network功能给出miRNA-lncRNA和mRNA-lncRNA共表达网络的信息;(9)TF基序功能预测特定lncRNA的TF基序,提供TF基序序列LOGO图。
3. 数据库用户使用界面
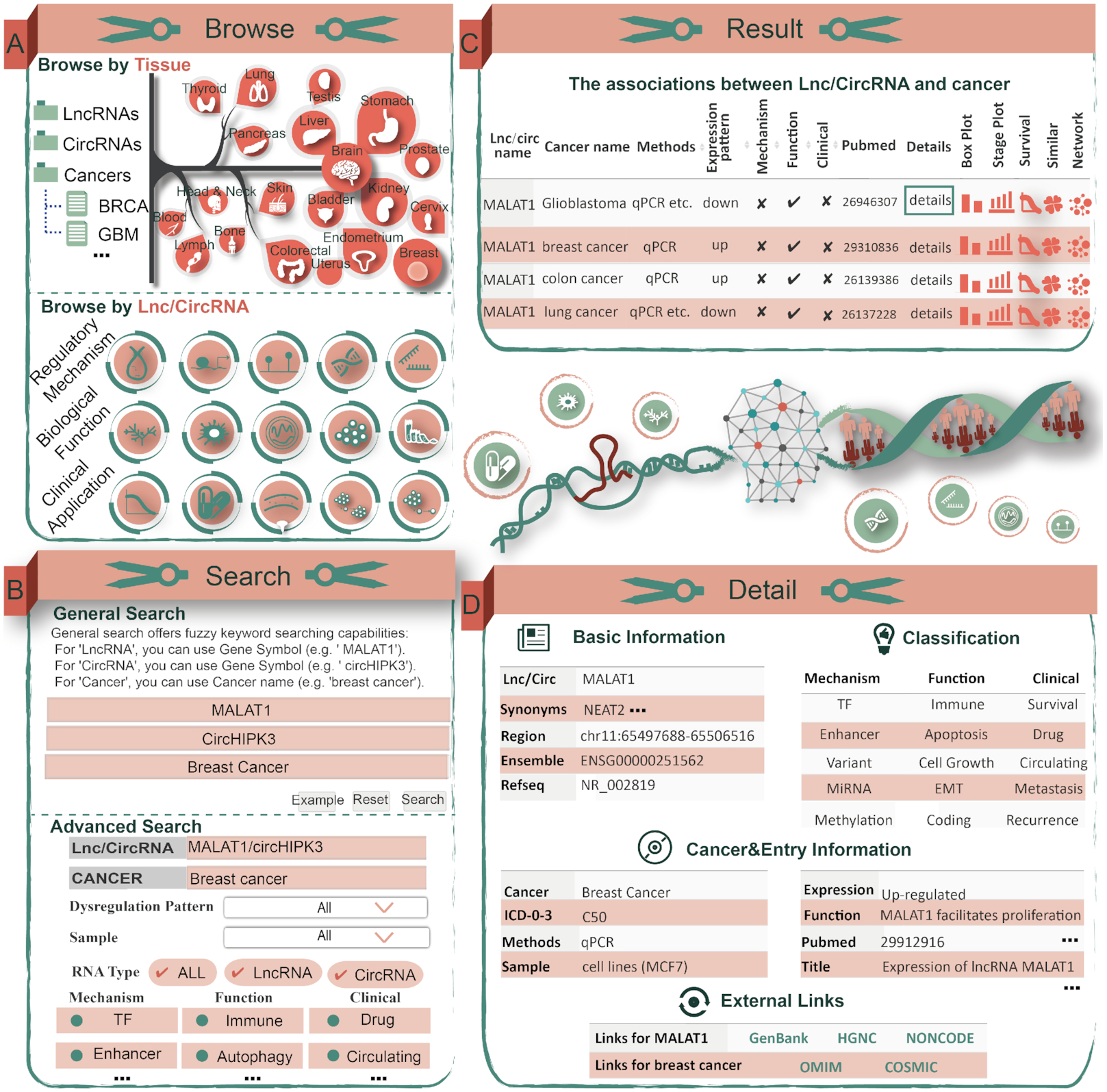
Lnc2Cancer 3.0中所有数据都使用MySQL(5.7.18版本)数据服务器进行存储和管理,web接口是在Linux和Apache平台上用JSP构建的。用户通过以下步骤查询数据库:(1)在“浏览”页面,用户可以浏览所有实验支持的lncRNA、circRNA和原发癌症组织的关联(图A)。在以癌症为中心的部分,有两种方法搜索数据。第一种包括使用人体图的解剖学分类,而第二种是癌症类型列表。在lncRNA和circRNA中心部分,用户可以浏览lncRNA和circRNA的多种调控机制、生物学功能和临床应用。(2)“搜索”页面提供“一般搜索”和“高级搜索”选项(图B-D)。利用一般的搜索,用户可以通过lncRNA和癌症名称搜索数据库。在高级搜索中,用户可以通过限制基于失调表达模式、样本、RNA类型、调节机制、生物学功能和临床应用的输出来输入更详细和系统的信息。
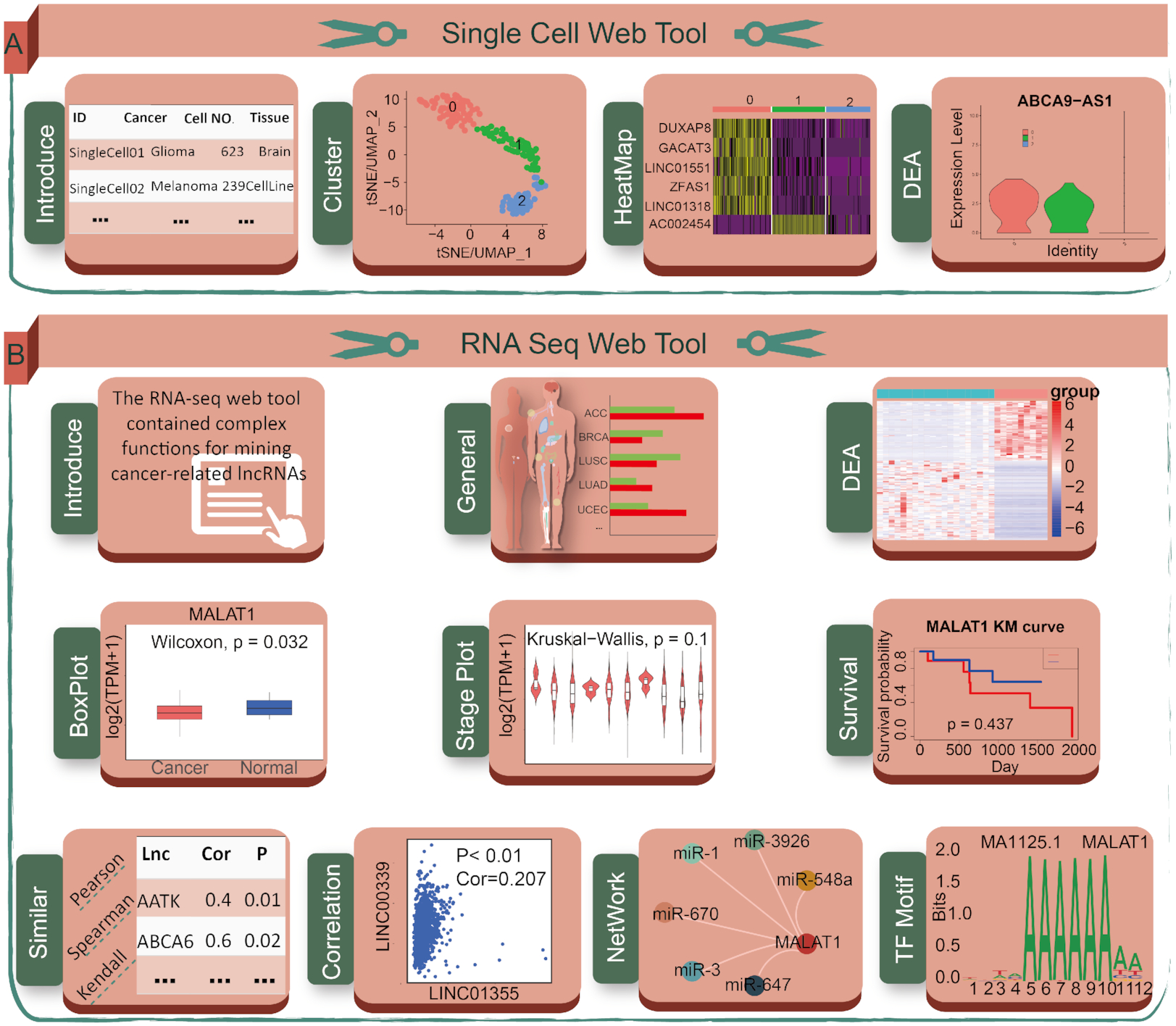
(3)从“单细胞Web工具”页面,用户可以利用交互式和可定制的功能,包括访问一般信息,基于49个单细胞数据集对lncRNA进行聚类、热图生成和差异表达分析(图A)。(4)从“RNA-seq网络工具”页面,用户可以进行复杂的功能,获得癌症相关lncRNA的详细数据,包括一般信息、差异表达分析、盒子图、小提琴样图、生存分析、相似 lncRNAs识别、关联分析、网络图构建和TF预测(图B)。(5)Lnc2Cancer 3.0是一个完全开放的资源,用户可以从“下载”页面获得所有数据。(6)从“帮助”页面,用户可以访问关于如何使用Lnc2Cancer 3.0的详细教程。
参考文献:
Yue Gao, Shipeng Shang, Shuang Guo, et al. Lnc2Cancer 3.0: an updated resource for
experimentally supported lncRNA/circRNA cancer associations and web tools based on RNA-seq and scRNA-seq data. Nucleic Acids Research. 2020.






