新的长读转录组测序技术“JAFFAL”被应用——可以检测融合基因
在癌症中,融合是重要的诊断标志物和治疗靶点。长读转录组测序可以发现全长亚型结构的融合。然而,由于较高的测序错误率,专为短读设计的融合查找算法并不奏效。在这里,作者介绍了JAFFAL,从长读转录组测序中识别融合。之后使用模拟、细胞系和来自Nanopore和PacBio的患者数据来验证JAFFAL。最终将JAFFAL应用到单细胞数据中,发现跨越三个基因的融合,证明从复杂重排中检测到的转录本。JAFFAL可在https://github.com/Oshlack/JAFFA/wiki获得。该研究于2022年1月发表于《Genome Biology》,IF:10.806。
技术路线:

主要研究结果:
1. JAFFAL管道
如图1,JAFFAL是一种新的多级管道,使用bpipe编写,其动机来自于作者从JAFFA的Direct模式中获得的方法。该流程包括以下步骤:(1)首先使用噪声耐受的长读对齐器minimap2将长读序列与参考转录组(hg38 gencode version 22)对齐,检测融合。(2)选择与融合基因一致的Reads,即与不同基因对齐的片段进行分析。(3)随后与参考基因组hg38对齐,同样使用minimap2。删除参考基因组比对后没有跨越多个基因的Reads。(4)JAFFAL利用参考基因组比对的末端位置来确定融合断点。(5)断点被分为“High Confidence”,“Low Confidence”和“Potential Trans-Splicing”。
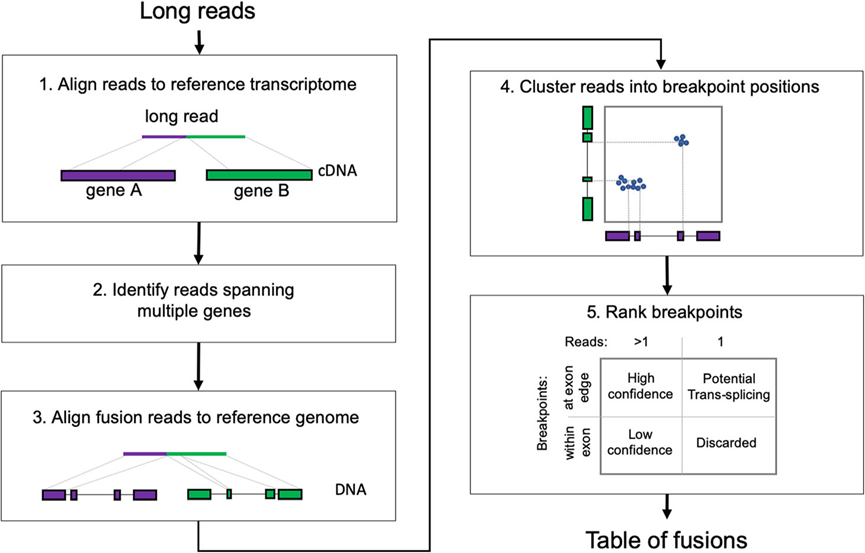
图1. 融合检测的JAFFAL管道步骤
2. JAFFAL融合排序在分离非肿瘤数据中的假阳性方面是有效的
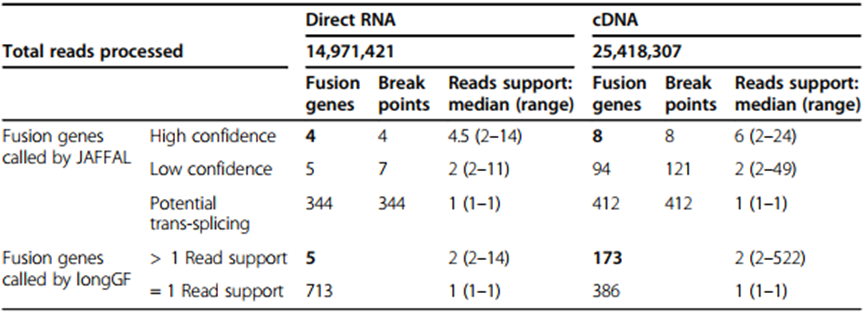
为评估JAFFAL在不同分类水平和不同测序方案的真实数据上的假阳性率,将Nanopore WGS产生的参考细胞系NA12878进行ONT融合,并称之为Direct RNA和Amplified cDNA测序。由于这是非肿瘤细胞系,融合应该很少,几乎所有报道的融合都是假阳性。如表1,对于两种方案,JAFFAL报告了很少的融合,与预期的High confidence排名。在cDNA数据中,LongGF报道了173个融合Multi-read support,JAFFAL只称8次融合为High confidence。相反,在JAFFAL对cDNA数据的Low confidence水平下报道了过多的融合(报道了94个融合),而这种过量在Direct RNA数据中未见(报告了5例融合)。综上所述,模拟和非肿瘤细胞系数据表明,被JAFFAL分类为High confidence的假阳性率较低。
表1. 从ONT直接RNA和扩增cDNA中提取非肿瘤细胞系NA12878的融合基因和断点数量
3. 利用JAFFAL可以在有噪声的长读取数据中准确地检测出模拟的融合
为模拟真实的背景,将模拟的ONT读取量与NA12878的2500万个cDNA读取量相结合。JAFFAL在无背景的ONT仿真、无背景的PacBio仿真和有背景的ONT仿真三个数据集上具有相似的融合发现灵敏度。JAFFAL检测到98%的模拟融合,当读取身份为90%或以上,覆盖率为10或以上(图2A)。在后台读取NA12878的情况下,JAFFAL的敏感性高于LongGF(图2B)。因此,利用JAFFAL可以在有噪声的长读取数据中准确地检测出模拟的融合。
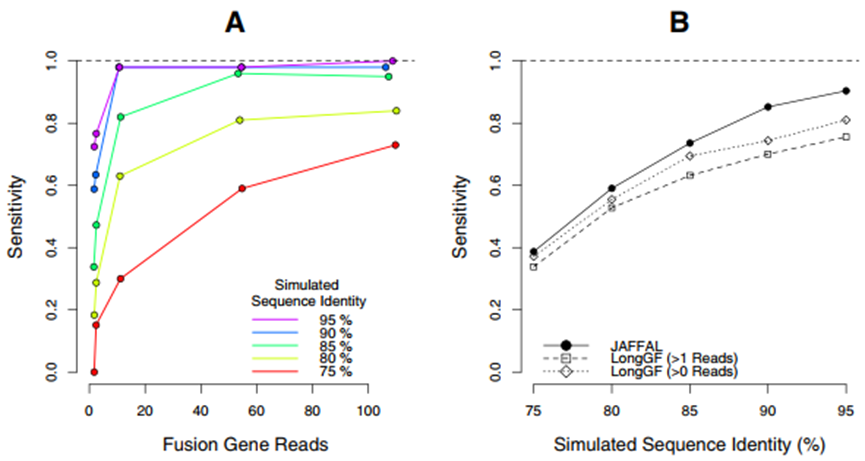
图2. 具有背景的模拟ONT数据的融合发现灵敏度
4. JAFFAL检测癌细胞系中已知的融合
为进一步证实JAFFAL的准确性,将其应用于6个癌症细胞系的公开长读转录组测序,融合之前已经使用RT-PCR和Sanger测序进行验证,或者有来自全基因组测序的正交证据表明发生了易位。JAFFAL重新发现了大约一半之前验证过的融合基因(表2)。相比LongGF,JAFFAL报告了所有数据集中相同或更多先前验证过的融合,并将其排名更高(图3A和B,表2)。仅MCF-7而言,JAFFAL之前在长读上验证的融合和报告的其他融合的数量都在短读重复的范围内,这更普遍地证明了JAFFAL的准确性和带噪声的长读数据在融合检测中的效用(图3C)。总的来说,在MCF-7 ONT细胞系数据上,JAFFAL的High confidence和Low confidence调用与之前验证的融合、匹配的短读数据中的融合以及LongGF调用的融合显示了一致性(图3D)。综合来看,这些结果表明JAFFAL是高度准确的,特别是在High confidence类别。
表2. JAFFAL和LongGF在7个长读测序数据集中重新发现了之前验证过的融合的数量
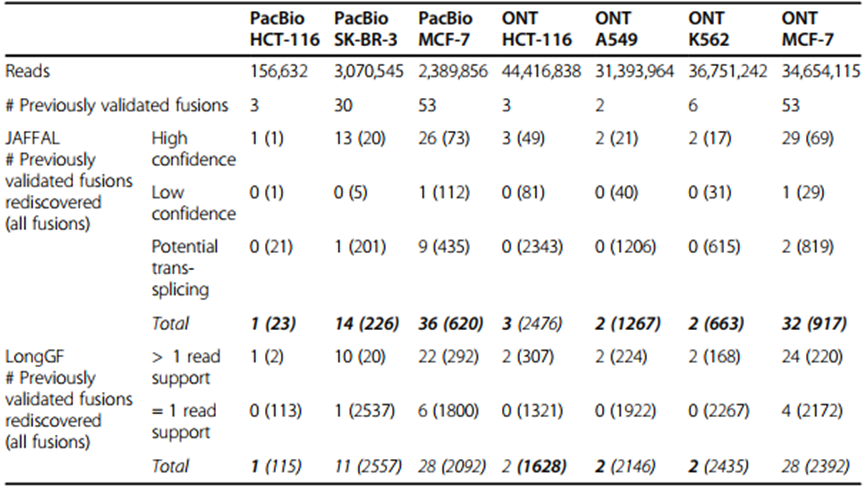
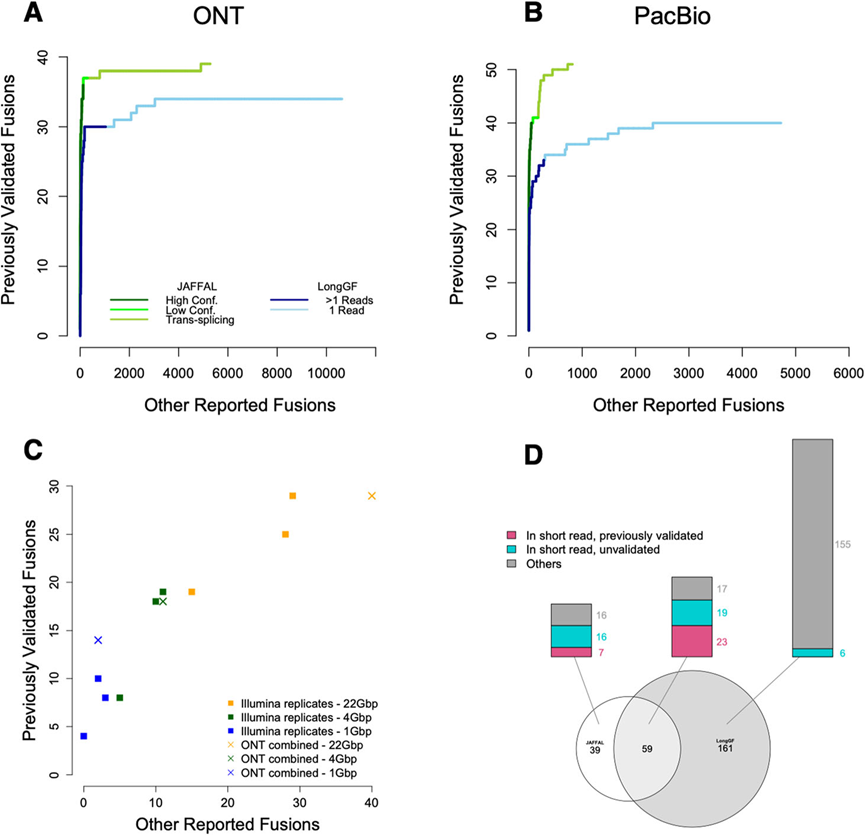
图3. JAFFAL和LongGF对癌细胞测序的比较
5. 用长读测序检测白血病中临床相关融合
接下来,将JAFFAL应用于两份来自白血病患者的样本,以评估其在现实环境中检测融合的能力。1例患者患有急性髓系白血病(AML)伴RUNX1-RUNX1T1融合。另一个病人患有B细胞急性淋巴细胞白血病(B-ALL),罕见的BCR-ABL1和IGH-CRLF2融合现象。JAFFAL检测到RUNX1-RUNX1T1和BCR-ABL1融合在他们各自的样本中17个和51个High confidence调用中排名第一和第五。与模拟和细胞系数据的结果一致,JAFFAL找到了确切的断点。
6. 单细胞水平的融合检测
利用长读测序的单细胞转录组学正在成为一种研究跨细胞类型转录多样性的强大系统。为证明在单细胞水平上调用融合的可行性,将JAFFAL应用于5个癌症细胞系混合样本的公开数据,这些样本使用ONT与10x Genomics和Illumina测序相结合进行测序。在557个细胞中,总共有1800万个ONT读取可以分配细胞条形码。正如预期的那样,根据短读数据中的基因表达,细胞聚集成五个不同的组(图4A)。在融合中,13例也出现在短读RNA测序中作为癌症细胞系百科全书CCLE的相同细胞系的一部分(图4B)。不同的融合集与每一个集群相关联,使集群注释到每一个细胞系(图4A)。一个融合,RP11-96H19.1-RP11-446 N19.1在所有五个集群中都可以看到。它不存在于CCLE中,与参考基因组中相隔264 kbp的组成基因的read-through转录一致(图4B)。在错误的细胞系簇中检测到一些融合(图4A)。然而,尽管有错误,这些结果表明JAFFAL能够在单个细胞水平上检测到融合。
7. JAFFAL检测到三种基因融合
JAFFAL发现的High confidence的三基因融合之一是在H838细胞系的单细胞测序的BMPR2-TYW5-ALS2CR11。这是由于2号染色体上2.5-Mbp区域的复杂重排导致的,并由CCLE全基因组测序发现的易位支持(图4C)。长读连接6个单元中的BMPR2-TYW5和TYW5-ALS2CR11断点。在46个细胞中,还发现了另一种截断的转录本,它将BMPR2-TYW5断点与TYW5中一个新的外显子扩展事件联系起来(图4C)。总之,作者鉴定了BMPR2-TYW5-ALS2CR11融合基因的6个不同亚型(图4C)。
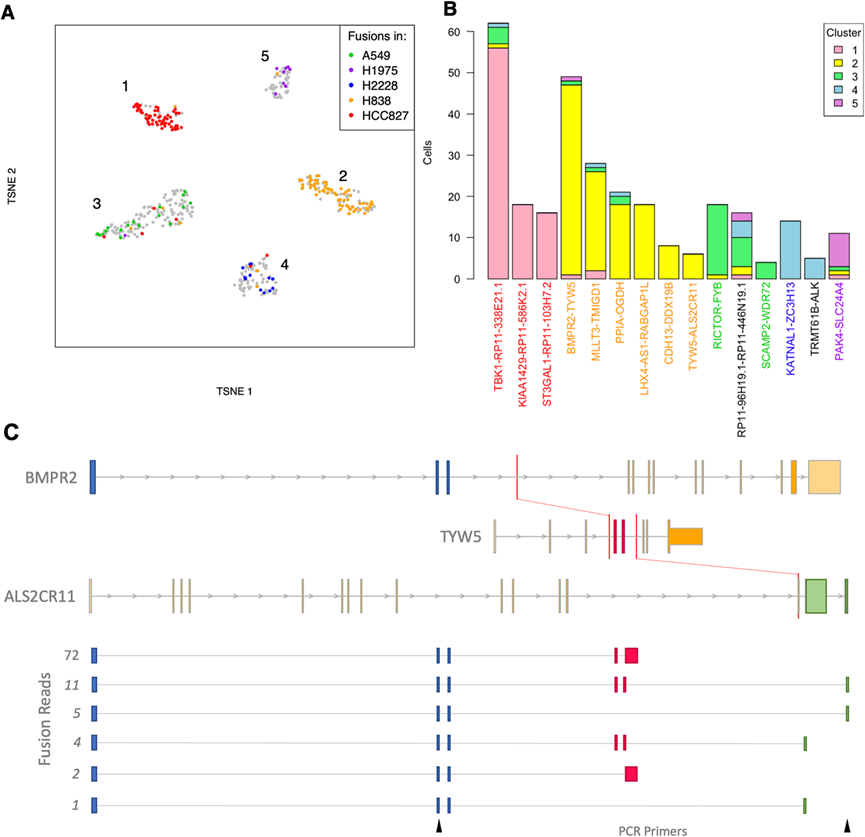
图4. 5株细胞系ONT测序中融合的检测
8. 计算资源
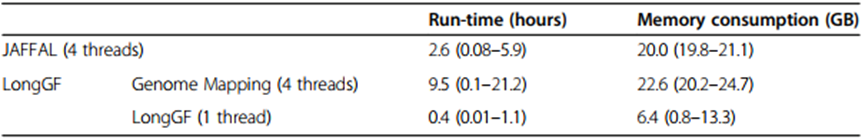
JAFFAL和LongGF所需的计算资源在一台拥有32 cores和190 GB可用内存的机器上进行了基准测试。JAFFAL在之前描述的9个健康和癌症细胞系批量数据集上分别用了不到6 h和21 GB内存完成(表3)。这些结果表明,大的长读序列的融合调用不太可能受到计算限制使用融合探测器。
表3. JAFFAL和LongGF在9个基准数据集上消耗的运行和内存的平均值和范围(括号内)
结论:
与短读相比,长读测序有许多新的优势。一项令人兴奋的发展是将长读测序技术与单细胞RNA测序技术结合使用,可以对单个细胞的全部转录组进行测序。在这里,作者证明融合可以在这些数据中被调用,为单细胞分析增加了一种额外的方式,为研究肿瘤的异质性提供了许多新的机会。






