一项多组学研究描述了食管鳞状细胞癌的新分子特征和治疗靶点
食管鳞状细胞癌(ESCC)是食管癌的主要组织学亚型,预后较差。有研究对未经治疗的食管鳞癌和配对正常癌旁组织进行了全面的转录组学、蛋白质组学、磷酸化蛋白质组学和代谢组学特征分析,以识别食管鳞癌新的分子弱点和潜在的治疗靶点。该研究发表在《Clinical and Translational Medicine》,IF:8.554。
实验方法:转录组学、蛋白质组学、磷酸化蛋白质组学和代谢组
技术路线:

主要研究结果:
1. 多层组学分析ESCC的分子图谱
我们纳入了24例食管鳞癌患者(初治病例)作为队列1,并收集了配对的肿瘤和NAT样本。我们对每个样本进行了RNA-seq、基于数据独立采集(DIA)的蛋白质组学和非靶向代谢组学研究(图1A)。由于组织样本的限制,我们只选择了3对肿瘤和NAT组织进行基于质谱的同位素标记相对和绝对定量(iTRAQ)的磷酸化蛋白质组学分析。此外,我们收集了对照小鼠和由致癌物4-NQO诱导的ESCC小鼠的食管组织,用于RNA-seq和非靶向代谢组学调查,以验证ESCC分子特征在不同物种之间的保守性(图1A)。图1B总结了ESCC患者队列1的临床特征。
mRNA的选择性剪接(AS)允许多种RNA亚型的表达,并有助于蛋白质组的复杂性。因此,我们从RNA-seq数据中检测了包括其各自亚型在内的基因表达。我们利用多层组学数据进行了无监督层次聚类和主成分分析(PCA)以确定人类食管鳞癌的分子特征。与NAT相比,ESCC组织在基因、基因亚型、蛋白、磷蛋白和代谢物水平显示出不同的标签(图1C)。由于磷酸化蛋白质组学研究只在三对样本中进行,因此磷酸化蛋白质组学数据被排除在后续的多变量分析中。接下来,我们使用cluster算法对RNA-seq、蛋白质组学和代谢组学数据进行了整合聚类,结果清楚地区分了ESCC和NAT样本(图1D)。最后,我们使用DIABLO算法35从所有患者样本的组学数据中提取分子谱,从而可以计算任意两个分子谱之间的相关性。结果显示了任意两个组学特征之间的强相关性(图1E),表明ESCC和NAT样本在不同的分子层之间存在一致的差异。

图1 ES的多层组学分析
2. 转录组和蛋白质组之间的不一致揭示了活跃的转录后和翻译后调控
下一步是确定ESCC和NAT样本的mRNA-蛋白关系差异。首先,我们进行了基因相关分析。尽管在两组样本中,mRNA和蛋白的表达水平存在显著差异,食管鳞癌组织的中位相关值为0.06,而NAT的中位相关值为0,但肿瘤组织的相关值相对于NAT的分布出现右移 (图2A),表明在肿瘤组织中有较强的正相关性。食管鳞癌和NAT组织的中位相关值分别为0.20和0.15。与NAT相比,食管鳞癌组织显示出明显的相关值分布右移 (图2B),表明在肿瘤组织中有较强的正相关性。发现肿瘤组织中大部分蛋白不仅在相应mRNA增加时上调,而且在相应mRNA不受干扰或减少时也上调(图2C)。然而,无论相应mRNA的表达模式如何,NAT组织中的大多数蛋白均下调(图2C)。此外,与NAT相比,在ESCC肿瘤中观察到更多上调的mRNA (图2C)。总之,这些结果表明,mRNA转录和翻译在食管鳞癌组织中增强,并且转录后调节过度活跃。
因此,假设在食管鳞癌肿瘤中,那些在mRNA和蛋白水平上表达增加的基因(即具有高转录和翻译活性)作为核心上游信号,导致了在食管鳞癌和NAT组织中观察到的转录后调节的差异。为了验证假设,我们纳入了在肿瘤组织中mRNA丰度增加,并且在所有组织中mRNA和相应蛋白呈正相关的基因进行分析。GO分析显示,RNA加工、RNA切片和基因表达的活性在食管鳞癌肿瘤中显著增强(图2D)。在这480个基因中,在ESCC组织中产生极高水平蛋白的基因包括SRSF10、SF3A2、CSTF2和RIF1(图2E)。在我们的ESCC肿瘤蛋白质组学数据中,SRSF10、SF3A2和CSTF2与增殖标志物Ki-67和/或PCNA呈正相关(图2F)。此外,SRSF10和SF3A2与分化标志物S100A14呈负相关(图2F)。SRSF10、SF3A2和CSTF2可能参与了食管鳞癌的发生发展。

图2转录组和蛋白之间的相关性
然后,我们假设上述的蛋白质活性参与上游信号传导,并通过改变蛋白质组影响转录后控制。因此,我们使用参与转录后和翻译后水平控制的所有蛋白质和选择的基因集进行了GSEA。正如预期的那样,在转录和翻译相关功能方面,肿瘤的富集程度显著高于NAT(图3A)。然后,我们提取了转录后和翻译后调节的关键蛋白,包括RNA转录成员、调节大量细胞RNA丰度的无义介导的mRNA衰变(NMD)途径、蛋白质翻译的关键真核起始因子(EIF)复合体和蛋白质降解的关键蛋白泛素-蛋白酶体系统(UPS)。其中,大多数在食管鳞癌和NAT组织中差异表达的蛋白质在肿瘤中表达上调(图3B-E),这与食管鳞癌组织中RNA转录、RNA衰变、蛋白质翻译和蛋白质水解的速度加快一致。
已知AS是一个关键的转录后过程,为了提供更多关于ESCC中活跃的转录后调控的证据,我们使用SpliceSeq工具分析了我们RNA-seq数据中7种常见的AS事件,包括替代受体位点(AA)、替代供体位点(AD)、替代启动子(AP)、替代终止子(AT)、外显子跳跃(ES)、互斥外显子(ME)和保留内含子(RI)。我们计算了RNA转录本的剪接百分比(PSI)值,该值揭示了这些序列在特定AS事件中剪接成转录本的效率39ESCC肿瘤和NAT之间有3883个PSI值有统计学差异的RNA转录本,在肿瘤中观察到更多的AA、AD、AT、ES和RI事件(图3F)。这与之前的研究一致,即与NAT组织相比,乳腺癌和结直肠癌组织中的AS事件增加。

图3转录、转录后、翻译和翻译后的机制研究
3. 分子途径和代谢特征的综合说明
由于mRNA和蛋白质之间的高度不一致性,我们使用蛋白质组学数据结合磷酸化蛋白质组学和代谢组学数据来描绘人食管鳞癌的多层分子改变。首先,我们利用蛋白质组学数据分析了食管鳞癌中失调的通路。食管鳞癌组织中有2890个DEPs (图S10A)。然后,我们构建了proteomaps,根据KEGG通路注释将DEPs聚类,并观察到ESCC肿瘤和NAT之间存在显著差异(图4A)。食管鳞癌肿瘤组织中剪接体、组蛋白和核糖体相关蛋白水平较高,细胞骨架蛋白水平较低。随后,我们使用这些DEPs进行了GSVA18,以量化通路激活。共有157条通路在ESCC肿瘤中被发现显著干扰 (图4B)。磷酸化蛋白质组学显示,这些磷酸盐的相应蛋白参与RNA转录、加工和代谢的通路在人食管鳞癌肿瘤中被激活 (图4C)。
随后,我们利用代谢组学数据分析了人类食管鳞癌的代谢特征。有56.50%(113/200)的代谢物在食管鳞癌组织中存在差异表达。在这些差异表达代谢物DEMs中,83.19%(94/113)表达上调(图4D),表明ESCC肿瘤具有活跃的代谢特征。然后,我们使用这113个肿瘤标志物进行MSEA,并在食管鳞癌肿瘤中观察到总共17条代谢通路有显著扰动 (图4E)。其中58.82%(10/17)的通路与氨基酸代谢有关,表明氨基酸代谢紊乱在食管鳞癌中占主导地位。

图4 ESCC分子特征的多组学分析
蛋白质组学和磷酸化蛋白质组学数据表明,与RNA转录、加工和代谢相关的通路活性增加,表明这些通路对食管鳞癌的病理生物学至关重要。下一节将详细分析这些途径。然后,我们利用代谢组学和蛋白质组学数据,对食管鳞癌中最受干扰的代谢通路精氨酸和脯氨酸代谢进行了综合分析。该网络强调指出,ESCC肿瘤选择性地表达了几种代谢酶,从而加快了氨基酸的生成(图5A)。鉴于代谢物在细胞表型确定中的重要性,我们对代表ESCC关键细胞表型的DEMs和DEPs进行了相关性分析(图5B)。这些代谢物包括5个氨基酸(半胱氨酸、谷氨酰胺、氨基丙二酸、甲基半胱氨酸和3-亚砜-丙氨酸)、2个碳水化合物(山梨醇和阿拉伯糖醇)、1个核苷酸(胞苷)、2个有机酸(苹果酸和脱氢抗坏血酸)、1个维生素(吡哆醇)和2个未分类的代谢物(组胺和2-羟基吡啶)。这些结果表明,改变的代谢物可能参与影响食管鳞癌的干性。

图5利用蛋白质组学和代谢组学数据综合分析ESCC的分子特征
4. 通过蛋白质组学和磷蛋白质组学分析,RNA转录、加工和代谢的刺激途径被揭示
如上所述,蛋白质组学和磷酸化蛋白质组学数据确定了参与RNA转录、加工和代谢的通路(图4B,C),因此提示了这些通路在ESCC恶性程度中的重要性。因此,这些通路的特征被彻底解剖。各通路中显著上调的蛋白占全部富集蛋白和差异蛋白的百分比分别为:剪接体(KEGG) 97.75%、含内含子的pre-mRNA的加工100.00%、mRNA转录形成和成熟100.00%、mRNA剪接100.00%、来自含内含子转录本的成熟mRNA的转运100.00%、延伸和处理封顶转录物100.00%和转录100.00% (图6)。而食管鳞癌肿瘤中显著增强的磷酸化位点占各通路中完全富集和差异磷酸化位点的百分比值分别为87.50%、96.15%、100.00%、95.83%、95.00%(19/20)、93.75%、100.00% (图6)。综上所述,这些发现表明这些分子通路在食管鳞癌肿瘤中受到异常刺激。
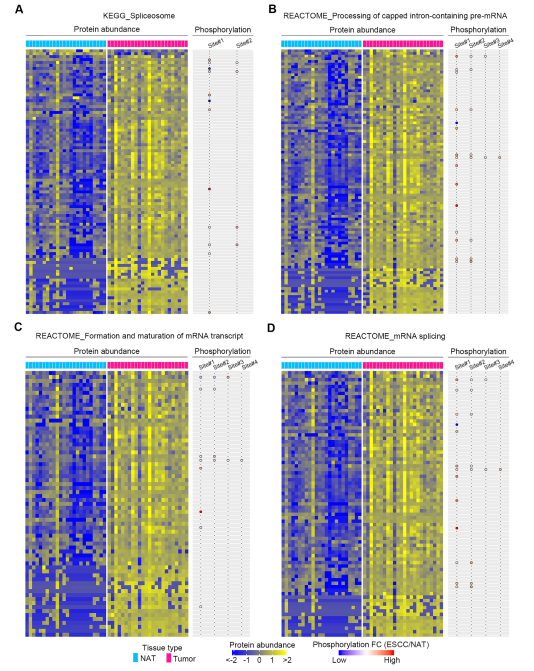
图6 通过蛋白质组学和磷蛋白质组学数据的综合分析,确定了ESCC的基本途径
5. 具有预后潜力的蛋白质标记物的鉴定
对于患者队列1的食管鳞癌组织中的DEPs,我们试图鉴定那些与患者生存密切相关并可能参与食管鳞癌进展的蛋白质。采用单因素Cox模型分析各蛋白与患者生存期的关系,共鉴定出118个具有预后潜力的蛋白。在这些蛋白质中,66个与疾病复发或死亡风险呈正相关(图7A)。KEGG通路分析显示,这118个预后蛋白主要富集在吞噬体、补体和凝血级联通路、p53信号通路、果糖和甘露糖代谢通路 (图7B)。
分析66个与HR正相关的预后蛋白在食管鳞癌患者组织中蛋白及相应mRNA水平的变化。其中23个蛋白与其对应的mRNA表现出一致的表达方向,表明这些蛋白在转录水平受到调节(图7C)。其中14个基因包括HACD2、RBM3、MRPL14、PCNP、XPNPEP3、TBC1D5、BPTF、CEP170B、PTPN13、NAA50)、SNRPF、NUP43、FBL和CHCHD2的蛋白和mRNA水平均显著上调(图7C)。在4-NQO诱导的ESCC小鼠中检测这些基因的mRNA转录水平。结果显示,与对照小鼠相比,其中RBM3、BPTF和FBL在ESCC小鼠的食管组织中显著增加(图7D),表明这三个基因在ESCC中的上调在不同物种中是保守的。接下来,我们选择了5个在ESCC肿瘤中上调最多的蛋白质,包括HACD2, RBM3, MRPL14, PCNP和XPNPEP3,以及BPTF和FBL进行进一步研究。

图7 有预后价值的蛋白质标记物的发现
6. 验证和功能分析强调FBL是一个新的不利预后的生物标志物
随后,我们纳入了食管鳞癌患者队列2 (n = 41),并进行了Western blot检测,以验证上述7种具有预后潜力的蛋白在食管鳞癌组织中的表达。结果显示,FBL, XPNPEP3和BPTF在ESCC肿瘤中相对于配对NAT显著增加(图8A)。然后我们探讨了FBL、BPTF和XPNPEP3对ESCC细胞恶性肿瘤的影响。利用两种人ESCC细胞系KYSE150和Eca109分别删除FBL、BPTF和XPNPEP3,通过对每个基因使用两个不同的引导RNA。体外研究显示,FBL的去除显著下调了细胞增殖标志物PCNA的表达,并显著抑制了ESCC细胞的生长(图8B)。体内研究显示,FBL缺失显著损害KYSE150异种移植瘤的肿瘤生长(图8C,D)。最后,我们纳入了一个ESCC患者队列3 (n = 100),对FBL进行了免疫组化染色研究,结果证实了FBL在ESCC肿瘤中的表达相对于NAT上调,以及其作为预后生物标志物的有用性(图8E)。重要的是,FBL高表达预示着ESCC患者较差的总生存期和无复发生存期(图8F)。

图8预后蛋白标记物的验证和功能分析
7. 参与ESCC发育的关键分子事件综述
如上所述,FBL在体外和体内对ESCC细胞的生长至关重要。FBL是一种核仁甲基转移酶,主要作用于rRNA和组蛋白H2A的位点特异性甲基化,从而促进核糖体组装和早期胚胎发育。因此,它在肿瘤中增加的存在表明,表观遗传调节和核糖体中的mRNA翻译可能参与了ESCC的发展。其次,作为一个重要的转录后过程,AS在ESCC肿瘤中是活跃的(图3F)。此外,在ESCC肿瘤中刺激了与RNA转录、加工和代谢相关的通路(图6)。总之,这些结果表明转录后和翻译后调控参与了食管鳞癌的发展。最后,一系列代谢物和代谢通路在食管鳞癌肿瘤中上调(图4D,E),这意味着激活的代谢是维持食管鳞癌恶性所必需的。综上所述,我们推断表观遗传、转录后、翻译后和代谢层的分子事件密切合作,促进了ESCC的发生和进展(图9)。

图9 通过多层研究描述ESCC发育过程中的分子事件的模型
8. 肿瘤抗原和潜在癌症驱动因素和关键激酶的药物注释
包括CT抗原在内的新肿瘤抗原的鉴定将为癌症免疫治疗疫苗的开发提供更多的机会通过蛋白质组学数据,我们发现四种已知的CT抗原MAGEB2、MAGEA4、MAGEA8和SPAG9在ESCC肿瘤中明显升高,与NATs相比,FC范围为1.84至223.50 (图10A)。此外,识别改变的潜在癌症驱动因子和激酶将提高我们对癌症生物学的理解,并产生新的治疗靶点。我们使用我们的蛋白质组学数据进行了研究。通过将我们的数据与之前描述的潜在癌症驱动因素列表进行比较21,我们确定了32个在ESCC组织中增加4倍以上的潜在驱动因素 (图10B)。此外,通过与PhosphoSitePlus和NetworKIN的激酶列表进行数据比较,我们发现29种已知激酶在ESCC组织中增加了4倍以上 (图10C)随后,我们利用两个药物数据库DrugBank和PKIDB 对这些在食管鳞癌中升高的潜在癌症驱动因子和激酶进行了药物注释。结果显示,21.88%(7/32)的癌症驱动因子和86.21%(25/29)的激酶具有靶向抑制剂(图10B,C)。这些抑制剂可作为食管鳞癌的新疗法。

图10 ESCC中肿瘤抗原的鉴定及潜在肿瘤驱动因子和关键激酶的药物注释
结论
通过使用多组学方法,我们破译了与食管鳞癌发生和发展有关的新分子事件,包括甲基转移酶的异常表达,转录后和翻译后过度活跃的调节,以及重塑的代谢。这些发现加深了我们对食管鳞癌病理生物学的理解,并为食管鳞癌患者的危险分层提供了新的预后生物标志物。这些发现为食管鳞癌的治疗提供了新的靶点和策略。






