CRAMdb:微生物组在动物中的组成和作用的综合数据库
动物微生物组包括细菌、病毒、真菌、古细菌和原生动物,它们几乎在动物的所有身体部位定植,并在动物宿主的健康、疾病、生长、发育和性状中发挥重要作用。近年来,动物宏基因组数据集(包括扩增子和宏基因组测序数据集)的数量迅速增加。随着高通量测序技术的快速发展,微生物组学进入蓬勃发展时期,助力解决动物健康与疾病的诊断、治疗、预防等一系列生命科学领域的重要科学难题。面对科学数据的爆炸式增长,亟需建立以动物宏微生物组为核心的开放数据库,以存储世界范围内的组学研究数据,推动微生物组学基础研究和实际转化应用的快速发展。
最近,石河子大学生命科学院团队发布了一个综合性的微生物组数据库——CRAMdb(http://www.ehbio.com/CRAMdb),该数据相关文章发表在Nucleic Acids Res期刊(IF:19.160)。CRAMdb是非人类动物精选和一致注释宏基因组的综合资源。它侧重于微生物组在各种动物物种中的组成和作用。CRAMdb的主要目标是促进动物宏基因组数据的重用,并实现跨宿主和跨表型比较。

CRAMdb数据库特别关注动物微生物组成和微生物群与动物各种表型之间的关联性。整理和分析了世界范围内516种动物、43种表型和475个项目的微生物组数据集,包含9430种细菌、278种古细菌、2216种真菌和458种病毒等共计12382种微生物。CRAMdb数据库包括两个主要模块内容: “微生物组成”和“微生物关联”。

微生物的组成旨在探索不同宿主或不同部位微生物群的分布,允许用户跨宿主或跨样本位点比较微生物的相对丰度和流行度的差异。
微生物关联旨在揭示微生物群在不同表型中的作用,用户可获得两种表型之间具有统计学差异的关联微生物,同时可以进行关联微生物的跨表型比较。
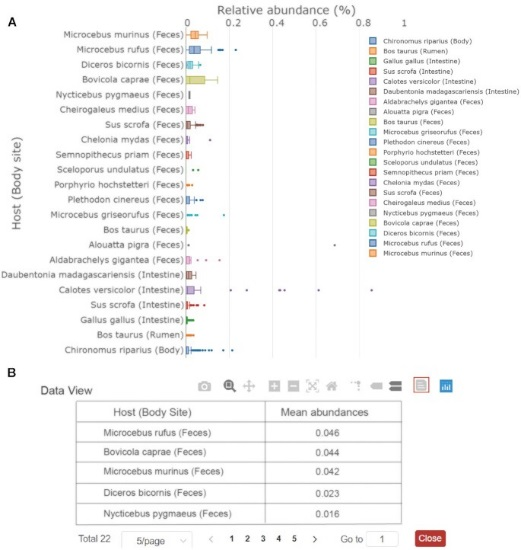
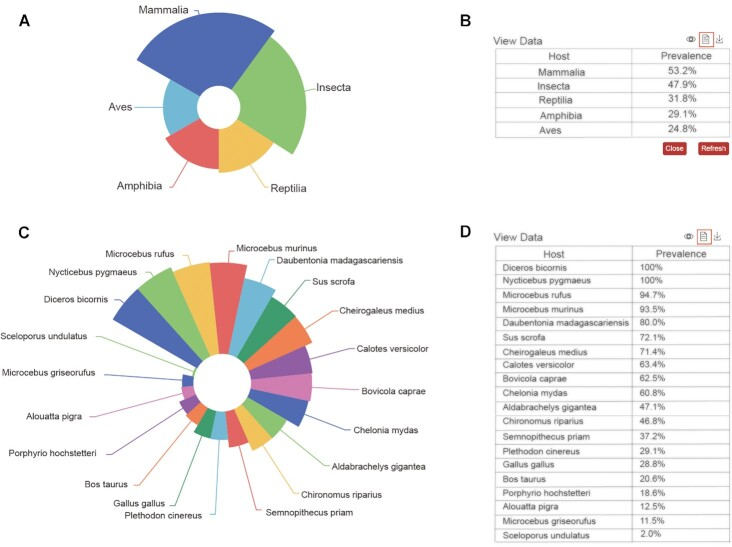
CRAMdb是一个包含与动物相关的细菌、古细菌、真菌和病毒数据的综合数据库,用于探索各种动物物种中微生物的组成和作用,将有助于生物学家从微生物的角度入手,审视动物宿主的性状,选择具有潜在重要作用的微生物开展深入分析和功能验证,为动物健康监测、疾病早期诊断、生产性能提升等方面研究提供重要数据支持。例如,利用微生物名称,用户能快速获得不同宿主和样本位点下的相对丰度和流行度、相关联的表型和对应的丰度变化;利用动物名称,用户能够快速获得融合多数据集的微生物组成、多表型之间具有统计学差异的关联微生物;利用表型名称,用户能快速获得多物种多数据集下的关联微生物并进行比较分析。






