m6A甲基化酶ZCCHC4介导的LncGHRLOS参与结直肠癌的发生
m6A修饰目前被认为是维持癌细胞稳态的RNA功能的主要驱动因素。LncRNA控制细胞增殖,在结直肠癌(CRC)的发生和发展中发挥重要作用。ZCCHC4是一种新发现的m6A甲基转移酶,其在肿瘤中的作用和机制尚未阐明。我们报道了ZCCHC4-LncRNAGHRLOS-KDM5D轴在体外和体内调控CRC的生长。我们发现ZCCHC4在原发性结直肠癌样本中表达上调,可以预测结直肠癌患者的不良临床结局。机制上,ZCCHC4下调LncRNAGHRLOS促进结直肠癌的发生。KDM5D作为LncRNAGHRLOS的下游分子,直接控制结直肠癌细胞的增殖、迁移和侵袭。本研究提示ZCCHC4轴参与结直肠癌的肿瘤发生和进展,ZCCHC4可能是这种恶性肿瘤的潜在生物标志物。本文于2024年2月发表于“Journal of Experimental & Clinical Cancer Research”(IF=11.3)上。
技术路线
结果:
1)ZCCHC4在结直肠癌组织中表达上调,预示结直肠癌预后不良
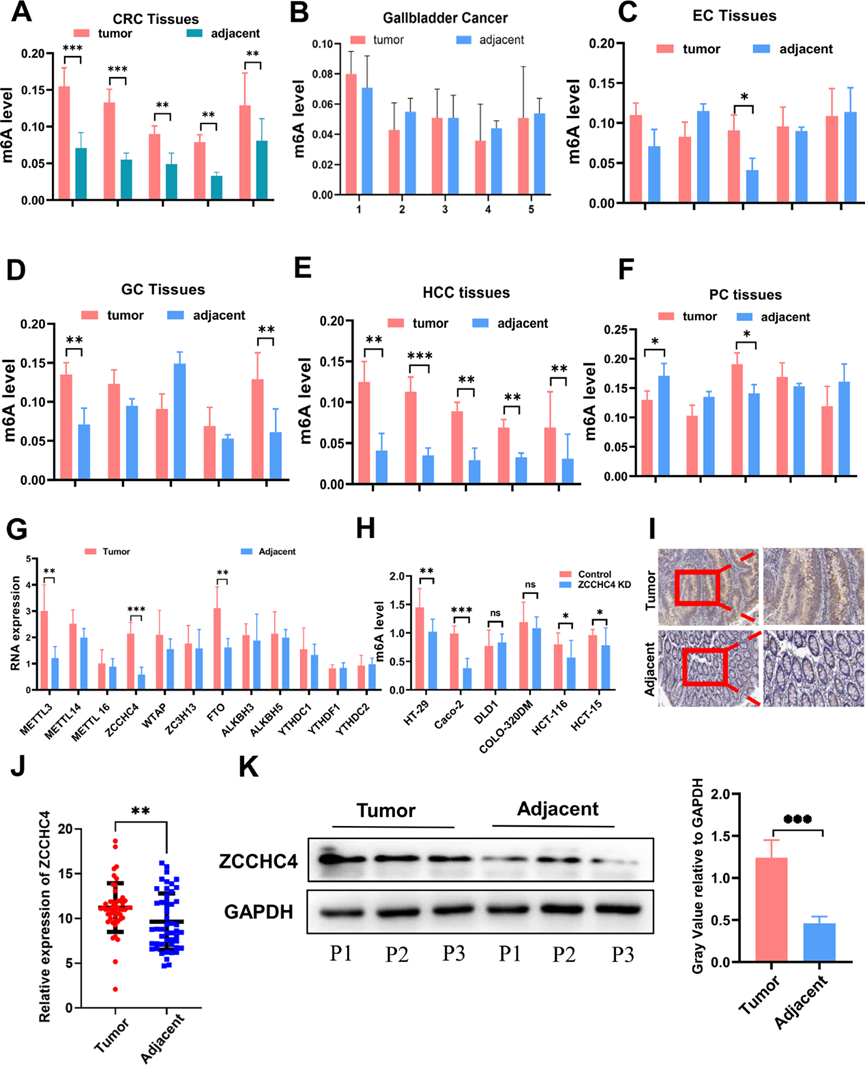
为了确定m6A在不同消化道肿瘤中的表达程度,我们随机选取5对有相邻组织样本的临床肿瘤组织,评估m6A的总体修饰水平(图1A-F)。HCC和CRC组织中m6A水平明显高于邻近正常组织。考虑到m6A在HCC中的广泛研究,我们选择CRC作为本研究的对象。同样,我们在5个CRC标本中检测到一些关键的m6A成分。最后,我们证实了METTL3、FTO和ZCCCHC4在癌组织和癌旁组织中的表达存在显著差异(图1G)。在敲除ZCCHC4后,在大多数CRC细胞系中观察到m6A表达下降(图1H)。为探讨ZCCHC4在结直肠癌中是否存在异常表达,我们采用免疫组化方法检测243例结直肠癌患者ZCCHC4的表达水平,并采用qPCR方法分析30例结直肠癌组织样本中ZCCHC4 mRNA的表达情况。免疫组化结果显示,175例患者肿瘤组织中ZCCHC4高表达,低表达患者仅占26.47%(图1I)。ZCCHC4的mRNA水平在大多数结直肠癌组织中较高(图1J)。对三对结直肠癌及其邻近癌组织进行western blot分析后也得到了类似的结果(图1K)。此外,ZCCHC4高表达的结直肠癌患者预后明显差于低表达组(图2A)。这些结果表明,ZCCHC4高表达与较差的OS相关。
2)ZCCHC4在体内和体外均与结直肠癌细胞的生物学行为有关
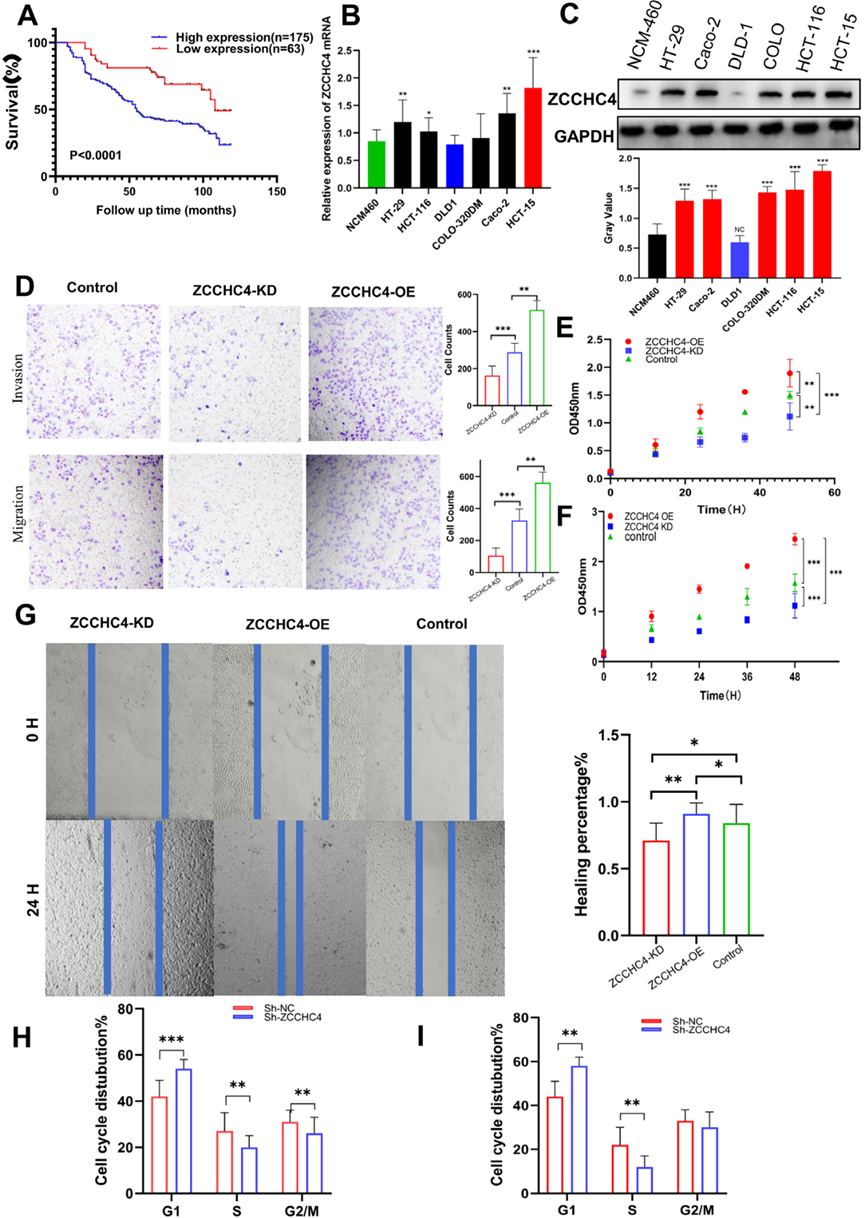
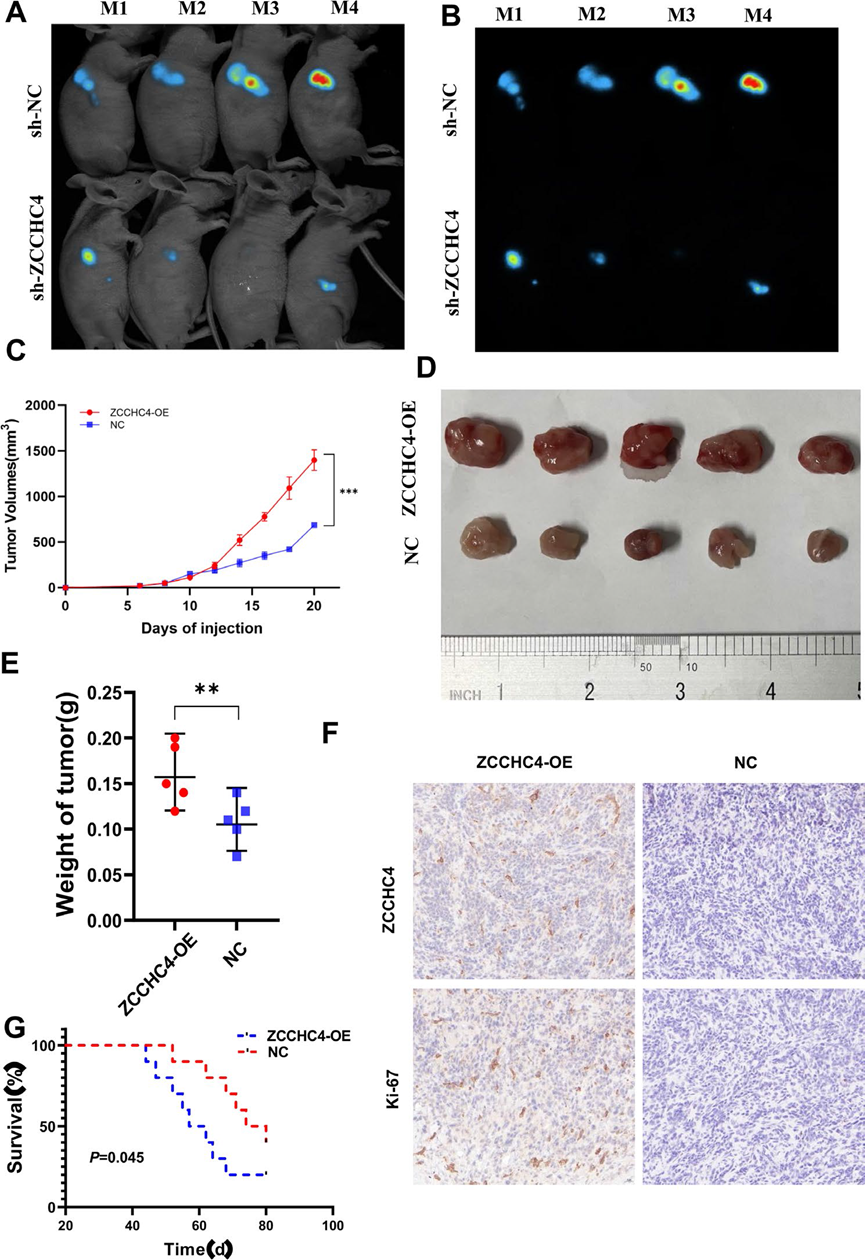
为了确定ZCCHC4在结直肠癌进展中的潜在作用,我们首先评估了ZCCHC4在结直肠癌细胞系中的表达。我们检测了几种结直肠癌细胞系中ZCCHC4的水平,发现与正常结肠上皮细胞系NCM460相比,大多数结直肠癌细胞系中ZCCHC4的mRNA和蛋白水平均升高(图2B-C)。通过Transwell细胞实验研究ZCCHC4对CRC细胞侵袭和迁移能力的影响(图2D)。结果显示,在HCT-15细胞系中,与对照组相比,下调ZCCHC4表达可显著降低CRC细胞的侵袭和迁移能力。CCK-8结果显示ZCCHC4下调可显著抑制HCT-15和HCT116细胞的增殖(图2E-F)。这些结果被伤口愈合实验证实(图2G)。通过检测细胞周期,我们观察到ZCCHC4表达下调导致HCT-116和HCT-15细胞系中处于G1期的CRC细胞比例更高(图2H-I)。鉴于ZCCHC4对维持结直肠癌细胞增殖至关重要,我们通过裸鼠异种移植模型验证了ZCCHC4在结直肠癌肿瘤发生中的体内作用。ZCCHC4高表达组CRC细胞在裸鼠体内的生长能力增强,体积增大(图3A-D),肿瘤重量增加(图3E),预后差(图3F)。免疫组化结果显示,与过表达组相比,ZCCHC4缺陷肿瘤中细胞增殖的分子标志物Ki-67减少,这表明ZCCHC4可能是CRC肿瘤发展的驱动因素(图3G)。
3)LncGHRLOS被鉴定为ZCCHC4的下游靶点
为了探索ZCCHC4在结直肠癌中的潜在靶点,我们对表达ZCCHC4和对照结直肠癌细胞系进行了RNA分析。RNA-seq结果显示,ZCCHC4过表达后,大部分转录本下调。火山图用于可视化差异表达基因的总体分布(图4A)。基因表达聚类分析用于确定不同处理下基因表达的聚类模式(图4B)。将所有基因的蛋白序列和鉴定出的差异表达基因与UniProt数据库中的蛋白序列进行比对,并根据UniProt数据库中已知的蛋白GO注释对比对结果进行标注(图4 C)。使用R中的“clusterpro消化”包富集鉴定的差异表达基因在KEGG代谢途径中(图4D)。为了鉴定在ZCCHC4下游发挥关键作用的主要靶基因,基于MeRIP-seq结果,结合RNA seq结果中ZCCHC4过表达后发生显著变化的基因,我们鉴定了11个共有基因(图4E)。通过文献检索,我们选择了CPA4、Col5A3、lncGHRLOS和CLEC18B作为ZCCHC4的潜在下游靶点(图4F)。在10对结直肠癌组织和邻近正常组织中检测这些分子的mRNA表达后,我们发现两组之间只有lncGHRLOS存在差异,我们将lncGHRLOS作为候选分子进行进一步验证(图4G-J)。我们首先利用以上50个样本的PCR结果描述了lncGHRLOS与ZCCHC4之间的相关性,结果显示两者呈负相关关系(图4K-L)。此外,在敲除ZCCHC4的表达后,我们检测到CRC细胞系中lncGHRLOS mRNA水平显著升高(图4M)。RNA pull-down和RIP实验也显示了ZCCCHC4和lncGHRLOS转录本之间的结合(图4N-O)。值得注意的是,ZCCHC4消融减缓了CRC细胞中lncGHRLOS的降解,而ZCCHC4过表达加速了lncGHRLOS转录本的降解(图4P)。因此,我们推测lncGHRLOS可能是ZCCHC4的下游效应物。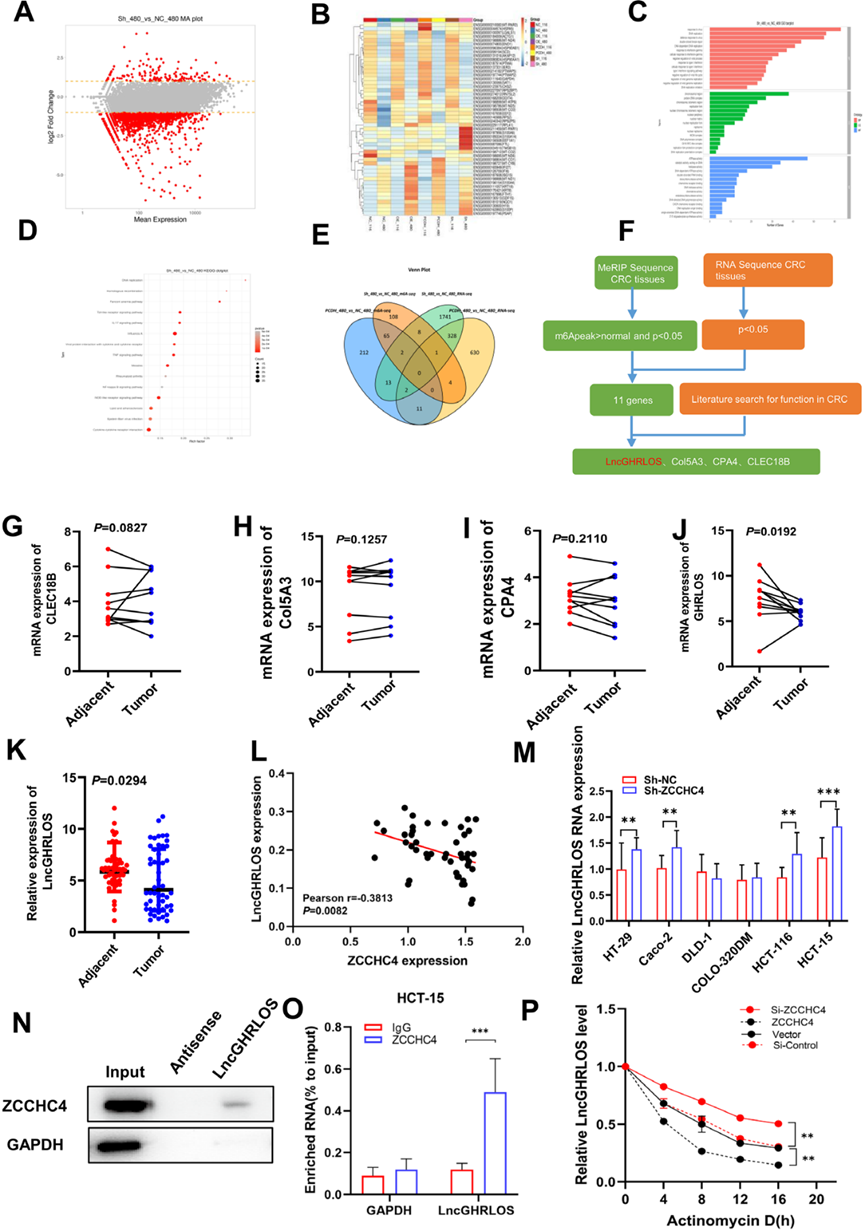
4)LncGHRLOS在体外和体内均能抑制结直肠癌的增殖、侵袭和迁移
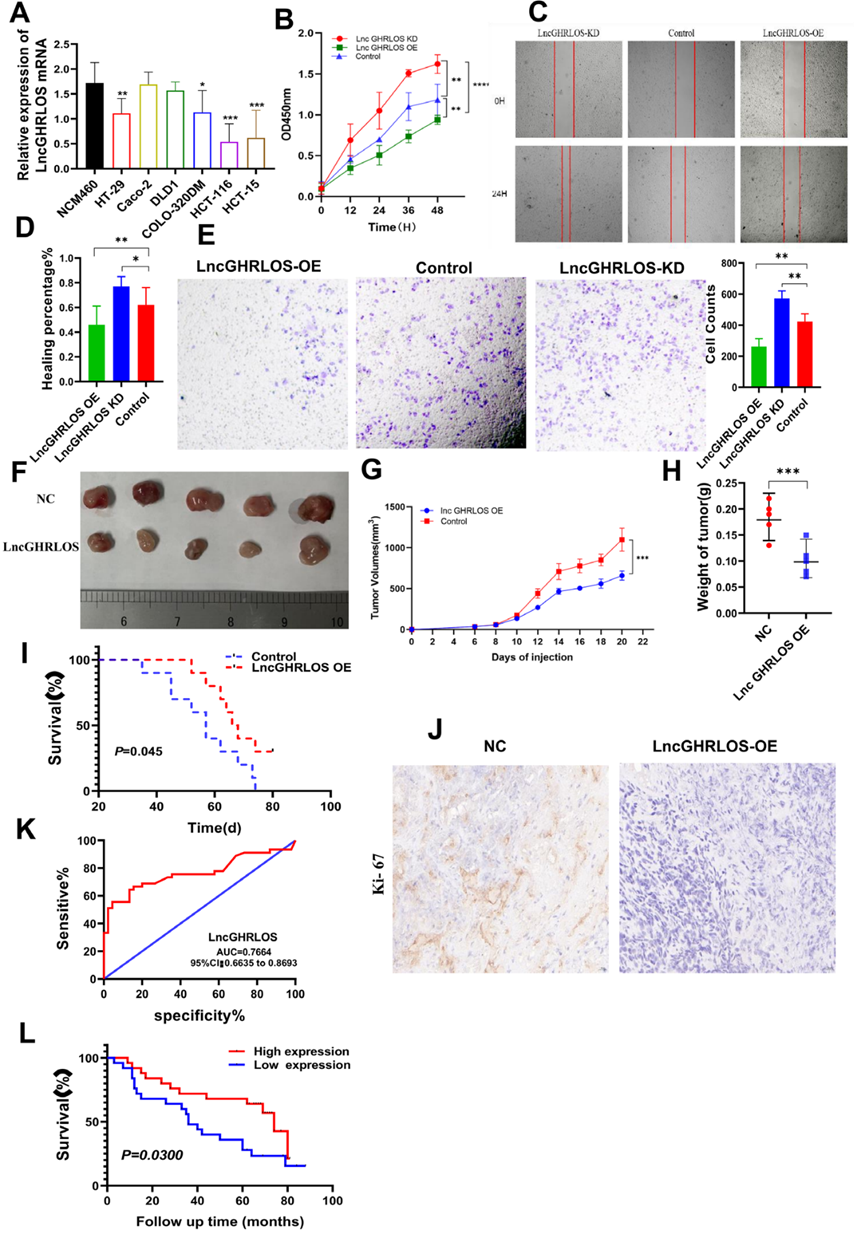
为了验证lncGHRLOS在CRC中的独特作用,我们首先分析了CRC细胞系中的lncGHRLOS (图5A)。CCK-8活性实验表明,沉默lncGHRLOS可有效增强CRC细胞的增殖能力,而增强lncGHRLOS的表达则会产生相反的生物学效应(图5B)。通过伤口愈合实验来确定细胞的迁移能力(图5C-D)。我们的研究结果显示,过表达lncGHRLOS后,CRC细胞的迁移能力与普通CRC细胞相比明显降低,与低表达lncGHRLOS组相比存在显著差异(图5E)。随后,我们用稳定过表达lncGHRLOS的CRC细胞接种裸鼠,研究lncGHRLOS对致瘤性的影响。值得注意的是,lncGHRLOS的过表达减少了裸鼠CRC细胞的生长,导致肿瘤体积变小(图5F-G),肿瘤重量减轻(图5H),预后更好(图5I)。免疫组化结果显示,与相应的对照组相比,lncGHRLOS过表达的肿瘤中细胞增殖的分子标志物Ki-67下调(图5J)。对结直肠癌患者样本进行PCR检测和ROC分析显示,lncGHRLOS在临床结直肠癌诊断中具有显著的分化作用(图5K)。根据随访结果绘制患者生存曲线。结果显示,lncGHRLOS高表达组患者生存期较长(图5L)。
5)ZCCHC44和LncGHRLOS共同调控KDM5D的表达
通过RNA下拉实验和质谱分析研究LncGHRLOS的潜在下游靶点。质谱分析表明,KDM5D可能在LncGHRLOS的m6A修饰中发挥作用(图6A)。我们利用PCR结果的中位数将临床标本中KDM5D高表达和低表达的患者分组,发现KDM5D在结直肠癌中表达较低,其低表达在结直肠癌患者中预后较差(图6B)。ELISA结果也显示KDM5D有可能成为CRC的标志物(图6C)。选择结直肠癌及癌旁组织进行PCR实验,确定KDM5D mRNA水平(图6 D)。此外,在结直肠癌中,LncGHRLOS与KDM5D mRNA表达呈显著正相关(图6E)。细胞系验证实验证实,敲低LncGHRLOS后,KDM5D的表达显著降低(图6F-H)。重要的是,我们评估了敲低和过表达KDM5D后LncGHRLOS mRNA的稳定性。结果发现,放线菌素D处理下,过表达KDM5D后,LncGHRLOS的mRNA稳定性显著提高,而敲低KDM5D后,LncGHRLOS的mRNA稳定性显著降低(图6I)。在许多CRC细胞系中观察到KDM5D mRNA的表达显著降低(图6J)。接下来,我们使用伤口愈合实验(图6K)、CCK-8实验(图6L)、transwell实验(图6M)和流式细胞术(图6N)来验证KDM5D敲低和过表达对结直肠癌细胞生物学行为的影响。我们发现KDM5D的高表达降低了HCT-116细胞的增殖和迁移能力。细胞周期在G0/G1期停滞。此外,我们进行了体内实验来阐明KDM5D对CRC的影响。裸鼠成瘤实验显示,KDM5D低表达组CRC细胞在裸鼠体内的生长能力增强,体积增大(图6O-P),肿瘤重量增大(图6Q), Ki-67指数增大(图6S)。但与以往实验不同的是,两组小鼠的生存时间没有显著差异(图6R)。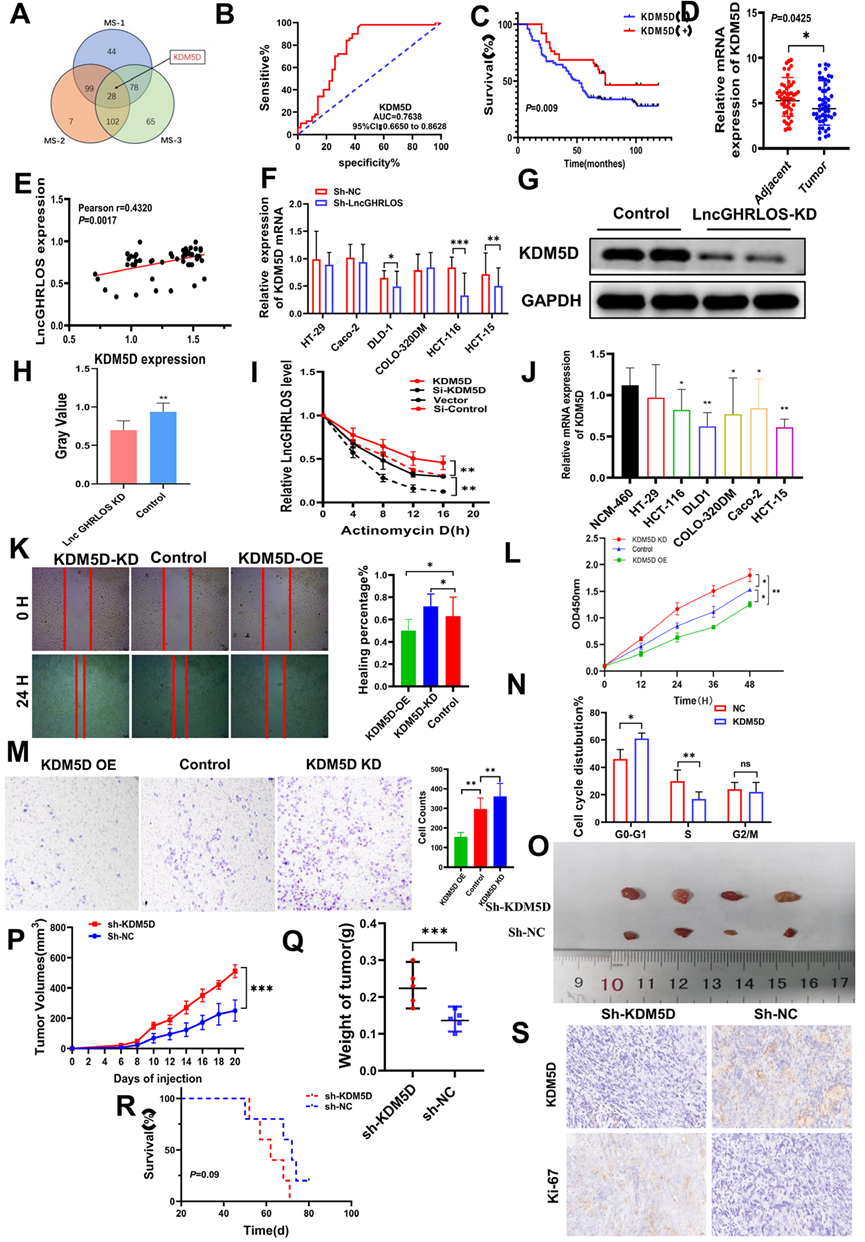
结论:
我们的研究确定了ZCCHC4是CRC不良预后的新预测因子和潜在的治疗靶点,为其在癌症进展中的作用提供了机制见解。
实验方法:
m6A RNA甲基化试验,ELISA,IHC,Western blot,qRT-PCR,MeRIP-seq,RIP,transwell,伤口愈合试验,RNA下拉,质谱。
参考文献:
Chen K, Zhang J, Meng L, Kong L, Lu M, Wang Z, Wang W. The epigenetic downregulation of LncGHRLOS mediated by RNA m6A methylase ZCCHC4 promotes colorectal cancer tumorigenesis. J Exp Clin Cancer Res. 2024 Feb 7;43(1):44. doi: 10.1186/s13046-024-02965-5.






