单细胞全长转录测序

单细胞全长转录测序
单细胞测序只能从 cDNA 模板的一端产生短读长数据,从而限制了序列完整性的重建。单细胞全长转录组测序利用Nanopore长读长实时测序技术结合10X单细胞测序技术可以直接读取带有细胞标签的全长cDNA序列。此方法获取单个细胞中所有ployA+ RNA分子的全长序列,从而识别各种细胞亚型中可变剪切转录本(isoform)类型及丰度。
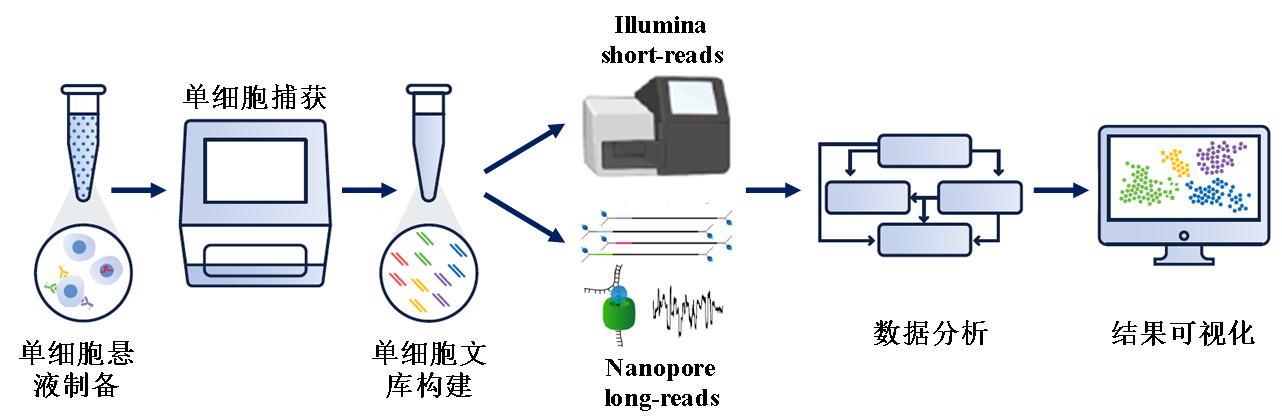
实验流程
研究案例
High throughput error corrected Nanopore single cell transcriptome sequencing (Nat Commun,IF: 16.6 Q1)
基于液滴的高通量单细胞分离技术极大地提高了单细胞转录组分析实验的通量。然而,但基于短读测序只能分析转录本的一个末端序列。本研究介绍了一种结合牛津纳米孔测序和独特的分子标识符的方法,通过10xGenomics单细胞分离系统获得纠错的全长序列信息。此方法可以在单细胞水平上检查不同的RNA剪切和RNA编辑。
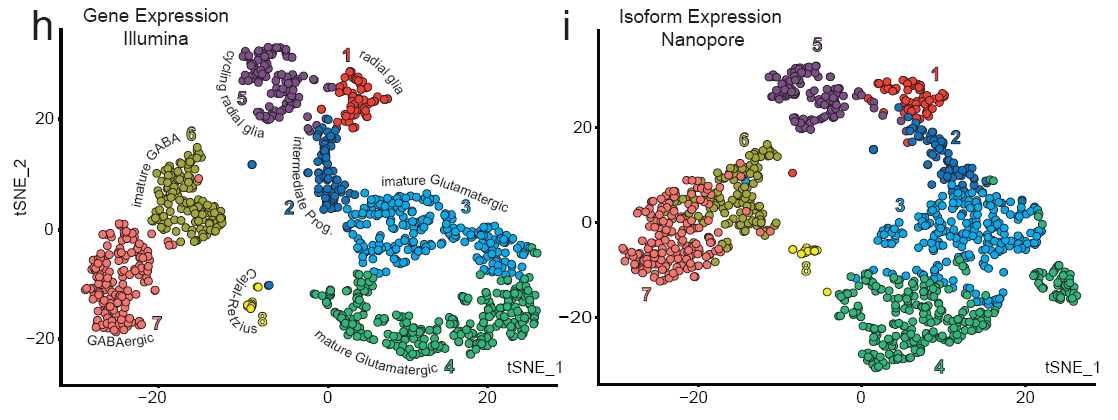
图1 基于Illumina基因表达和Nanopore可变剪切转录本表达量的t-SNE图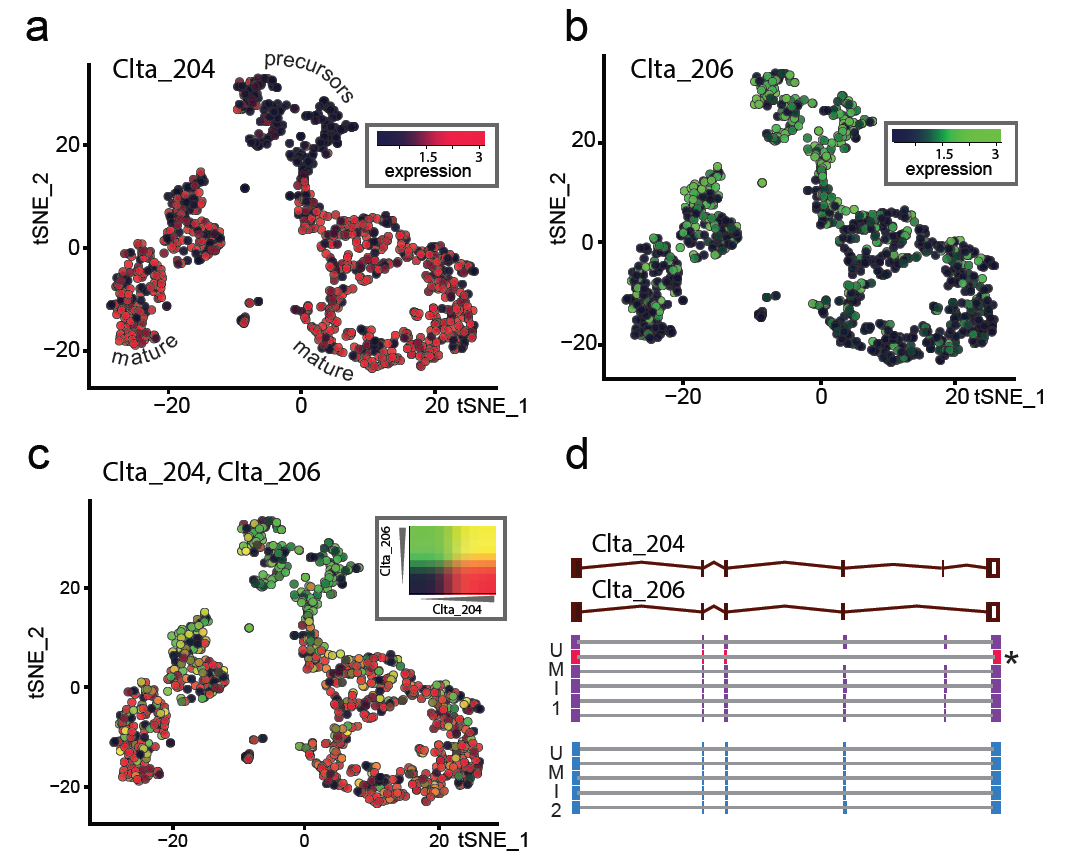
图2 神经元成熟过程中Clta可变剪切转录本t-SNE图
本公司单细胞测序分析内容:
|
|
分析结果可视化结果
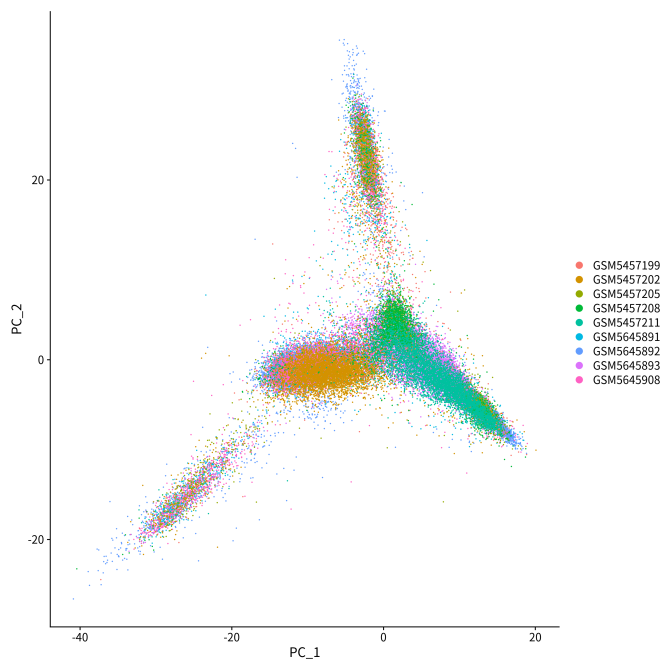

图1 PCA聚类分析 图2 UMAP细胞聚类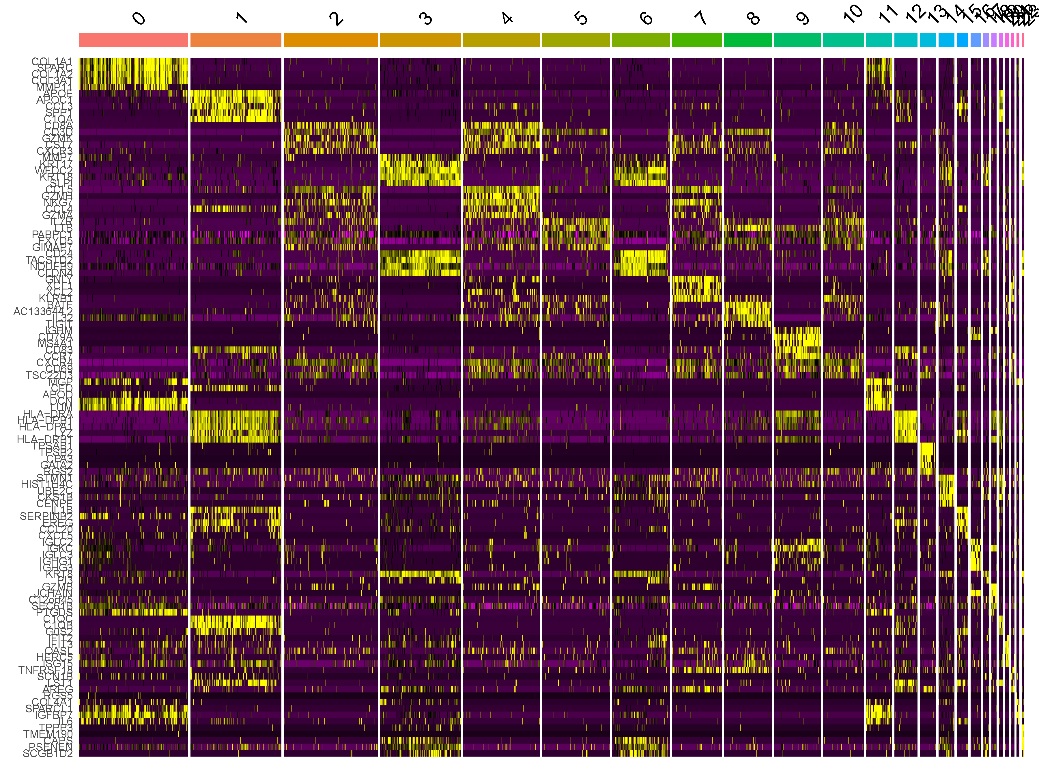

图3 鉴定到marker基因 图4 注释细胞类群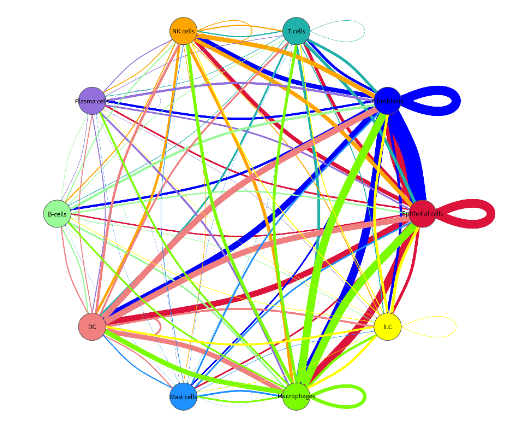
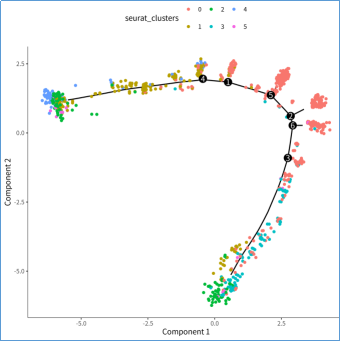
图5 细胞间通讯分析 图6 拟时序分析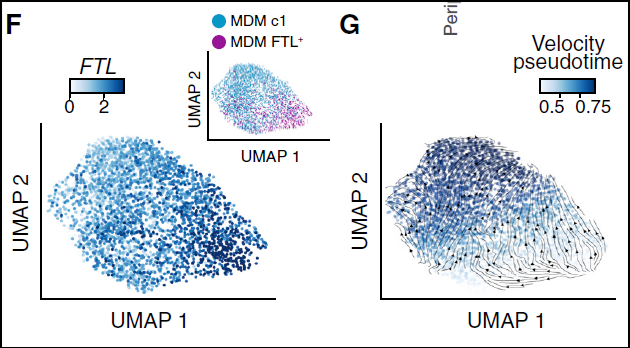
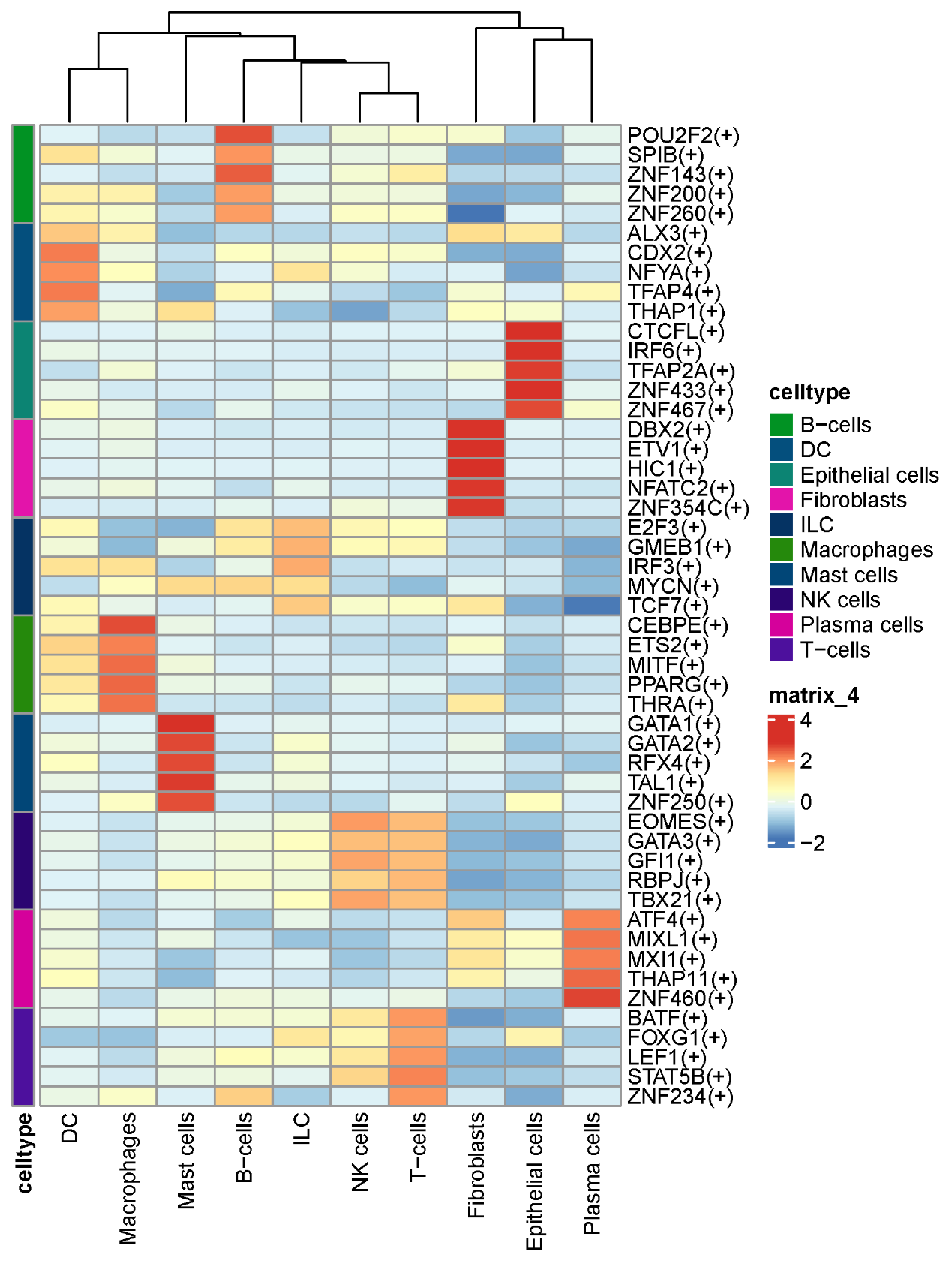
图7 RNA速率分析 图8 转录因子活性分析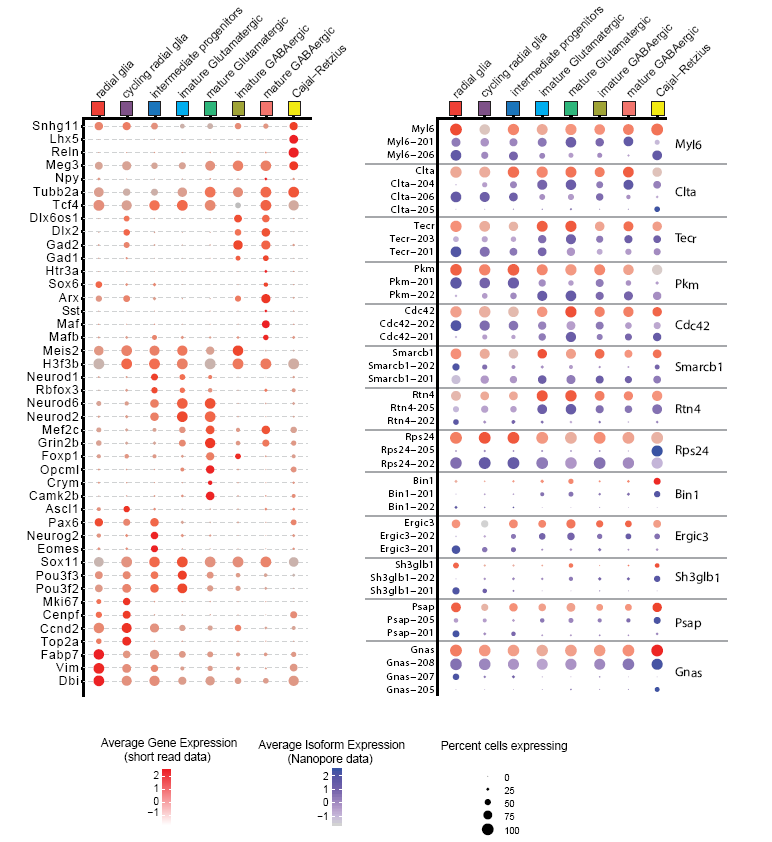
图9 各细胞亚型中差异表达基因及可变剪切转录本
送样要求:
新鲜组织、血液样本、单细胞悬液寄送:
组织样品 | 血液 | 单细胞悬液 | |
样品量 | 组织>0.2g | 人4mL,鼠1-2mL | 细胞量>10^6 |
保存 | 组织保存液 | 抗凝管 | 自选缓冲液 |
运输 | 冰袋运输4℃左右 | 冰袋运输4℃左右 | 冰袋运输4℃左右 |
参考文献
Lebrigand K, Magnone V, Barbry P, Waldmann R. High throughput error corrected Nanopore single cell transcriptome sequencing. Nat Commun. 2020;11(1):4025. Published 2020 Aug 12. doi:10.1038/s41467-020-17800-6 IF: 16.6 Q1