新技术—不使用抗体的m6A测序技术
美国杜克大学医学院的Kate D. Meyer取得一项新进展。研究人员开发了一个名为DART-seq的技术,该方法能够不使用抗体来对全局的m6A修饰进行检测。相关论文“DART-seq: an antibody-free method for global m6A detection” 于2019年9月23日在线发表于《自然—方法学》, IF= 28.467。
原理:在DART-seq中,胞苷脱氨酶APOBEC1与m6A结合的YTH结构域融合。细胞中APOBEC1-YTH的表达可在与m6A残基相邻的位点诱导C-U脱氨,可使用标准RNA序列检测到。 DART-seq可从低至10ng的总RNA中识别出细胞中的数千个m6A位点,并可随时间检测细胞中m6A的积累。此外,研究人员使用长读本的DART-seq来深入研究了m6A沿单个转录本长度的分布。
研究人员表示,改变甲基化位点附近序列的策略将能够通过标准RNA-seq检测m6A,从而克服了当前方法的主要局限性,APOBEC1是一种胞嘧啶脱氨酶,其靶向DNA和RNA以诱导胞嘧啶-尿嘧啶(C-to-U)编辑。尽管最初发现它具有编辑ApoB mRNA的能力,但是APOBEC1自此被用于基于CRISPR–Cas9的基因组编辑方法中,以在目标单链DNA位点诱导C-U转换。我们推测,可以通过将APOBEC1与m6A结合的YTH域融合并用RNA-seq检测随后的编辑事件,采用类似的策略来编辑RNA中的m6A邻近胞苷。在这里,我们展示了这种方法的实用性,可使用转录组范围的作图法以低至10ng的总RNA作为输入来检测细胞RNA中的m6A位点。我们的策略综述了基于抗体的甲基化RNA检测方法,并提供了对单个转录亚型中m6A残基聚类的见解。这种方法大大改善了与全局m6A检测相关的时间和成本,并将能够在有限的RNA样品中进行转录组范围的作图。
一、制定有针对性的脱氨策略以检测m6A
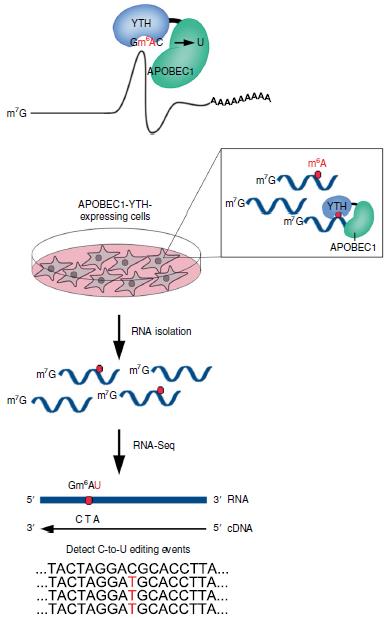
开发用于m6A检测的无抗体方法。m6A的首先选用共有序列包含紧接m6A位点的恒定胞苷残基(Rm6ACH,其中R = A或G; H = A,C或U),作者推测将APOBEC1募集到m6A位点将使m6A残基紧接着的胞苷脱氨。为了测试这一点,我们将APOBEC1与YTHDF2的m6A结合YTH域融合。然后将APOBEC1-YTH融合蛋白与含有单个内部腺苷的合成RNA孵育。反转录和Sanger测序表明在甲基化RNA中紧接m6A后立即对胞苷进行频繁编辑,而在未甲基化RNA中则不是如此(补充图1a)。APOBEC1与YTH域融合,以指导在m6A位点附近的胞苷残基上进行C-to-U编辑。DART-seq方法的示意图。APOBEC1-YTH在细胞中表达,分离出总RNA并进行RNA测序。然后检测到C到U突变以鉴定m6A的位点。
二、DART-seq可识别整个转录组中包含m6A的RNA
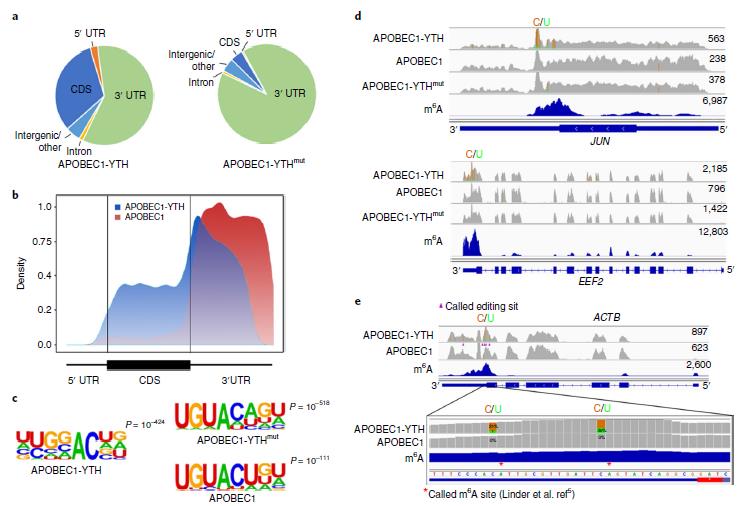
A:饼状图,显示了表达 APOBEC1-YTH(左)和 APOBEC1-YTHmut(右)的细胞中不同转录本区域中 C-U 编辑位点的分布(每个 n = 3 个独立样本)。B: Metagene 分析显示了 DART-seq 检测到的 C 到 U 编辑事件分布的密度图。结果代表三个独立实验。C:在表达 APOBEC1-YTH(n = 39,069), APOBEC1-YTHmut(n = 21,841)或单独 APOBEC1 的细胞中,在 C-U 编辑位点周围的± 4-nt 区域内发现的最丰富的基序( n = 6,814)。使用累积二项式分布来计算单个基序富集的 P 值。 D: DART-seq 数据的整合基因组学查看器(IGV)浏览器跟踪,这些数据在 JUN 和 EEF2 mRNA 中表达所示的构建体。在至少 10%的读数中发现的 C 到 U 突变由金色/绿色表示(金色表示 C 位点的丰度,绿色表示每个位置的 U 位点的丰度;两个基因均从负链转录) 。由 MeRIP-seq(蓝色轨道)标识的 m6A。结果代表三个独立实验。 E:在 ACTB 位点的 DART-seq 读取覆盖率。在至少 10%的读数中发现 C 到 U 突变由单个位点的金色/绿色表示(金黄色表示 C 位点的丰度,绿色表示每个位置的 U 位点的丰度; ACTB 指从阴性位置转录)。
三、DART-seq监测 m6A 随时间的变化情况
![说明: C:\Users\y506\AppData\Roaming\Tencent\Users\3003467659\QiDian\WinTemp\RichOle\OQ3`ZFB_(QS9]35$5UMUJ23.png](https://img1.dxycdn.com/2019/1119/536/3380408646642396569-14.jpg)
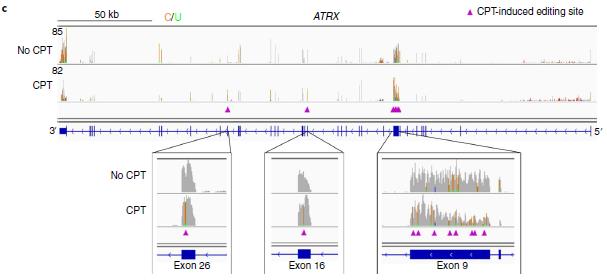
A:与未经处理的对照(UT; n = 40,594)相比, DART-seq 在经 CPT 处理的 HEK293T 细胞(n =5,689)中发现的 C 至 U 突变的分布。 ** P <0.0001。 B-C: IGV 浏览器图像显示了 CPT 处理后 BPTF(b)和 ATRX(c)转录本的 CDS 中的C 到 U 突变。在至少 10%的读数中发现的 C 到 U 突变由蓝色/红色(C / U)着色表示BPTF 转录本(正链),以及 ATRX 转录本(负链)中的金/绿(C / U)着色。 CPT 处理后富集的 C-U 站点由紫色三角形表示。 n = 2 个独立样本。D: MeRIP-RT-qPCR 证实了 CPT 处理后 BPTF 和 ATRX mRNA 中 m6A 的富集。E: 使用未经处理和经 CPT 处理的细胞中的 RNA 进行的 RT–qPCR 分析显示,经过 CPT 处理后, BPTF 和 ATRX 转录本的丰度降低。
四、长时间阅读的 DART-seq 显示了单个 RNA 分子中的 m6A 分布
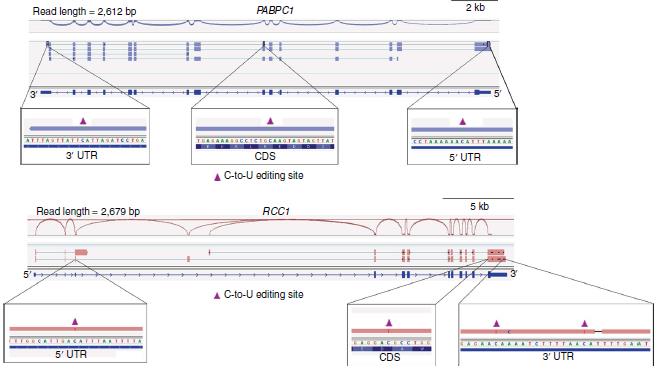
长时间阅读的 DART-seq 显示了同工型特异性甲基化模式。基于免疫沉淀的 m6A 检测策略先前已报告了 m6A 位点的聚类 3–5.但是,尚不清楚这是否反映了 m6A 在相同或不同的 RNA 分子上的聚集。由于 DART-seq 诱导单个转录本中的编辑事件,因此我们认为可以检查单个测序读数以确定在同一 RNA 分子中是否存在 m6A 位点。为了对此进行调查,我们使用 PacBio 平台进行了长时间读取的 DART-seq。单个 mRNA 的检测表明,尽管某些转录本表现出同工型特异性区域编辑,但其他转录本在5'UTR, CDS 和 3'UTR 中包含 DART-seq 位点。此外,有 41%的读取至少跨越两次。






