单细胞转录组测序在病毒感染研究中的应用
往期文章中给大家介绍了单细胞转录组测序的原理及其在系统发育、肿瘤异质性中的研究实例。单细胞分析可以揭示细胞的异质性,不仅是本身,而且能病毒的感染。同样的,在病毒感染研究中,单细胞转录组分析可以发现特定分子标志,鉴定特定表型相关的亚细胞类群,对研究病毒和宿主间的关系很大帮助[1]。今天小编收集了近期发表的病毒感染过程中的单细胞转录组测序研究,供大家参考。
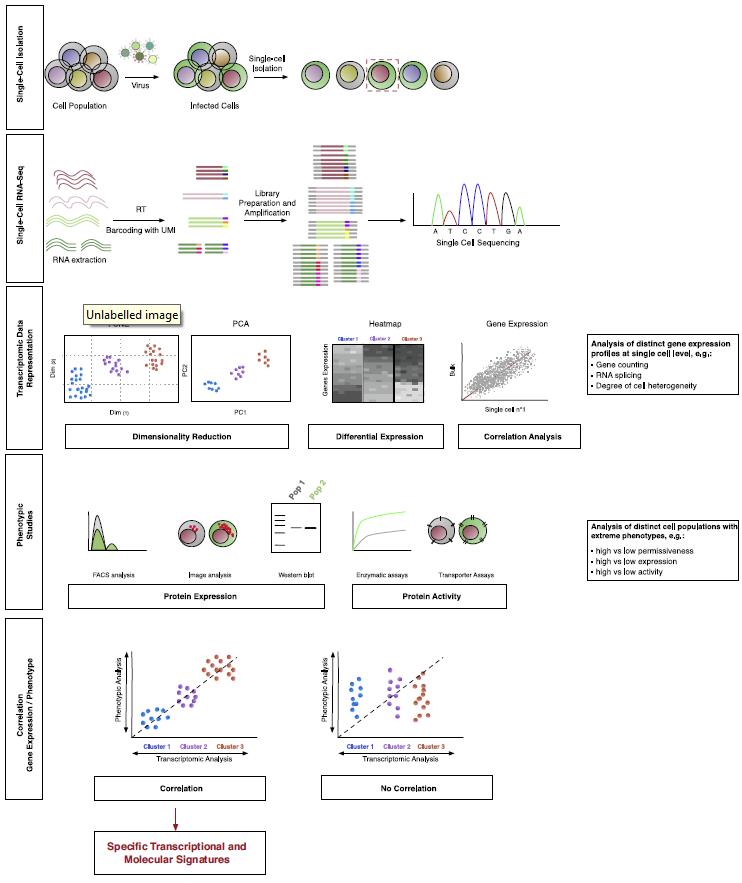
图1 单细胞转录组测在病毒感染中的应用[1]
首先是近期发布于Nature Communication上的Single-cell RNA-sequencing of herpes simplex virus 1-infected cells connects NRF2 activation to an antiviral program (DOI: 10.1038/s41467-019-12894-z)。该文在Hsv-1裂解性感染0、1、3、5小时的原代成纤维细胞中使用Drop—seq的方法进行了单细胞转录组测序。该文对感染时间、宿主细胞周期、病毒基因表达与宿主基因表达间的关联进行了分析并通过标志基因的分析发现了NRF2活性与Hsv-1的复制水平相关[2]。在Hsv-1裂解性感染中,传统bulk RNA-seq测序中所有细胞可能处于不同的感染状态,而单细胞转录组测序很好的解决了这一问题,能在病毒感染过程中提供更准确详细的信息。
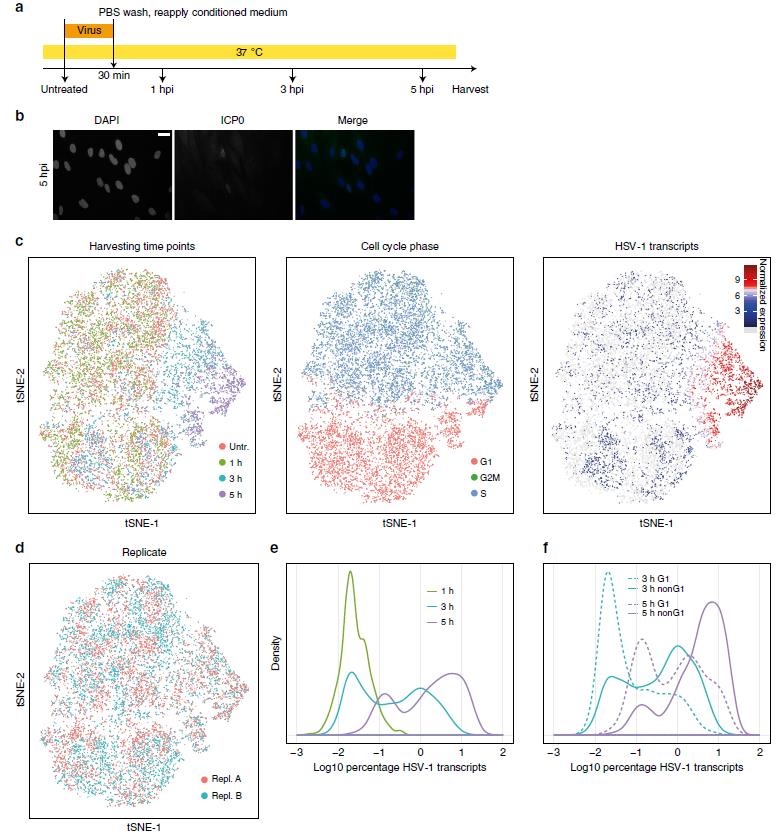
图2 Hsv-1感染原代成纤维细胞的单细胞转录组测序结果[2]
一篇发表于mBio的文章:Defining the Transcriptional Landscape during Cytomegalovirus Latency with Single-Cell RNA Sequencing (DOI: 10.1128/mBio.00013-18) [3]。为了针对性的区分潜伏感染和少数以及进入裂解性感染的细胞,该文对潜伏性感染人巨细胞病毒(human cytomegalovirus , HMCV)的人CD14阳性单核进行了单细胞转录组测序。通过该数据,作者得出新的结论:潜伏性感染相关的基因表达能极大的反映裂解性感染下的基因表达,只是表达量上的差异。并给出了全新的推论:HCMV进入潜伏性感染的过程主要是病毒基因调控表达数量,而不是表达模式。
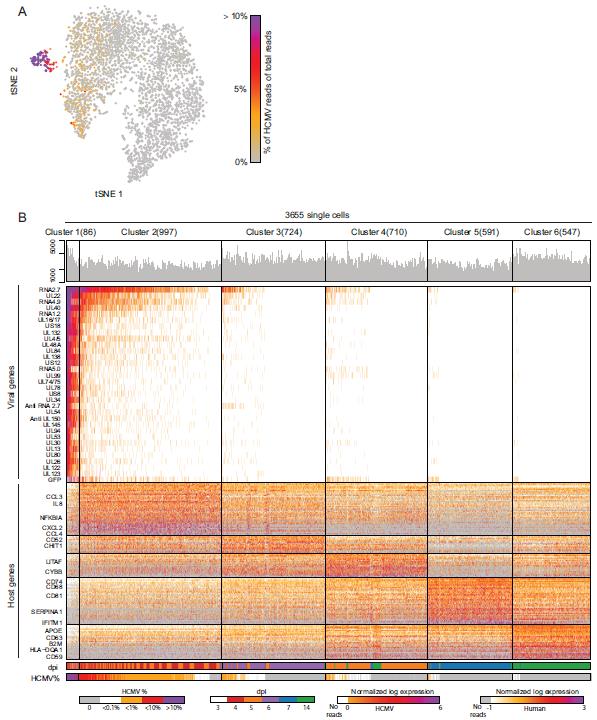
图3 HCMV感染人单核细胞单细胞转录组测序结果[3]
在Single-cell transcriptional dynamics of flavivirus infection[4]一文中,作者在Smart-seq2的基础上进行调整开发了virus-inclusive single cell RNA-Seq (viscRNA-Seq)的研究方法,并对黄病毒属的登革病毒、寨卡病毒的感染进行了测序研究。
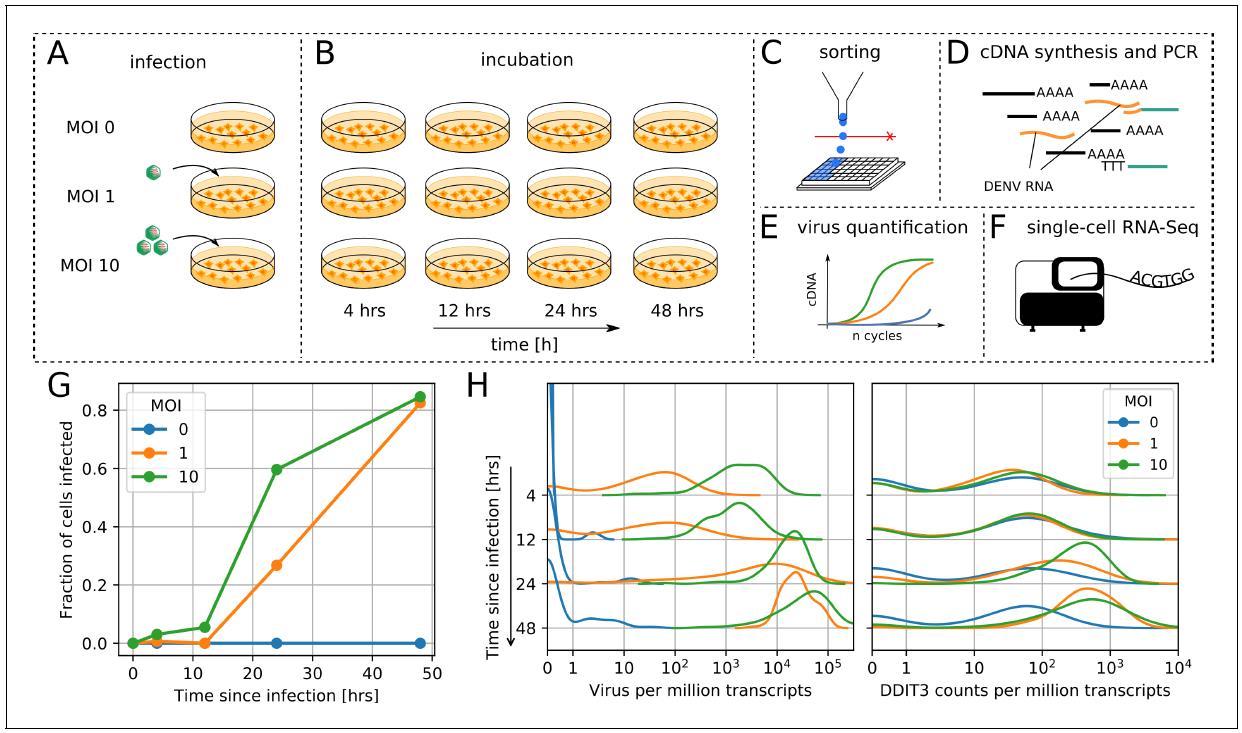
图4 viscRNA-Seq方法模式图[4]
在Dissection of Influenza Infection In Vivo by Single-Cell RNA Sequencing一文中,作者对感染流感病毒的小鼠模型的肺进行了单细胞转录组测序,通过感染和未感染细胞类型的对比分析,找到了新的分子标志物[5]。
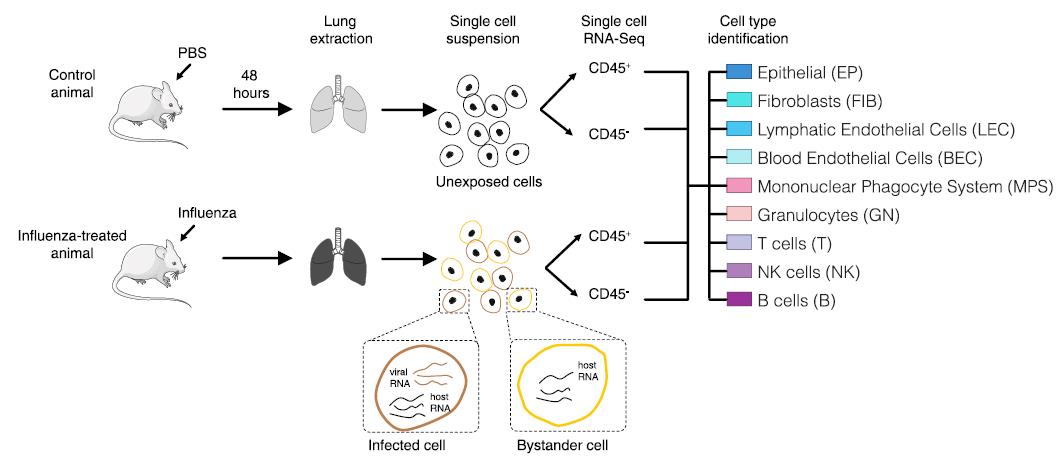
图5 单细胞转录组测序分析流感病毒感染模型小鼠肺组织[5]
其他病毒如HIV [6] [7]也有相关测序结果发表,这里就不一一列举了。最后,单细胞技术也是近期国自然的热点项目。
参考文献:
1. Cristinelli, S. and A. Ciuffi, The use of single-cell RNA-Seq to understand virus-host interactions. Curr Opin Virol, 2018. 29: p. 39-50.
2. Wyler, E., et al., Single-cell RNA-sequencing of herpes simplex virus 1-infected cells connects NRF2 activation to an antiviral program. Nat Commun, 2019. 10(1): p. 4878.
3. Shnayder, M., et al., Defining the Transcriptional Landscape during Cytomegalovirus Latency with Single-Cell RNA Sequencing. MBio, 2018. 9(2).
4. Zanini, F., et al., Single-cell transcriptional dynamics of flavivirus infection. Elife, 2018. 7.
5.Steuerman, Y., et al., Dissection of Influenza Infection In Vivo by Single-Cell RNA Sequencing. Cell Syst, 2018. 6(6): p. 679-691 e4.
6.Rato, S., et al., Single-cell analysis identifies cellular markers of the HIV permissive cell. PLoS Pathog, 2017. 13(10): p. e1006678.
7. Golumbeanu, M., et al., Single-Cell RNA-Seq Reveals Transcriptional Heterogeneity in Latent and Reactivated HIV-Infected Cells. Cell Rep, 2018. 23(4): p. 942-950.






