血清胞外小泡RNA测序的新视点
导语:
细胞外囊泡(EVs)作为临床相关生物标志物的来源具有巨大潜力,因为它们可以很容易地从生物流体中分离出来,并携带可反映疾病状况的microRNA(miRNA),mRNA和蛋白质。但是,EV内含物的生物学和技术性可变性是未知的,这使得健康受试者与患者之间的比较难以解释。在这项研究中,作者试图建立一个实验性和生物信息学分析渠道,以分析患者血清EVs中的小RNA含量,并评估健康个体中EVs的RNA含量的生物学和技术差异,说明这些小RNA可以用作生物标记。
技术路线:
1.细胞外囊泡的提取及其特征鉴定
2.对5个健康供体分离的EV进行了小RNA Seq以评估生物变异
3.对每个供体进行了5个重复的小RNA Seq,以评估技术变异
4.spikein分析揭示了Truseq方法的测序偏差
5.文库的选择影响RNAseq检测的miRNA序列
研究结果:
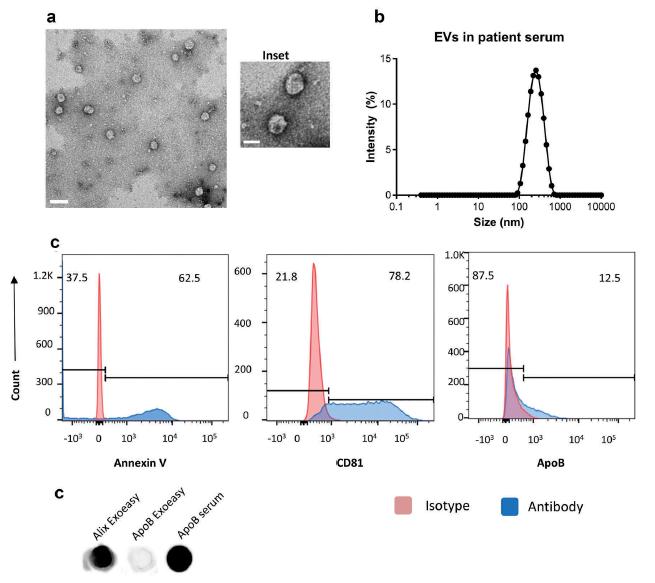
1.从5例健康供体的1毫升血清中分离出EV。透射电镜显示完整的圆形颗粒,颗粒一般小于200nm。用动态光散射法测定其粒径分布,发现其峰值约为200nm左右。通过流式细胞术观察到Annexin V和CD81的存在,但无法检测到CD9或CD63的存在,且表明脂蛋白复合物出现的频率较低。我们发现Alix存在于分离物中,而ApoB不存在。来自患者样本的代表性电图显示了使用exoRNeasy试剂盒获得的RNA的大小分布,这表明该方法产生了预期的RNA大小分布轮廓。
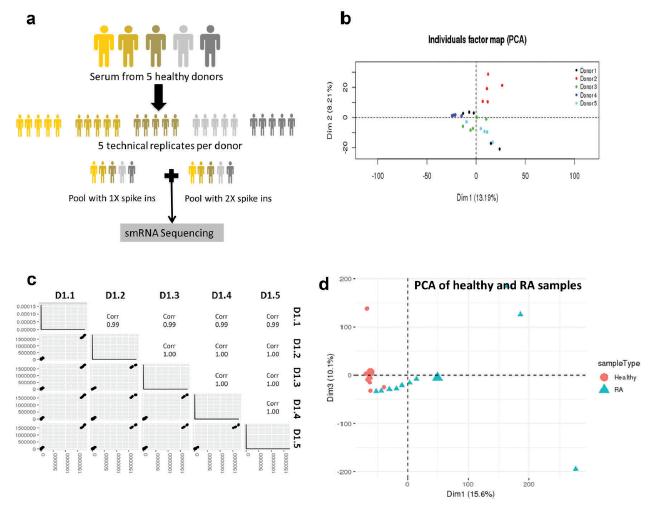
2.为了解决EV小RNA Seq的技术和生物学变异,对从5个健康供体分离的EV进行了小RNA Seq,以评估生物变异,并对每个供体的5个重复进行了小RNA Seq,以评估技术变异。此外,在5个供体中加入了52个浓度为1X或2X的合成spikein,以评估文库制备方法的敏感性和特异性。使用主成分分析评估数据集内数据可变性的来源。这种非监督分析表明,给定供体的技术重复之间的差异远远小于个体间的差异。采用两两离散点图来测量同一供体重复之间的技术方差,估计供体之间的技术方差平均为0.25%(0.10- 10 0.50%)。虽然个体间方差大于技术方差,但未超过健康受试者与类风湿关节炎(RA)患者之间的差异。表明该方法能够识别健康人群与患者人群之间的差异。
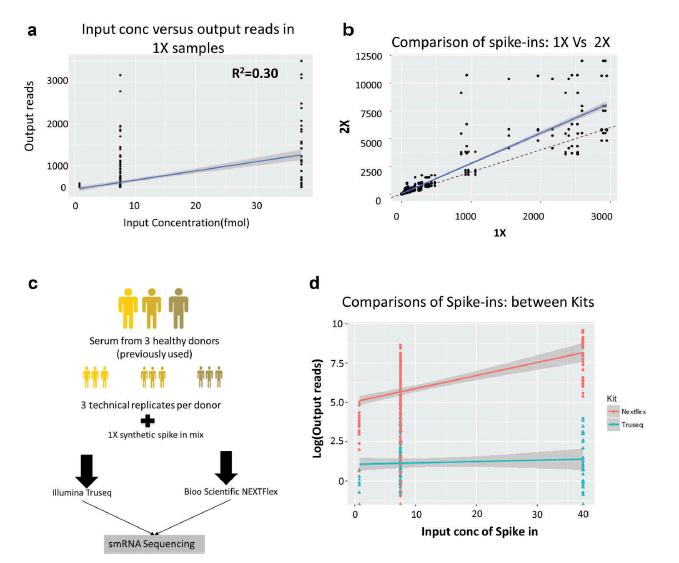
3. 掺有1X和2X数量的合成寡核苷酸的样品用于评估文库制备中的miRNA偏倚。每个样品(1X或2X)被测序3次以创建总共6个文库,每组三个样品的平均值用于分析。具有1X和2X的样品在所有方面都是相同的,除了掺入的浓度不同。我们通过绘制1x和2x样品的已知输入浓度与输出读数的关系图,分析了52个刺入的读数数,以评估文库制备方法的完整性。我们在图3(a)中显示了1X的三个重复样本的输入尖峰浓度与RNA-Seq输出读数之间的相关性。在没有偏见的情况下,我们预计输入的刺入浓度与测序后的输出刺入读数数量相关(接近蓝色所示的最佳拟合线)。但是,我们观察到输入浓度和输出读数之间的相关性很差(R2 = 0.30),表明该方法存在问题。对三个原始供体的一个子集进行了一项后续研究,每个供体包含三个技术重复(总共九个样品),在其中添加了1倍的掺混混合物。然后使用Illumina Truseq小RNA方案或针对小RNA的Bioo Scientific Nextflex方案对9个样品中的每一个进行测序。从第二个研究中获得的加标读物从所有文库中提取出来,并通过方案与输入浓度进行比较,结果表明Nextflex方案在整个输入浓度范围内具有线性范围,总体相关性为0.67,而Truseq的相关性为0.33。此外,在Truseq和Nextflex方案之间,通过卡方检验(p值<0.0001),输出读数与输入浓度之间的相关性在统计上有显着差异。因此,Nextflex方案产生的结果更适合引入的尖峰浓度。
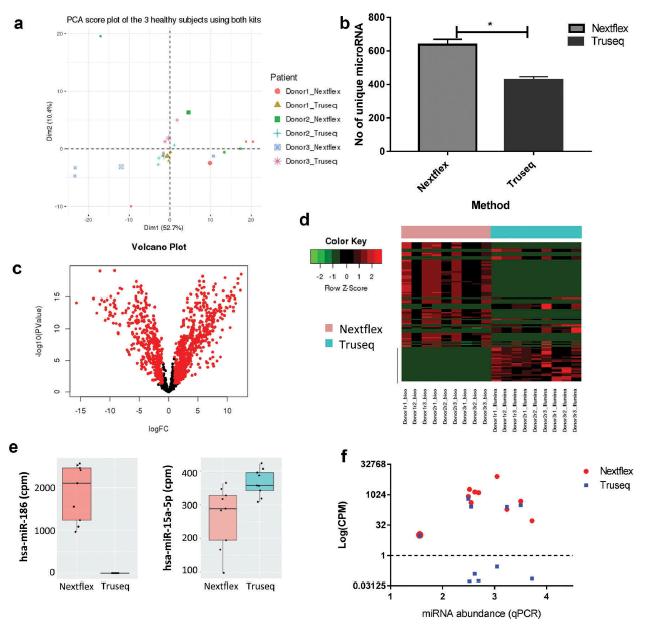
4. 为了进一步评估Truseq和Nextflex文库制备方法之间的差异,比较了每个协议生成的读操作。由供体标记的校正数据集的主成分评分图显示,供体聚集的样本符合预期。然而,kit的选择也影响聚类,无论是生物方差还是kit的选择有助于数据集的可变性。此外发现,与Truseq试剂盒相比,Nextflex试剂盒在供体中识别出了大约150多个独特的mirna,。然而,Truseq方案比Nextflex方案产生了更大比例的miRNA,这一差异具有统计学意义,表明后者可能过度表达了某些miRNA序列而遗漏了其他miRNA,这与spikein分析一致。
使用Truseq和Nextflex方法生成的数据集的技术差异非常小。但试剂盒之间的相关性较差,相关系数很少大于0.55。因此,不能直接比较不同协议生成的数据。对两种文库制备方案获得的所有转录本进行了差异表达分析。差异表达分析显示两种方案之间有1075个转录本存在显著差异。火山图的差异表达基因显示大部分的显著差异是基于>双重的表情变化和p值小于0.05,与前100个差异表达基因的热图显示这些记录的表达水平差别很大。
对10个差异表达的miRNA进行了定量PCR (qPCR),其中包括前5位上调和前5位下调的miRNA。将qPCR数据转化为miRNA丰度并与Truseq和Nextflex方法中相同miRNA的reads进行比较。所有基因在Nextflex协议中都有可检测的读计数,但在Truseq (log (TMM归一化读)>1)中只有5/10可检测到。根据qPCR,未被Truseq方案检测到的5个miRNA分子中,没有一个是低丰度的,这与Truseq文库制备方法存在方法学偏差一致,而不是较差的检测下限。此外,还计算了qPCR和每个文库制备试剂盒之间的Pearson相关性。Truseq和Nextflex的相关系数分别为0.05和0.1。因此,在这两种库准备方法中,Nextflex方法生成的数据更有可能反映mirna的表达水平。
总结:
使用Illumina Truseq方法对来自多个人的EV小RNA进行了测序(生物学重复),并每个人进行了多个重复的测序(技术重复)。我们观察到受试者聚集样本的重复数表明,生物变异(约95%)大于技术变异(约0.50%)。我们观察到约30%的测序读段是miRNA。我们通过将EV RNA制备物掺入合成的小RNA混合物来评估测序的技术参数,并证明了添加RNA的投入浓度和测序Read的频率之间有脱节,这表明在文库制备过程中引入了偏差。为了确定文库制备平台之间是否存在差异,我们将Truseq与Nextflex方法进行了比较,旨在减少文库制备偏差。尽管这两种方法在技术上都非常可靠,但Nextflex方法降低了偏差,并且合成spike-ins的输入浓度呈现出线性分布。总之,我们的结果表明,技术变异性远小于生物学变异性,从而支持了将EV小RNA用作潜在的生物标记。我们的研究结果还表明,文库制备方法的选择导致数据的人为差异,从而导致通过文库制备平台的测序数据的可比性失效。






