PROTAC-DB:PROTAC的在线数据库
蛋白降解靶向嵌合体(Proteolysis-Targeting Chimeras, PROTACs) 是一种新的具有良好前景的药物类型,其结构类似于哑铃,通过一个连接子(linker)连接 “靶蛋白的配体”以及“E3泛素连接酶的招募配体”,即泛素-蛋白酶体系统选择性降解靶蛋白。已成为一种新型治疗技术,具有优于传统抑制策略的潜在优势。
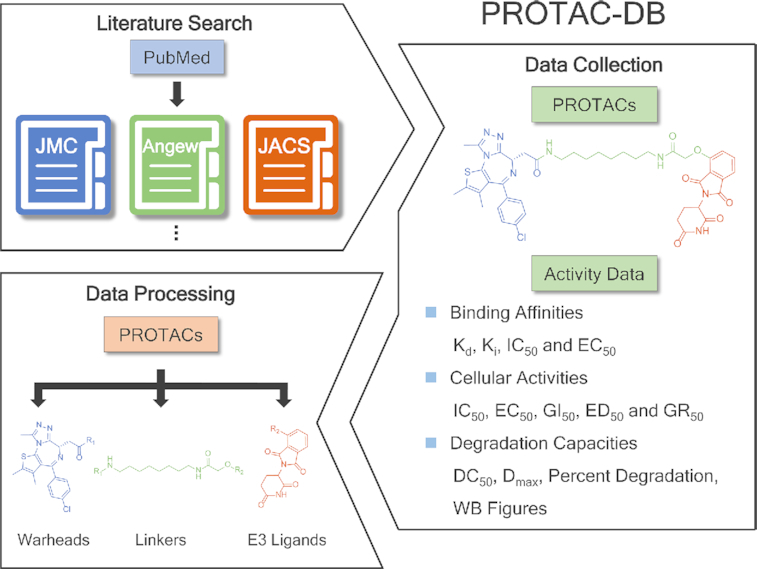
今天讲一篇浙江大学侯廷军教授团队发表在Nucleic Acids Research期刊上的关于PROTAC在线数据库的文章,文章提名为PROTAC-DB: an online database of PROTACs。PROTAC这项技术仍日趋成熟,但PROTAC的设计仍然是一个巨大的挑战。为了促进PROTAC的合理设计,文章提出PROTAC-DB,这是一个基于Web的开放访问数据库,它集成了PROTAC的结构信息和实验数据。
目前,PROTAC-DB包括1662个PROTAC,202个弹头(靶向目标蛋白质的小分子),65个E3配体(能够募集E3连接酶的小分子)和806个接头,以及它们的化学结构,生物学活性和理化性质。除了弹头和E3配体的生物活性外,PROTAC-DB还提供PROTAC的降解能力,结合亲和力和细胞活性。
数据库的查询和浏览
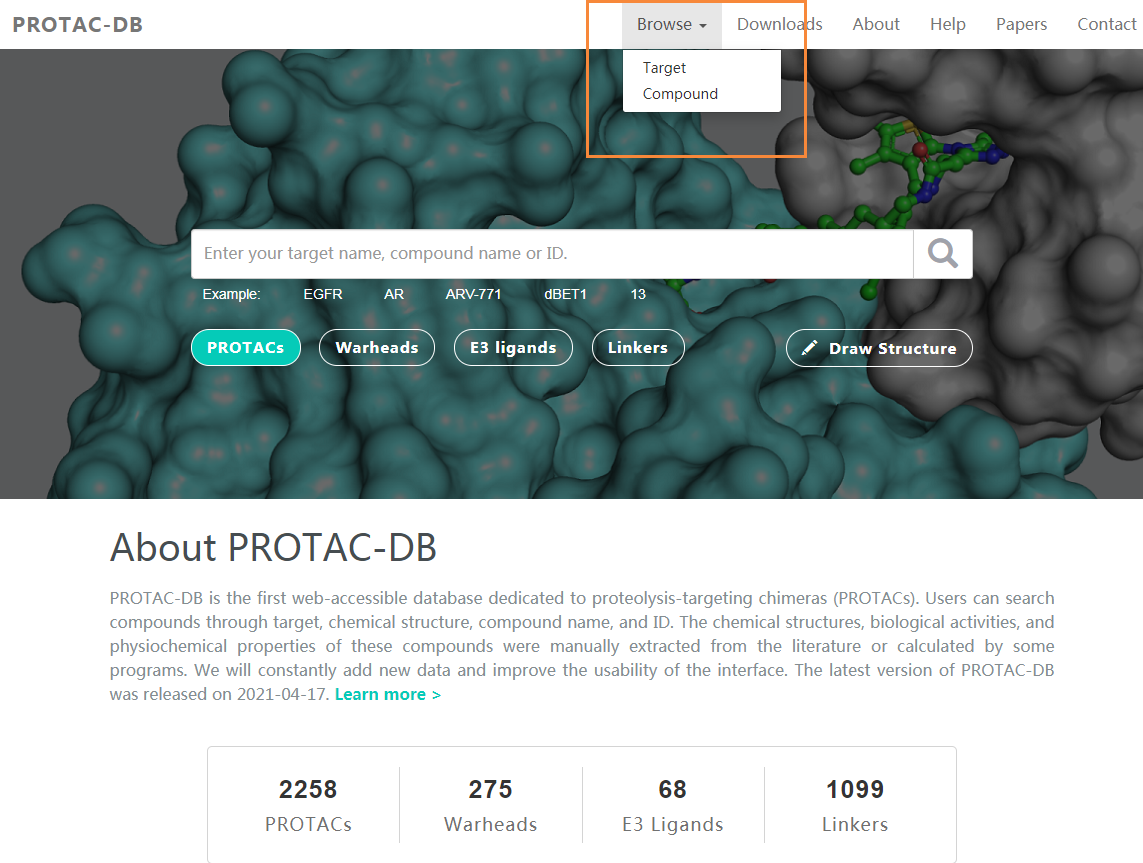
为了方便对PROTAC-DB中的数据进行检索,作者提供了检索和浏览工具。在检索工具上,PROTAC-DB可以进行基于文本的检索和基于结构的检索。基于文本的搜索是在整个PROTAC-DB中进行搜索的一种简单方式,只需输入单个术语,如目标名称、化合物名称或ID。对于基于结构的搜索,用户可以在ChemDoodle编辑器中输入SMILES字符串、上传MOL/SDF文件或绘制分子草图。导入自编辑的分子后,可以从三个搜索选项(similarity、substructure或exact)中选择一个。在相似性搜索中,利用类FCFP指纹中的位向量Morgan指纹来计算两个分子之间的Tanimoto相似度。可以选择数据集(PROTAC、弹头、E3配体或Linker)进行搜索。
浏览工具将PROTAC-DB中的数据归纳为两类:“目标浏览”和“化合物浏览”。目标浏览将在“PROTAC”、“弹头”、“E3配体”和“Linker”类别选项卡下显示目标蛋白质的名称列表,点击列表中选定的蛋白质将跳转到与该蛋白质相对应的所有化合物的列表。化合物浏览主要用于可视化“PROTAC”、“弹头”、“E3配体”和“Linker”类标签下所有化合物的二维结构。此外,在“PROTAC”、“弹头”和“E3配体”类标签下还将展示生物活性。
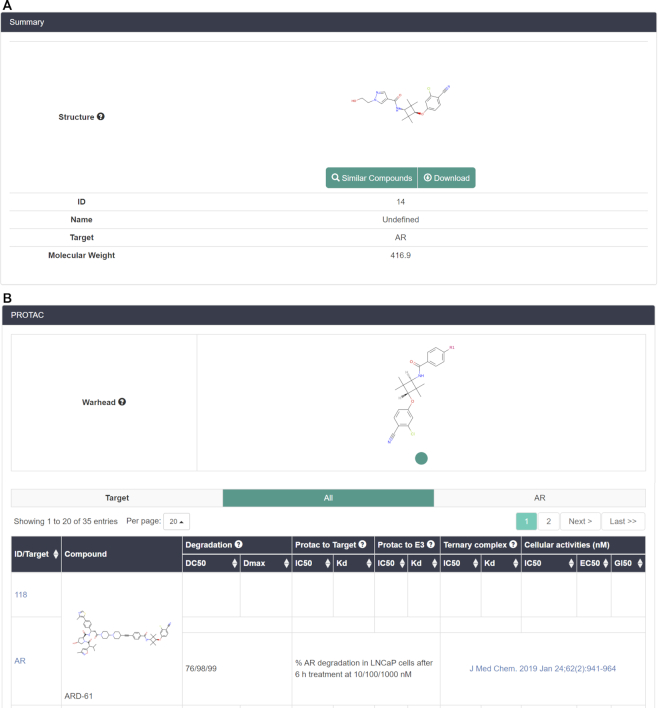
可视化和过滤数据表中的结果
查询或浏览结果显示为数据表,包含2D结构和其他信息,如化合物ID、目标蛋白质和生物活性(图2)。点击该结构的图像可以获得放大的图像。为了帮助用户精细化搜索,PROTAC-DB还包含基于物理化学的过滤工具属性(例如分子量、log P、log S、拓扑极性表面积),包含了搜索结果中每个属性的最小值和最大值。对于PROTAC,除了二维结构、化合物ID和靶蛋白外,数据表中还显示了生物活性,包括降解能力、结合亲和力和细胞活性。该数据表可以根据生物活动的值进行排序。对于弹头和E3配体,搜索结果中只显示了初始结构。修改后PROTAC中的结构汇总在其相应的详细信息页面中。此外,数据表中还显示了初始结构的生物活性。同样,也可以根据这些标准对数据表进行排序。对于Linker,数据表中只显示了2D结构、化合物ID和目标蛋白。结构中的‘R1’和‘R2’分别代表弹头和E3配体的结合位点。