TEA-seq——同时测定转录本、表位和染色质可及性的三峰式单细胞的新工具

2021年,美国华盛顿大学生物医学信息学与医学教育系和华盛顿大学儿科等团队合作在Elife(IF=7.08) 杂志上发表了文章“Simultaneous trimodal single-cell measurement of transcripts, epitopes, and chromatin accessibility using TEA-seq.”。此报道开发了一种新的scATAC-seq工作流程,增加了信噪比,并允许对细胞表面标记物和染色质可及性进行配对测量:染色质环境和表位的整合细胞索引,称为ICICLE-seq。用基于液滴的多组学平台扩展了这种方法,开发了一种三峰分析方法,可以同时测量数千个单细胞的转录组学(scRNA-seq)、表位和染色质可及性(scATAC-seq),并称之为TEA-seq。这些多模式单细胞检测提供了一个新的工具箱来识别基于表型定义的细胞类型的类型特异性基因调控和表达。
外周血单个核细胞(PBMCs)梯度离心纯化是研究人体免疫健康和疾病的临床相关细胞的主要来源。像大多数其他人体组织一样,PBMC是一种复杂的、异构的细胞类型混合物,来源于共同的干细胞祖细胞。尽管不同的PBMC细胞类型之间的基因组大部分是不变的,但每种免疫细胞类型执行重要的和不同的功能。理解控制细胞系规范、细胞成熟、激活状态和响应细胞内外信号的功能多样性的基因组调控景观,是理解健康和疾病中的免疫系统的关键。
最近单细胞基因组方法的改进使复杂细胞类型混合物的调控染色质景观的轮廓成为可能。特别是,基于液滴的单核或单细胞转座酶可及染色质分析(snATAC-seq, scATAC-seq, dscATAC-seq, mtscATAC-seq)允许在单细胞分辨率下分析开放染色质。有前景的新方法将scATAC-seq与同时测量核mRNA或与细胞表面表位结合。
然而,在单细胞方法中,尚未出现一种适用于所有三种模式的统一方法,这种方法可以应用于高度特定的功能免疫细胞类型。我们系统地测试了PBMCs的全细胞和核纯化和制备方法,以克服以往的分析只限于测量细胞表面的核成分(ATAC和核rna)或蛋白质的局限性。我们发现完整的透性细胞的scATAC-seq表现非常好,在某些测量中超过了传统的细胞核scATAC-seq(图1b)。这一发现使得一种类似于转录组和表位的细胞索引(CITE-seq)的新方案能够测量表面蛋白丰度和染色质可及性:染色质景观和表位的整合细胞索引(ICICLE-seq,图1a和3)。最后,我们证明了我们优化的可渗透细胞方法可以与基于液滴的多组学平台相结合,从而能够同时测量细胞的三个不同的分子隔间:mRNA(通过scRNA-seq),蛋白质(使用寡聚标记抗体)和DNA(通过scATAC-seq),我们根据转录、表位和可及性将其称为TEA-seq(图4)。总之,对单细胞水平上基因调控和表达的分子基础有了新的、更统一的看法。
一、PBMC单核、单细胞ATAC序列优化

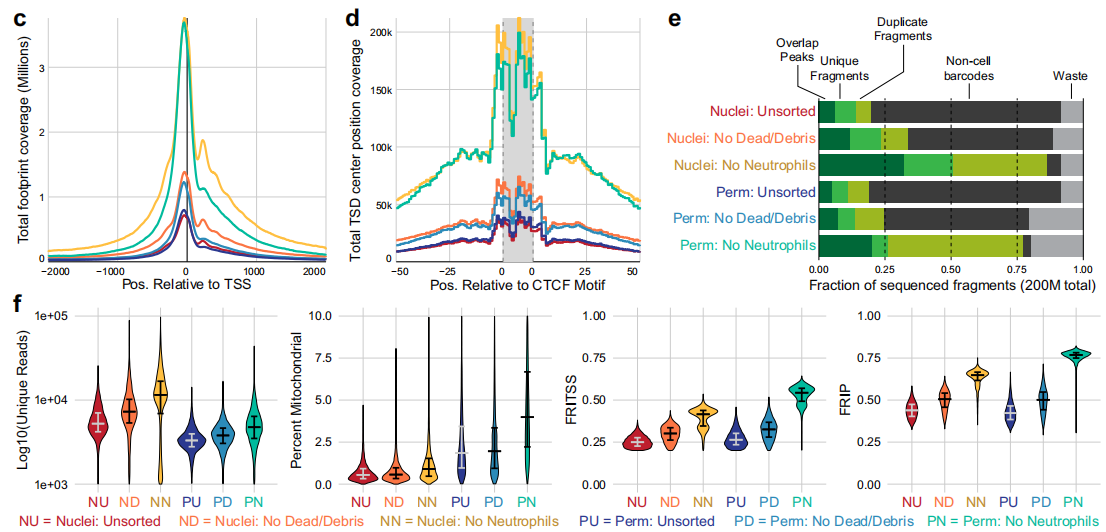
A. snATAC、scATAC和ICICLE-seq方法的主要步骤概要。ficoll纯化的PBMCs和白细胞分离纯化的PBMCs之间的一个主要区别是ficoll纯化样品中存在残留的中性粒细胞。我们使用荧光激活细胞分选(FACS)从高中性粒细胞含量的PBMC样本中检测去除死细胞和碎片的方法,包括去除中性粒细胞和不去除中性粒细胞。
B. 这种单核分析利用低渗裂解、洗涤剂和皂苷的组合来分离细胞核,而不保留线粒体DNA。使用这种方法进行snATAC-seq,测序,并将数据质量指标(材料和方法)制成表格后,鉴定了两个主要的细胞条码群体。细胞核制备的scATAC-seq文库包含更多来自核小体DNA片段的reads,而来自细胞核的非细胞条形码(灰线)包含的这些片段比细胞条形码更多。
C. 为了评估这种差异对每种方法获得的数据的影响,在TSS和CTCF转录因子结合位点附近覆盖了Tn5足迹。在透性细胞中,TSS的信号被保留,但是与孤立的核相比,TSS的侧翼位置(被邻近的核小体占据)的信号减少了
D. 用白细胞分离纯化的pbmc比用Ficoll梯度离心纯化的pbmc始终具有更高的FRIP评分和更少的非细胞条形码。
E. F. FACS获得的完整通透细胞具有最高的frp和FRITSS评分,最少的非细胞条形码和最大的细胞捕获效率,线粒体阅读量仅略有增加.
二、改进标签转移和差异分析
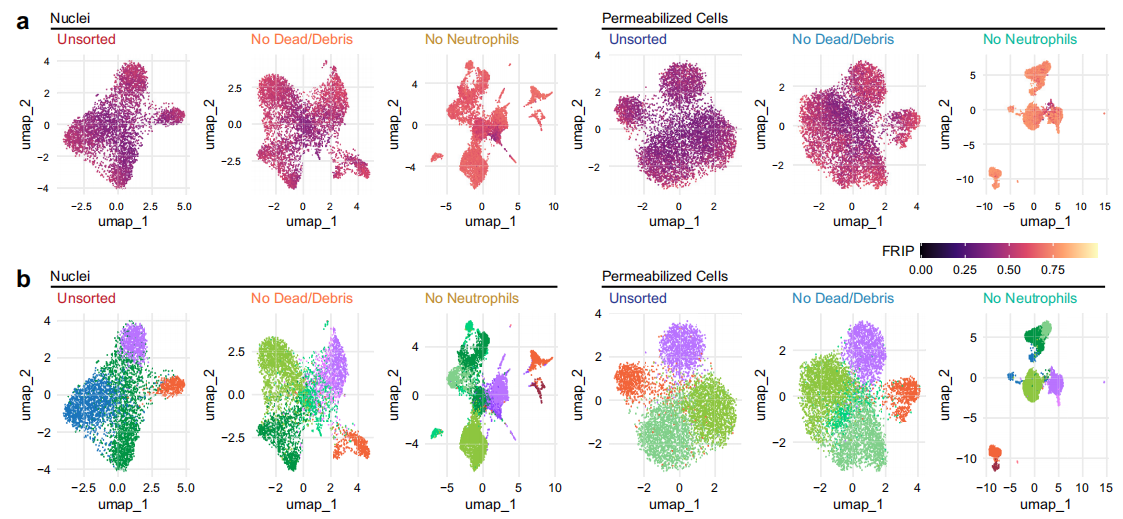
研究了方法差异对下游生物学分析的影响
A. B.去除中性粒细胞大大提高了在细胞核和细胞的均匀流形近似和投影(UMAP)投影中分离各种细胞类型的能力.
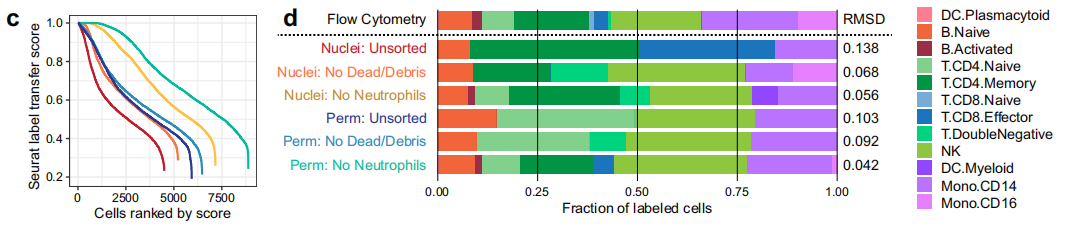
C使用这些工具,中性粒细胞的去除提高了标记转移评分,与核基方法相比,渗透性细胞产生了更多具有高标记转移评分的细胞.
D所有方法产生的CD16+单核细胞均少于流式细胞术观察到的CD16+单核细胞,这表明无论是使用核或通透细胞的scATAC-seq过程中,CD16+单核细胞可能丢失,或者标签转移方法不利于识别这种细胞类型.
三、ICICLE-seq联合测量可及性和表位
A. 在标准的scATAC-seq协议下,细胞膜的去除切断了细胞表面和细胞染色质状态之间的连接。为了测试我们在通透性细胞上同时测量细胞表面蛋白和染色质状态的能力,我们修改了我们优化的通透性细胞scATAC-seq方法,将其纳入使用商用条形码抗体试剂的测量,我们将这种新方法称为ICICLE-seq. ICICLE-seq协议利用定制的Tn5转座复合体,其捕获序列与基因组公司的10x scRNA-seq凝胶珠捕获反应兼容,可同时捕获ATAC片段和多聚腺苷化抗体条形码序列.
B. UMAP投影和ATAC标记转移在ICICLE-seq数据上的分辨率与scATAC-seq在死亡细胞和清除碎片后的完整通透细胞上的分辨率相似.
C. 然而,基于poly- based的捕获方法和置换片段的单端读出限制了数据质量。尽管如此,ICICLE-seq结果表明,通透细胞能够同时捕获细胞核染色质可达性和高质量的细胞表面表位定量。我们能够利用额外的ADT数据聚集并根据细胞表面抗原识别细胞类型.
D. E. 基于ADT数据的UMAP和Jaccard Louvain聚类可以根据细胞类型特异性标记与聚类的明确关联识别细胞类型特异性聚类。
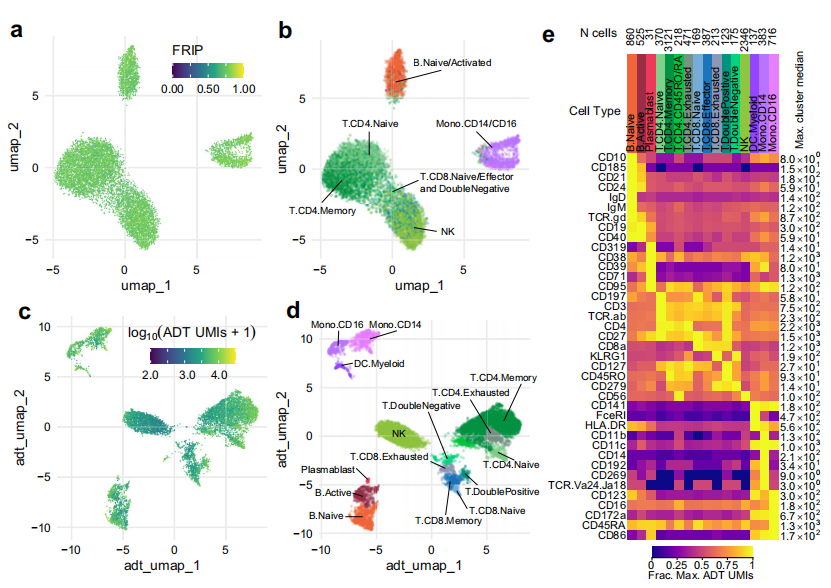
四、TEA-seq转录本、表位和可及性的三峰测量

A. 随着从单个核中同时捕获RNA-seq和ATAC-seq的商业平台的发布,我们推断通透细胞可以用于同时捕获三个主要的分子隔间:DNA可以用scATAC-seq捕获,RNA可以用scRNA-seq捕获,蛋白质表位丰度可以用聚腺苷化抗体条形码捕获,我们根据转录本、表位表位和可及性将其称为TEA-seq。经过试验和关键步骤的优化后,我们能够在10倍基因组Multiome ATAC +基因表达平台上获得文库,该平台使用46个低聚标记抗体组合了数千个单细胞的所有三种测量结果.
B. D.在对装入4孔的细胞进行初始数据处理后,我们鉴定出29264个通过上述scATAC-seq QC标准的细胞条形码,有2,500,500个独特的ATAC-seq片段(中值= 8762个独特的ATAC片段),有>500个基因被scRNA-seq检测到(中值= 2399个RNA UMIs;中位数=检测到129,249个基因),500个ADT UMIs.
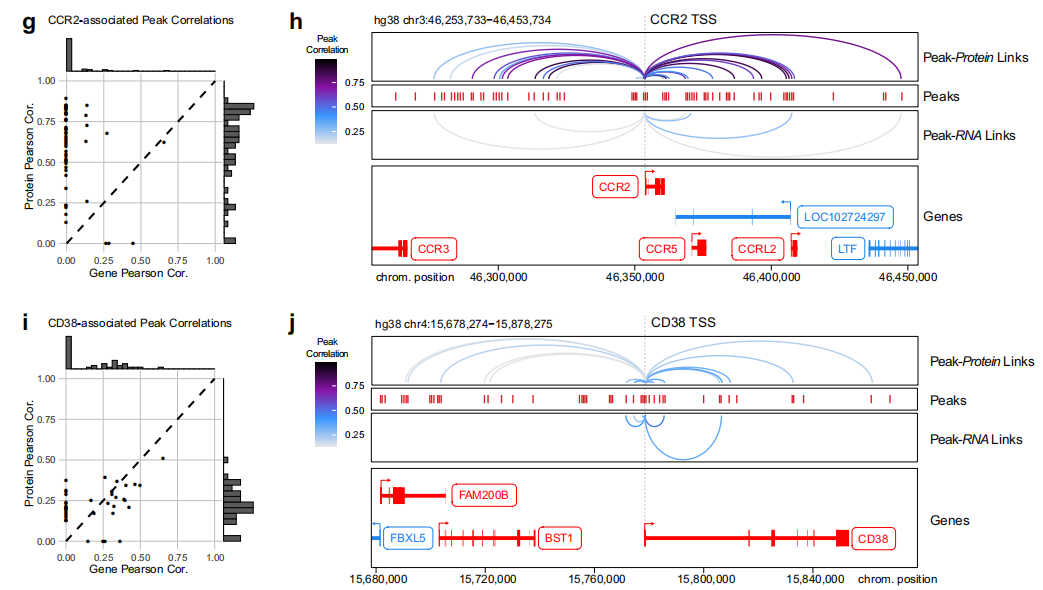
G.H.更敏感的蛋白质丰度测量允许检测许多附加的相关标准物质,例如CCR2/CD192.
I.J. 一种单核细胞上表达的趋化因子受体和CD38,一种表达于多种免疫细胞表面的糖蛋白.






