12+:预测癌症中改变自噬的基因突变

自噬“货物”是有选择性装载的,其中主要依靠LIR基序与LC3互作,形成自噬体。随后,自噬体进行运输、溶解。研究表明,自噬异常会影响疾病进程。肿瘤中的自噬具有双重作用:一方面自噬的发生会增强癌细胞的生存能力;另一方面,抑制自噬会造成“废物”的积累、基因突变等,诱发肿瘤。
本研究旨在探索人类癌症中是否有某些突变通过影响 LIR 基序来改变自噬选择性,进而影响疾病进程。

1. 构建LIR预测模型pLAM
收集人、酿酒酵母、其他物种LIR,并获取相应蛋白,确定了LIR的序列信息。
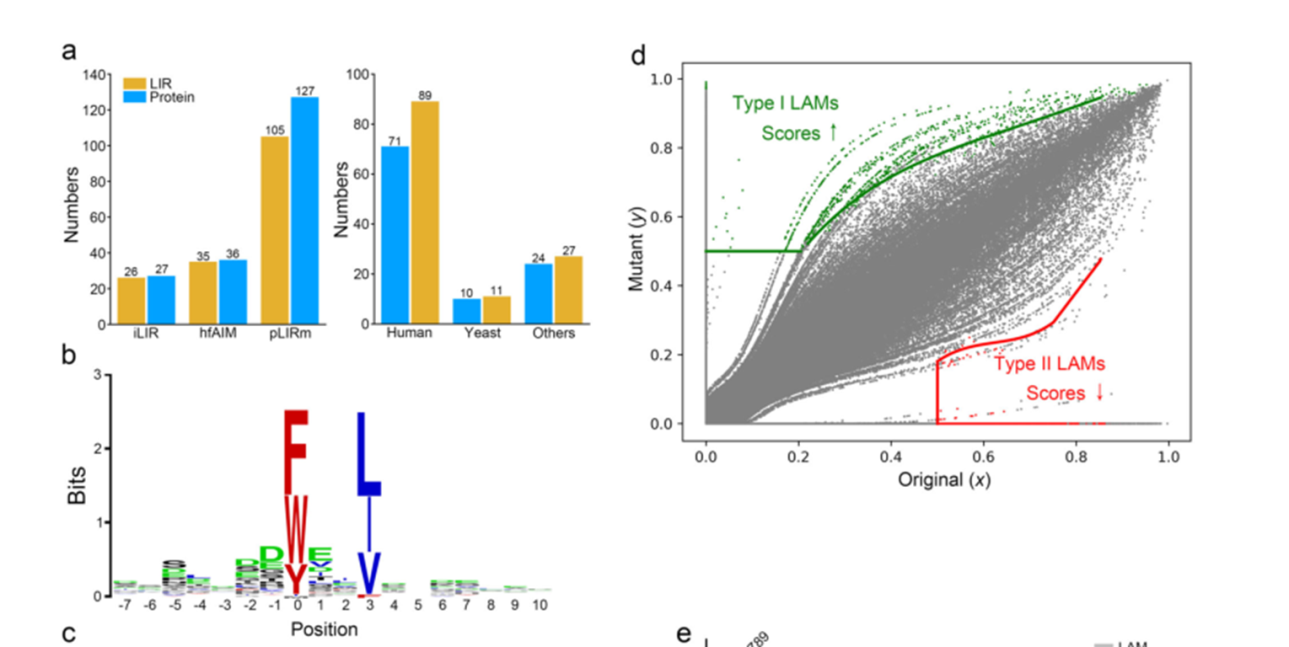
验证算法的可靠性,然后用于泛癌LIR基序预测,并对预测到的LIR基序对应的基因进行GO、Pathway富集分析。
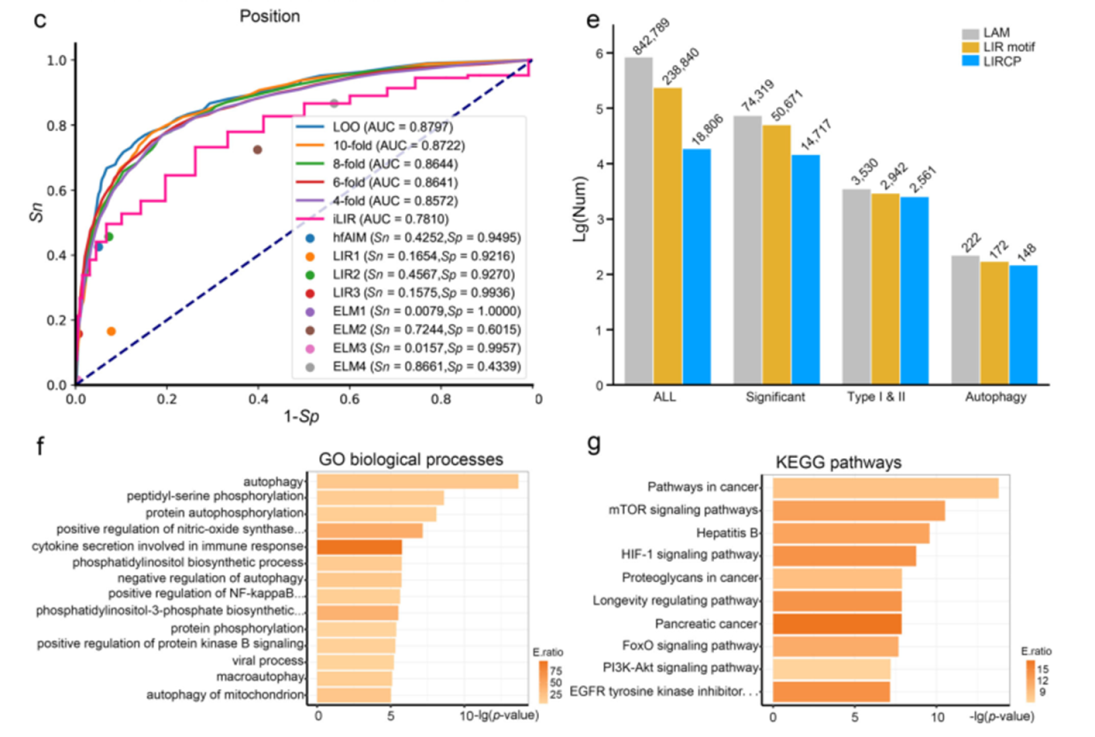
2. LIR基序突变预测
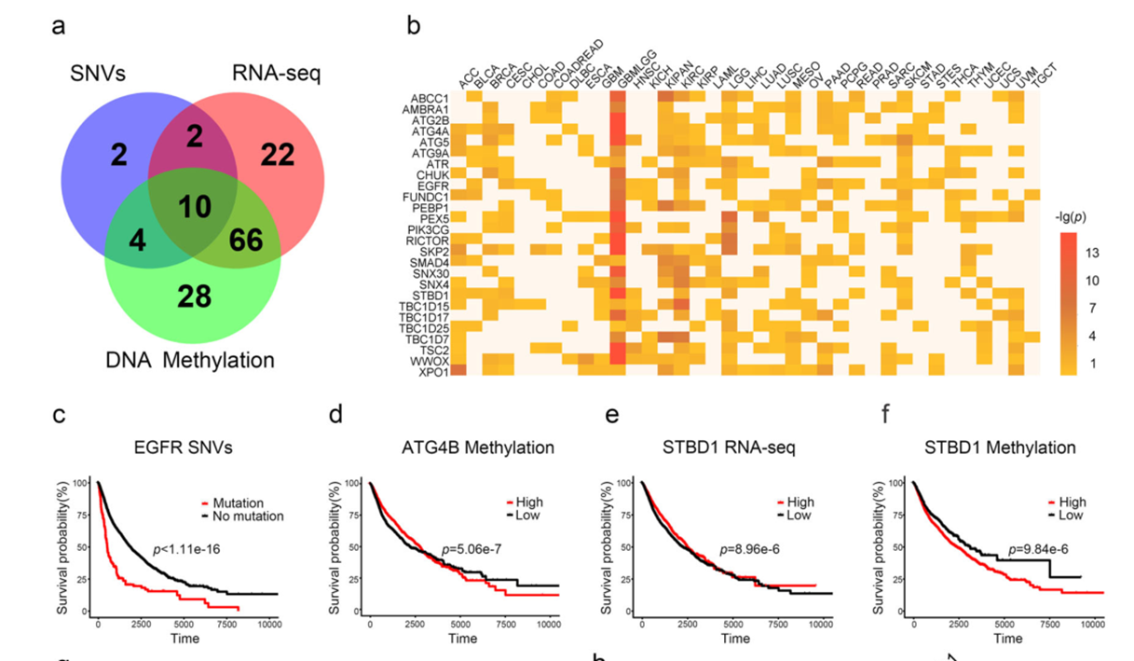
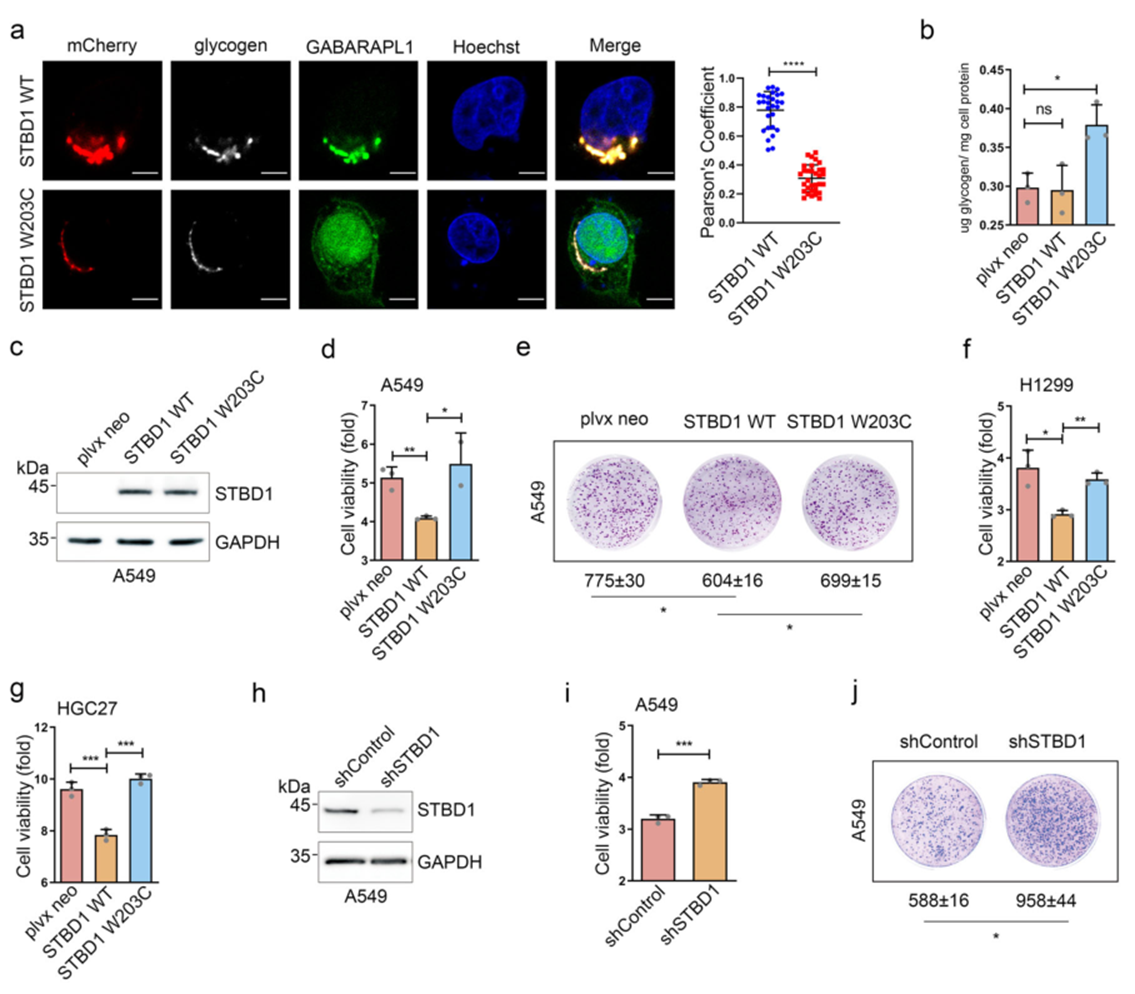
收集TCGA、ICGC 和 COSMIC 数据库突变数据并进行整合,获得2,963,952 个独特的SNV。提取其中LIR基序的突变信息。分析发现STBD1蛋白,参与糖原代谢,在之前尚未报道过与肿瘤自噬有关。

3. 分析突变的LIR基序(LAMs)在泛癌中的作用
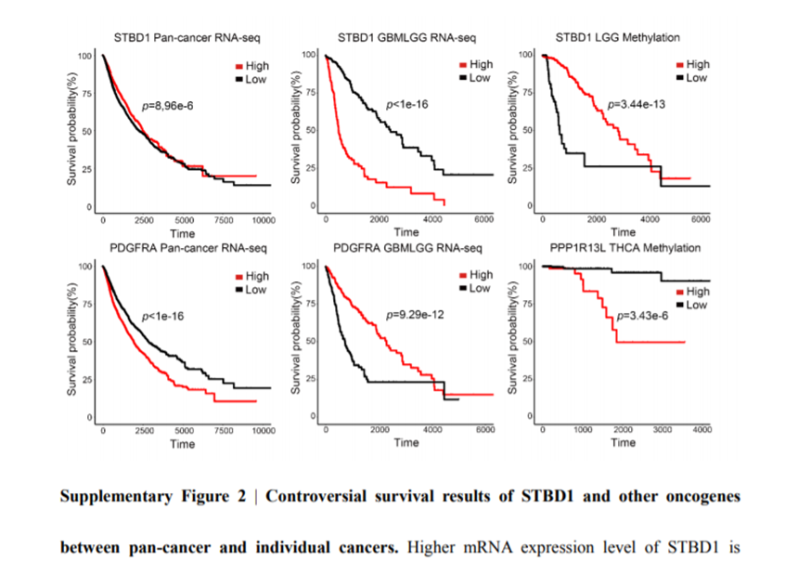
使用TCGA数据库分析发现,STDB1在泛癌中为抑制肿瘤;在胶质母细胞瘤中为促进肿瘤。
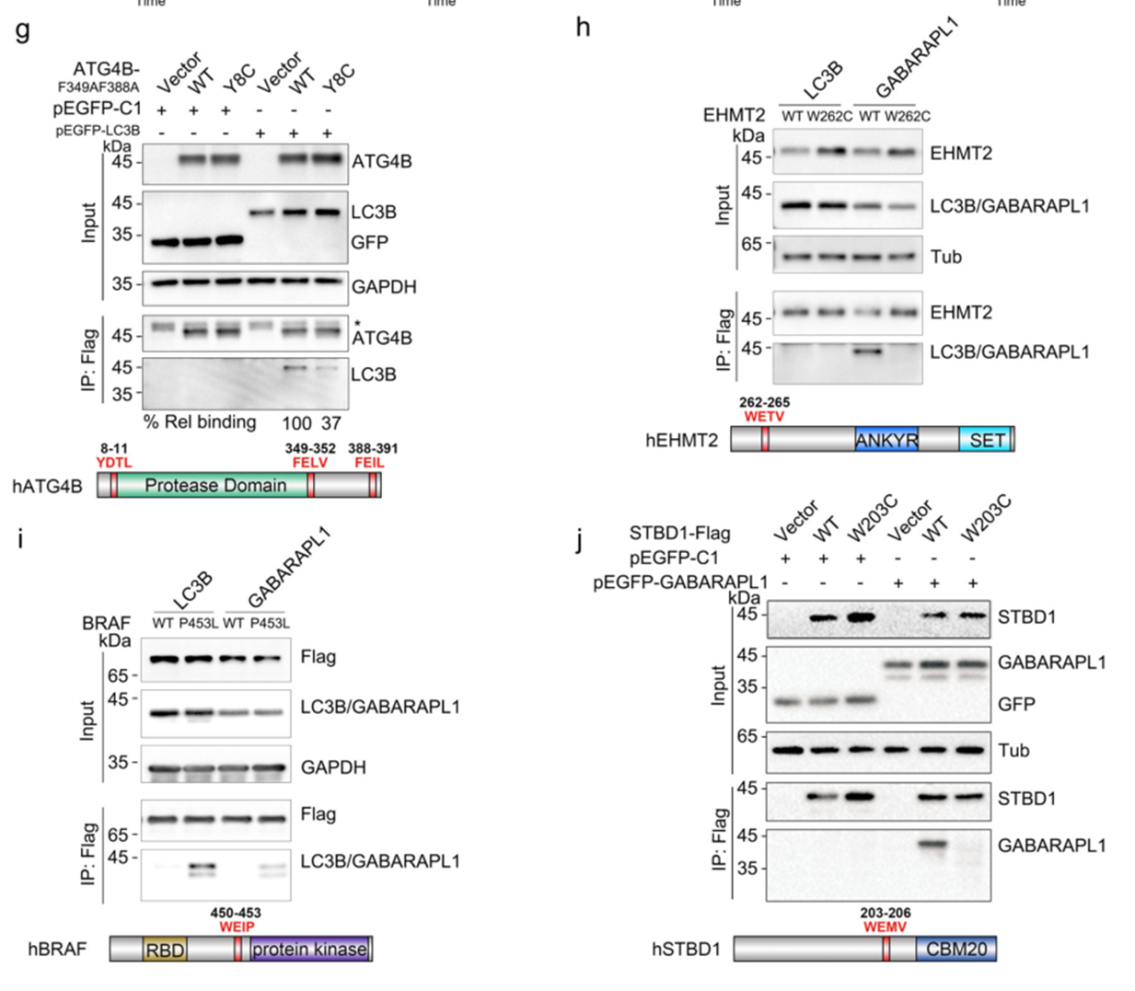
4. co-IP验证LIR基序突变影响自噬
在HEK293T细胞中进行co-IP,结果表明STBD1 LIR基序W203C是影响基因互作的重要结构。
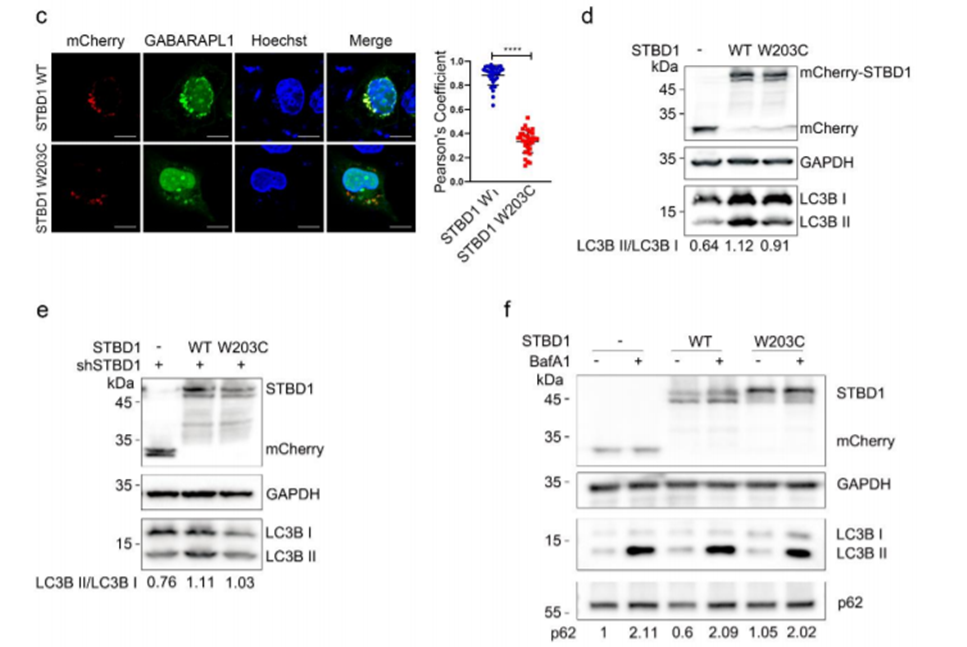
进一步研究发现,STBD中W203C突变影响其与GABARAPL1的结合,进而影响自噬指标的表达。
5. 在临床样本中检测STBD1的表达
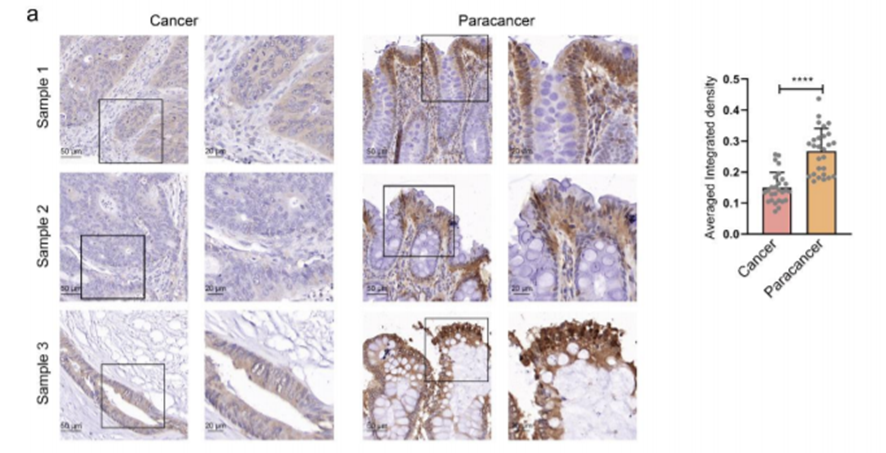
在癌组织、癌旁中检测STBD1的表达水平,结果表明STBD1在癌组织中显著下调,与之前的预测结果一致。
6. 细胞实验研究STBD1的功能
在多种癌细胞中进行细胞功能实验,STBD1 抑制癌细胞生长,突变W203C会部分回复STBD1的抑制作用。
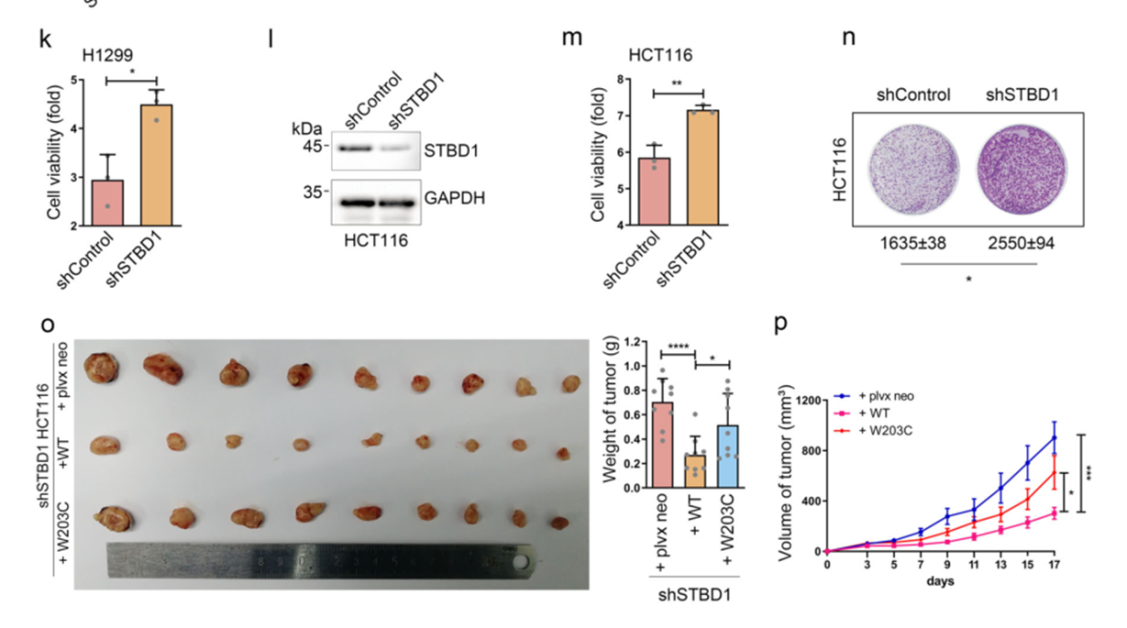
7. 动物实验验证STBD1的功能
裸鼠成瘤实验表明,STBD1抑制肿瘤生长。
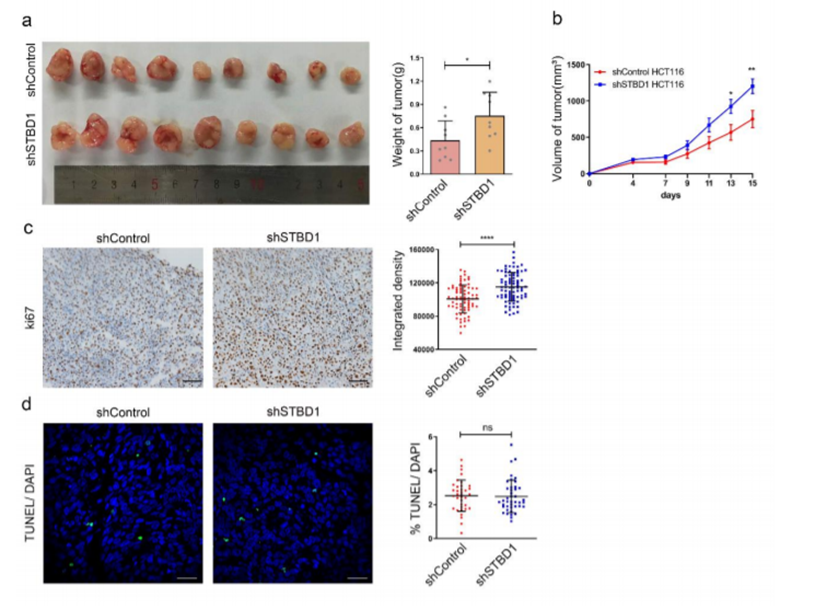
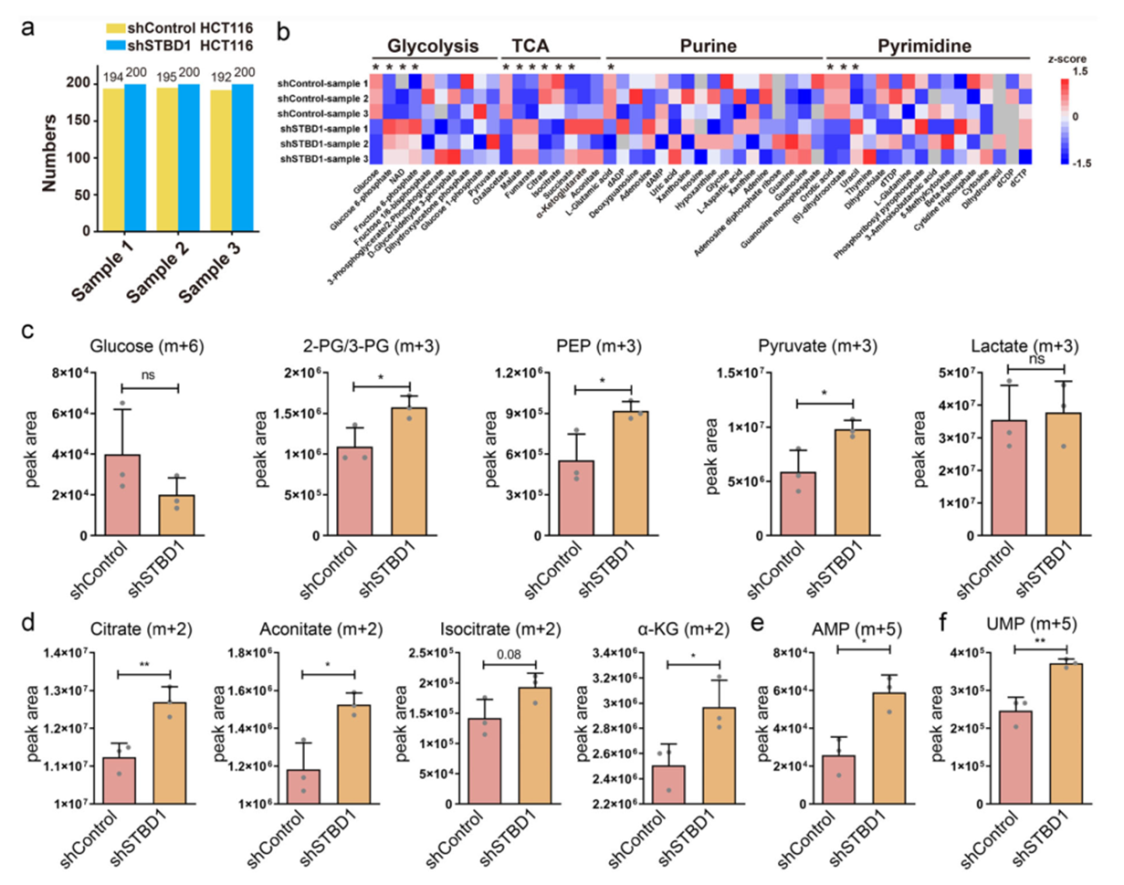
8. 转录组、代谢组分析
在细胞中干扰STBD1,然后进行转录组测序,分析表达水平显著改变的基因,并进行通路富集分析。分析发现,STBD1 抑制癌症生长可能是通过改变基因转录和重塑糖酵解/糖异生途径。
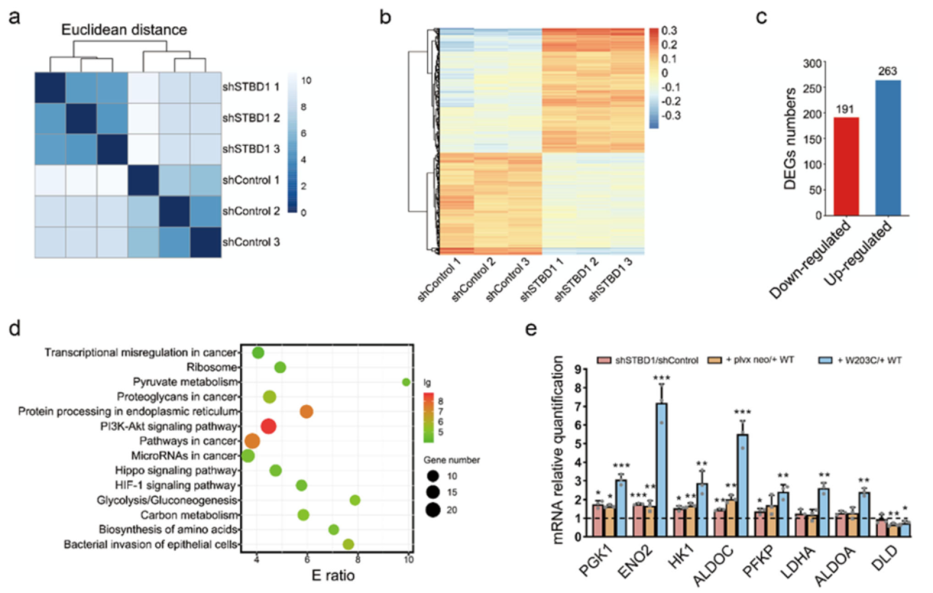
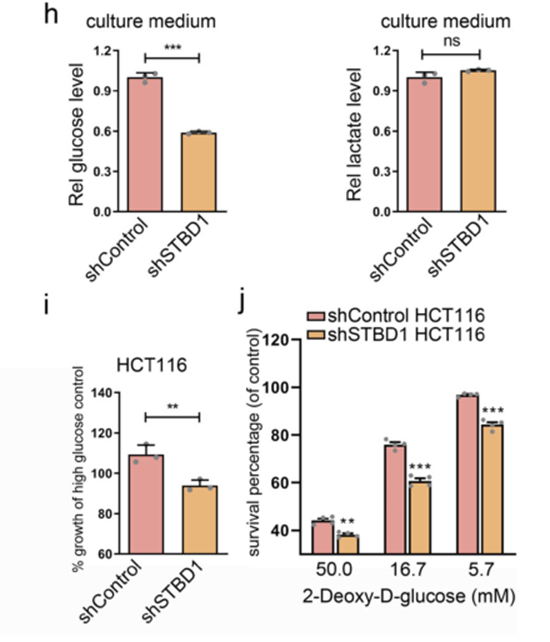
为了进一步验证STBD1是否重塑代谢,进行代谢组分析。结果表明,STBD1敲除后糖酵解增强。
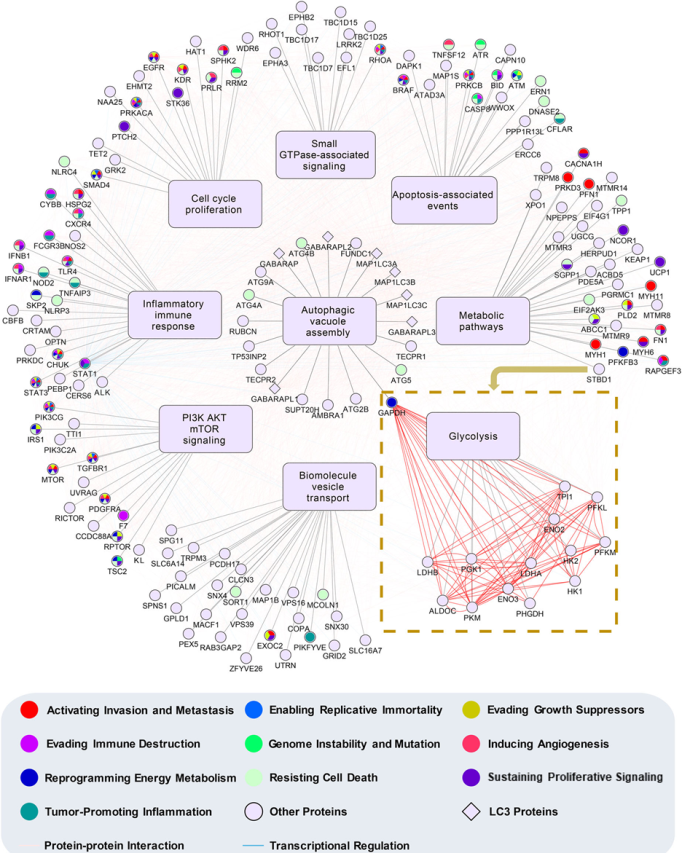
9. 构建LIRCP 调节网络
将LAMs对应蛋白、自噬相关基因按照生物学功能聚类,构建调节网络,将自噬与肿瘤联系起来。