复发性膀胱癌中癌干细胞维持和上皮-间质转化的机制研究
由于治疗抵抗和高复发率,膀胱癌治疗仍然是一个重大的临床挑战。分析肿瘤内异质性可以揭示膀胱癌复发的分子机制。2021年9月发表于“CLINICAL CANCER RESEARCH”(IF=12.531)的文章“Single-Cell Analyses Reveal Mechanisms of Cancer Stem Cell Maintenance and Epithelial–Mesenchymal Transition in Recurrent Bladder Cancer”对此进行了报道。我们的研究生成了一个由54971个单细胞组成的综合癌细胞图谱,并确定了不同的细胞亚群。我们发现膀胱癌复发时肿瘤干细胞亚群丰富,EZH2表达升高。我们进一步定义了一种亚群特异性分子机制,其中EZH2维持H3K27me3介导的NCAM1基因抑制,从而使细胞侵袭性和干细胞转录程序失活。此外,我们阐明了临床样本中上皮-间质转化(EMT)的谱,并揭示了与膀胱癌亚型相关的不同EMT特征。我们发现TCF7促进EMT与scATAC-seq分析相一致。我们的研究和分析方法为膀胱癌研究领域中肿瘤干细胞和EMT的进一步研究提供了丰富的资源。
技术路线

结果
1)膀胱癌的单细胞RNA和ATAC-seq
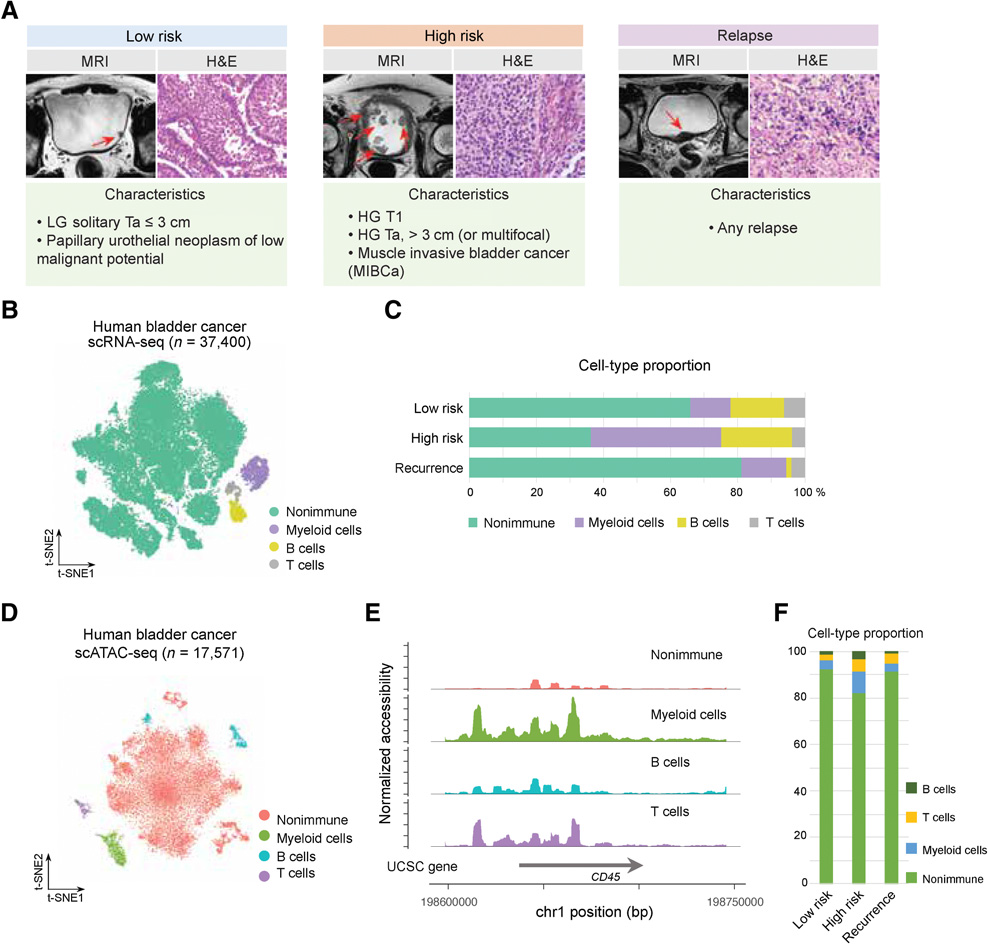
本研究将膀胱癌患者分为低危组、高危组和复发组(图1A)。根据美国泌尿外科协会(AUA)/泌尿肿瘤学协会(SUO)指南对低危和高危NMIBC进行分层,并将MIBC样本分为高危组。在去除低质量的细胞后,我们总共获得了37000个用于scRNA-seq的细胞(图1B和C)和17571个用于scATAC-seq的细胞(图1D-F)。我们整合了低风险、高风险和复发性膀胱癌的数据,并通过检测标记基因确定了scRNA-seq中的主要细胞群。我们还确定了scATAC-seq数据中的主要细胞亚群,并观察到标记基因的开放染色质(图1E)。scRNA-seq和ATAC-seq检索到的大多数细胞是非免疫细胞(图1C和F)。与低风险和复发性膀胱癌相比,我们通过scRNA-seq和scATAC-seq在高风险膀胱癌中检测到更多的免疫细胞(图1F)。我们在scATAC-seq数据中观察到的免疫细胞组分少于scRNA-seq数据(图1C和F)。
2)膀胱癌非免疫细胞的高度异质性
我们首先根据簇特异性DE基因将非免疫细胞(CD45阴性)重新分类为17个簇(图2A和B)。在所有簇中,鉴定出9个基底细胞簇(簇0、2、3、5、8、10、13、14),高度表达角蛋白基因,如KRT5、KRT13、KRT17和KRT19。簇1、6、9和15被指定为表达UPK3A、UPK1A、UPK2、UPK1B、UPK3B和EPCAM的上皮细胞。聚类11中血管内皮细胞富集,高表达CDH5和PECAM1。成纤维细胞(簇4、7、12和16)高表达胶原基因,如COL1A1、COL1A2和COL3A1。最近,据报道,胰腺癌和膀胱癌中的成纤维细胞分为两种不同类型,其中一种表现出多种细胞因子和趋化因子的强烈表达[称为“炎性癌相关成纤维细胞(iCAF)],另一种类似于肌成纤维细胞(mCAF)。我们对成纤维细胞进行了重新分类,发现表达RGS5的iCAF和表达PDGFRA的mCAF也存在于膀胱癌中(图2C和D)。有趣的是,我们发现这些iCAF和mCAF不是肿瘤特异性的,也可以在健康组织中检测到(图2D)。在注释了膀胱癌的细胞亚型后,我们接下来评估了这些膀胱癌细胞类型是否能够反映一致的分子分类。我们将单个细胞的转录组与1750名患者的MIBC转录组进行比较,发现管腔乳头状、管腔非特异性、管腔不稳定、基质丰富和基底/鳞状亚型与我们数据集的细胞类型相对应(图2E)。当将TCGA细胞分数数据与一致的分子亚型相关联时,我们注意到TCGA/BCLA队列可以重现我们的scRNA序列数据中确定的细胞类型(图2F)。

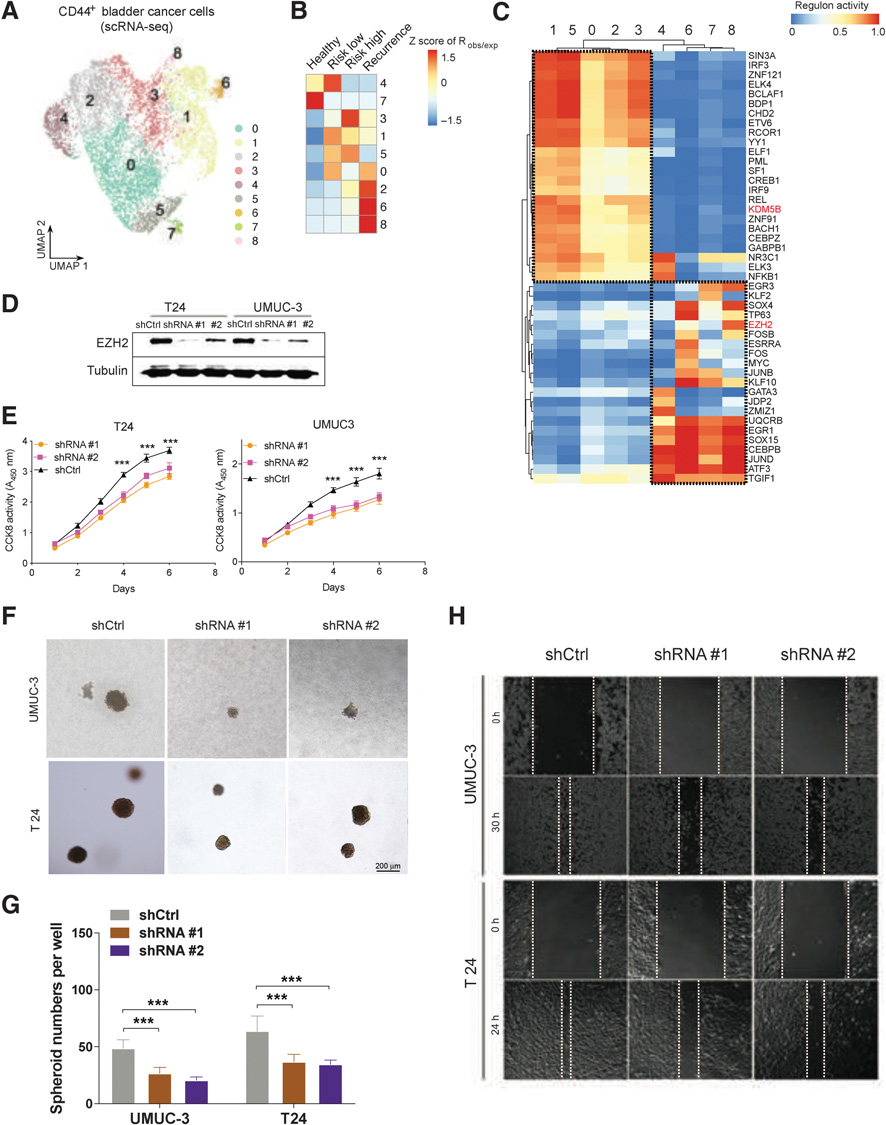
3)scRNA-seq显示复发性膀胱癌中丰富的异质性CSC亚群
我们接下来检查了膀胱癌干细胞(BCSC)标记物CD44和ALDH1A1的表达,发现它们在基底细胞簇2和5中富集,表明BCSC可能来源于基底细胞。这些细胞的重新聚集产生了8个亚群(图3A)。与健康、低风险和高风险人群相比,复发性膀胱癌亚群0、2、6和8更为丰富(图3B)。BCSCs的调控可分为两组,其中一组被CHD2、SIN3A、YY1和KDM5B富集(图3C)。KDM5B是组蛋白H3K4me3的去甲基化酶,在包括膀胱癌在内的多种人类癌症中都有过表达的报道。此外,KDM5B的磷酸化可能调节其在乳腺癌中的活性。另一组BCSC调控子包括SOX15、ATF3、KLF10和EZH2(图3C)。EZH2催化H3K27me3,从而抑制靶基因表达,EZH2的高表达也与膀胱癌的进展有关。因此,我们假设EZH2在BCSCs中起关键作用,从而促进膀胱癌复发。
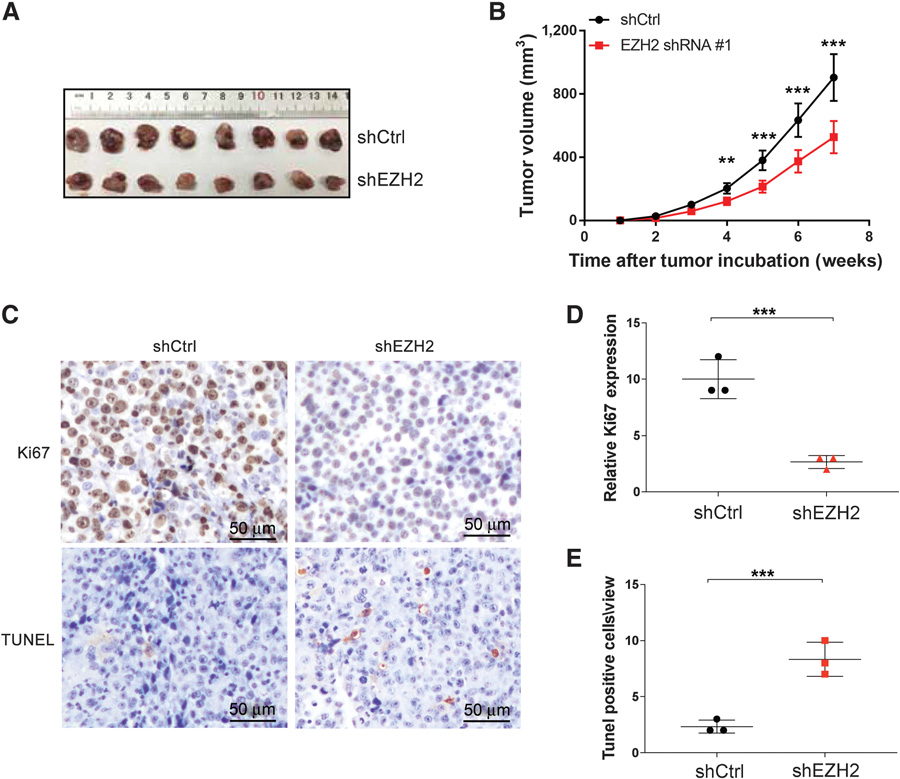
为了深入了解EZH2和H3K27甲基化在膀胱癌中的作用,我们在膀胱癌细胞系中使用shRNA构建了两种不同的shRNA构建物,构建了2个独立稳定的EZH2敲除(KD)细胞系(图3D)。值得注意的是,与野生型(WT)细胞相比,EZH2 KD细胞的增殖、集落形成和迁移能力受损(图3E-H)。此外,将EZH2 KD细胞移植到裸鼠体内形成的肿瘤比WT细胞更小(图4A和B)。与shRNA对照异种移植物相比,EZH2 KD异种移植物减少了细胞增殖标记物Ki67染色,但增加了细胞死亡标记物TUNEL染色(图4C-E)。总之,EZH2在尿路上皮癌细胞的干细胞获得中起着关键作用。
4)EZH2塑造膀胱癌细胞粘附和干细胞基因的表观遗传景观
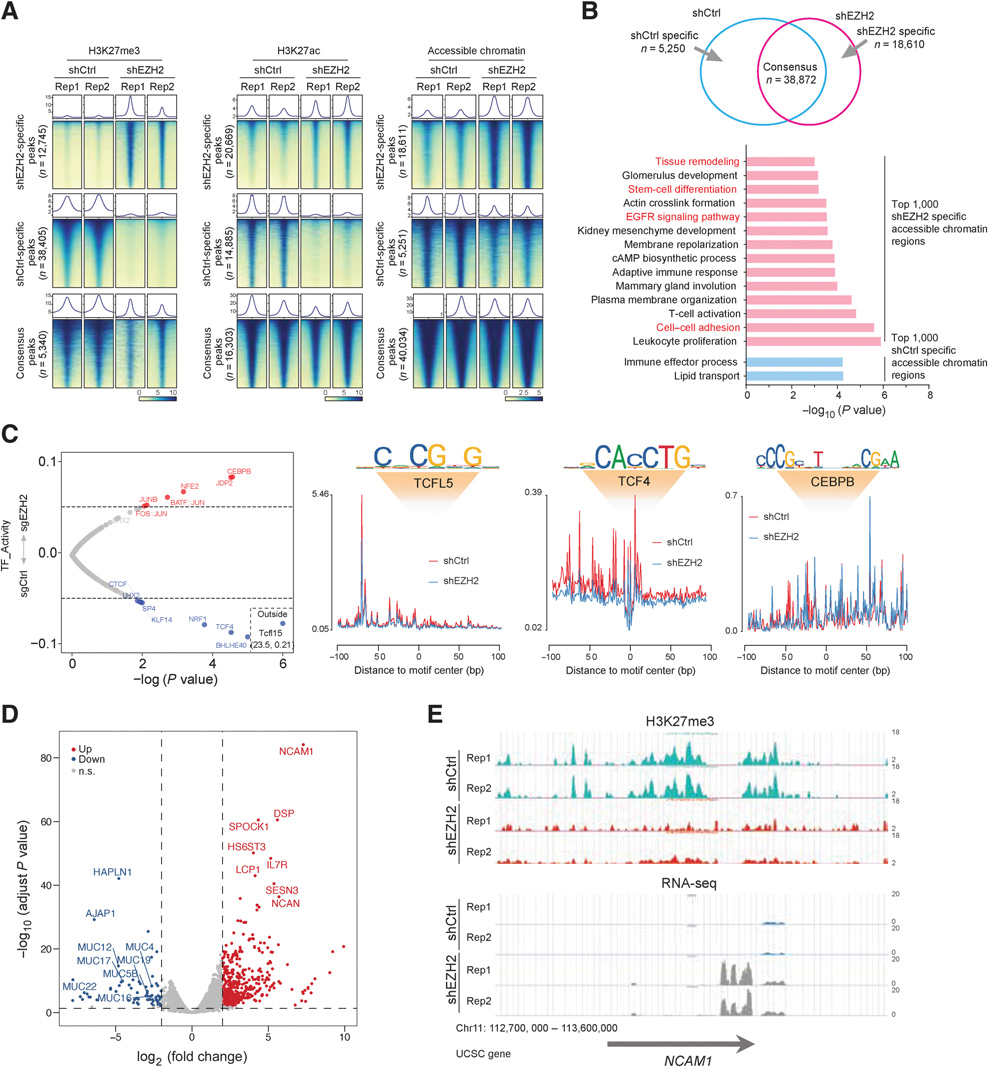
我们进行了CUT&Tag检测和ATAC-seq检测WT和EZH2 KD细胞中的H3K27me3, H3K27ac和可访问染色质(图5A)。EZH2的耗尽导致68%的H3K27me3峰的损失,这与其H3K27me3的催化活性相一致(图5A)。此外,EZH2的KD增加了55%的H3K27ac峰,而EZH2缺失时,可接近的染色质峰增加了29%。总体而言,EZH2的缺失与H3K27ac的显著增加和开放染色质的轻微增加是一致的,这是活性基因转录的标记(图5A)。
接下来,我们确定了EZH2 KD细胞中易接近染色质的相关功能。我们将可接近的染色质峰分为WT特异性、一致性和EZH2特异性峰(图5B)。与这些峰值相关的GO注释表明,它们与细胞粘附、EGFR信号通路、干细胞分化和组织重塑有关(图5B)。为了确定膀胱癌中EZH2调节的TFs,我们进行DNA足迹分析。我们在EZH2耗尽后观察到一些差异有统计学意义的TFs,如TCF4、TCFL5、CEBPB等,提示EZH2可能通过这些因子调控细胞的干性(图5C)。
为了进一步了解EZH2在膀胱癌复发中的机制,我们进行了RNA-seq,发现EZH2缺失后,大量据报道维持癌细胞干细胞的粘蛋白基因被下调(图5D),表明EZH2可能通过粘蛋白基因维持癌细胞干细胞。此外,EZH2的缺失导致NCAM1的表达显著增加。EZH2缺失后,NCAM1启动子和基因体中的H3K27me3水平显著降低,表明EZH2对该基因的直接调控(图5E)。总之,EZH2的KD减弱干细胞基因的表达,并直接激活NCAM1转录,促进膀胱癌细胞的细胞粘附。
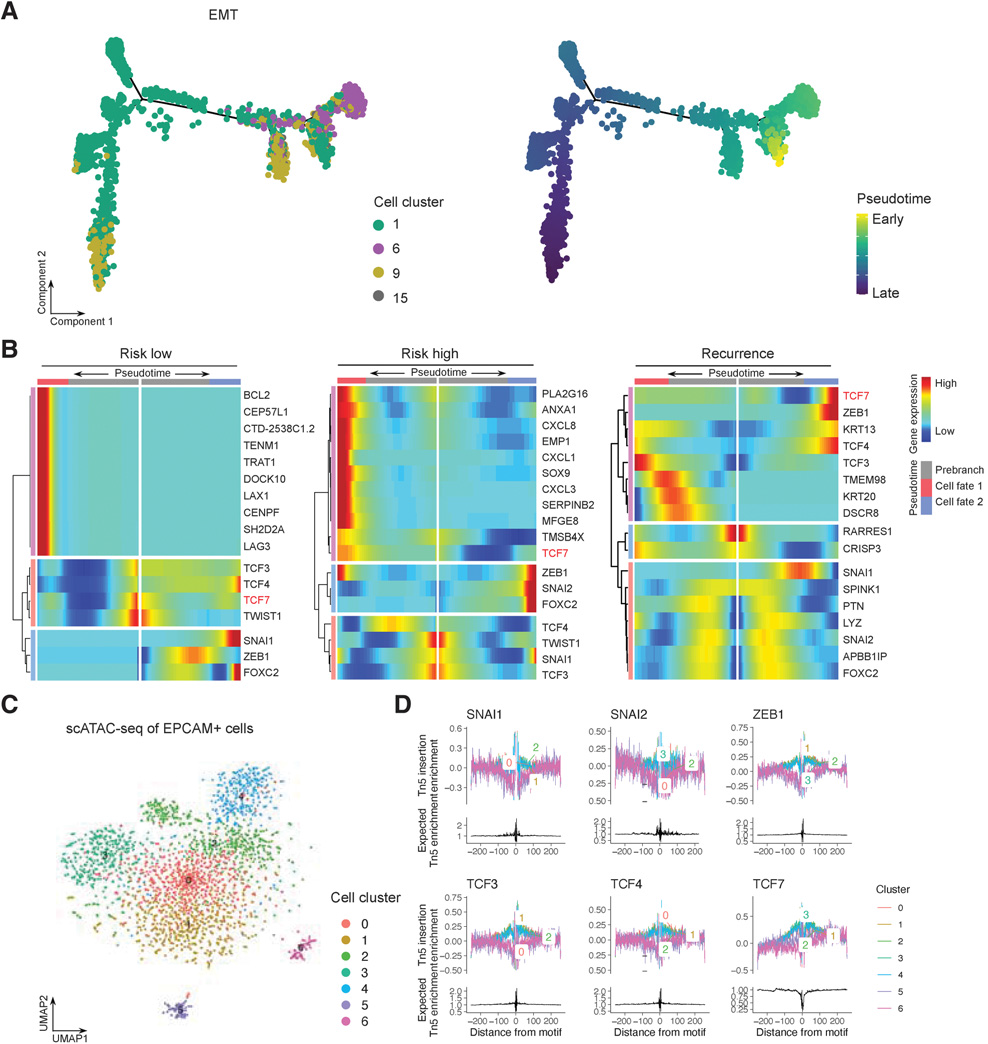
非免疫细胞簇1、6、9和15为上皮细胞(图2A)。根据转录组学,我们将这些细胞分为风险低、风险高和复发性膀胱癌亚型,以计算重建假时间路径(图6A)。我们在每个条件下鉴定了具有伪时间路径的共表达基因,发现几个诱发EMT的转录因子,如TCF3、TCF4、TWIST1、SNAI1和FOXC2,都与EMT轨迹相关(图6B)。这些诱导EMT的转录因子可以直接或间接结合E-cadherin启动子,抑制其转录,从而促进EMT。有趣的是,TCF7的表达模式与诱导EMT的TFs相似(图6B)。为了进一步了解TF在EMT中的调控网络,我们分析了上皮细胞的scATAC-seq数据,确定了7个亚群(图6C)。各亚群的DNA足迹显示,TCF7基序在0 ~ 3亚群中与其他诱导EMT的TFs一起富集,提示TCF7可能与SNAI1/2、TCF3/4、ZEB1等其他诱导EMT的TFs调控和诱导EMT的方式相同(图6 D)。
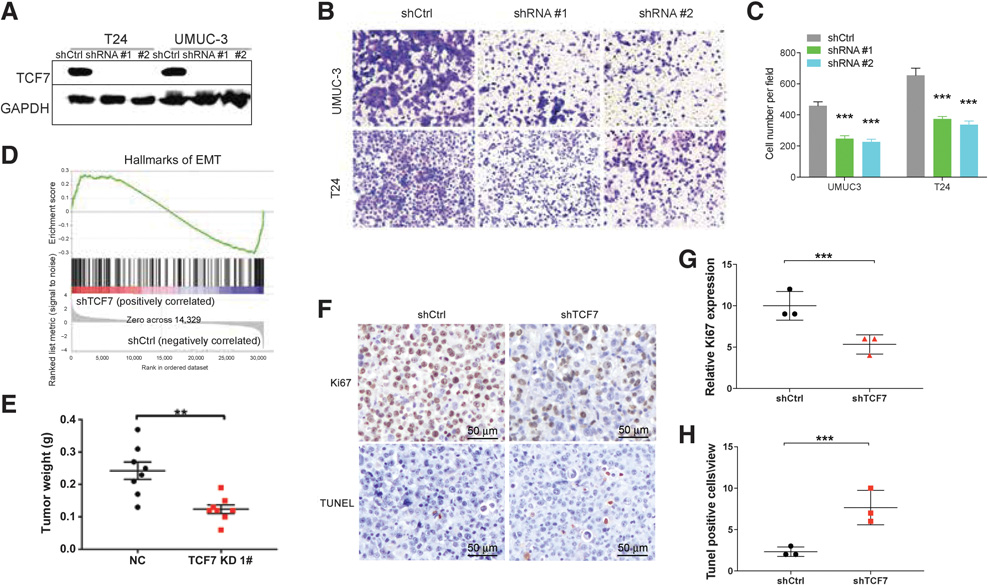
为了验证TCF7在膀胱癌中的功能,我们在膀胱癌细胞中使用shRNA介导的TCF7 KD(图7A)。TCF7的缺失导致细胞迁移和侵袭能力受损(图7B和C),表明TCF7 KD减弱了这些细胞中的EMT。为了深入了解TCF7在EMT中的功能,我们在TCF7 KD细胞中进行了大量RNA测序。GO富集分析表明TCF7靶基因与EMT标志基因相关(图7D)。与TCF7在EMT中的作用一致,清除TCF7可阻断裸鼠肿瘤的生长(图7E)。TCF7 KD肿瘤在体内表现出细胞增殖受损和凋亡细胞死亡增强(图7F-H)。总之,这些结果表明TCF7可以启动EMT并促进肿瘤生长。
结论:我们使用scRNA-seq进行转录组分析,使用scATAC进行染色质染色质景观分析,并生成了一个包含50000多个膀胱癌细胞的大型数据集,这些细胞来自13名膀胱癌患者,分为低风险、高风险和复发组。基于这个大数据集,我们描述了EZH2在CSC亚群中的功能,以及TCF7在EMT中的功能。我们相信我们的研究和分析方法为CSCs和EMT在膀胱癌研究领域的进一步研究提供了丰富的资源。
参考文献:Wang H, Mei Y, Luo C, et al. Single-Cell Analyses Reveal Mechanisms of Cancer Stem Cell Maintenance and Epithelial–Mesenchymal Transition in Recurrent Bladder Cancer[J]. Clinical Cancer Research, 2021. doi: 10.1158/1078-0432.CCR-20-4796






