单细胞测序揭示了乳腺癌淋巴结转移的细胞异质性和转录组谱
乳腺癌淋巴结转移的分子机制尚不清楚。通过单细胞测序,我们研究了来自15对原发肿瘤和腋窝淋巴结样本的96796个单细胞的转录组谱。我们鉴定了9个癌细胞亚群,包括CD44+ / ALDH2 +/ALDH6A1+乳腺癌干细胞(BCSCs)。重要的是,BCSCs只存在于原发肿瘤中,并演化为浸润到淋巴结的转移瘤。此外,转录组数据提示,NECTIN2-TIGIT介导了转移性乳腺癌细胞与肿瘤微环境(TME)细胞之间的相互作用,促进了免疫逃逸和淋巴结转移。这项研究是第一次使用单细胞RNA测序描述乳腺癌淋巴结转移的转录组谱。我们的发现为乳腺癌转移的机制提供了新的见解,并对开发新的疗法来抑制乳腺癌转移的起始有意义。本文于2021年10月发表于“Oncogenesis”(IF=7.485)。
技术路线
结果
1)原发性乳腺癌和淋巴结的单细胞转录组分析
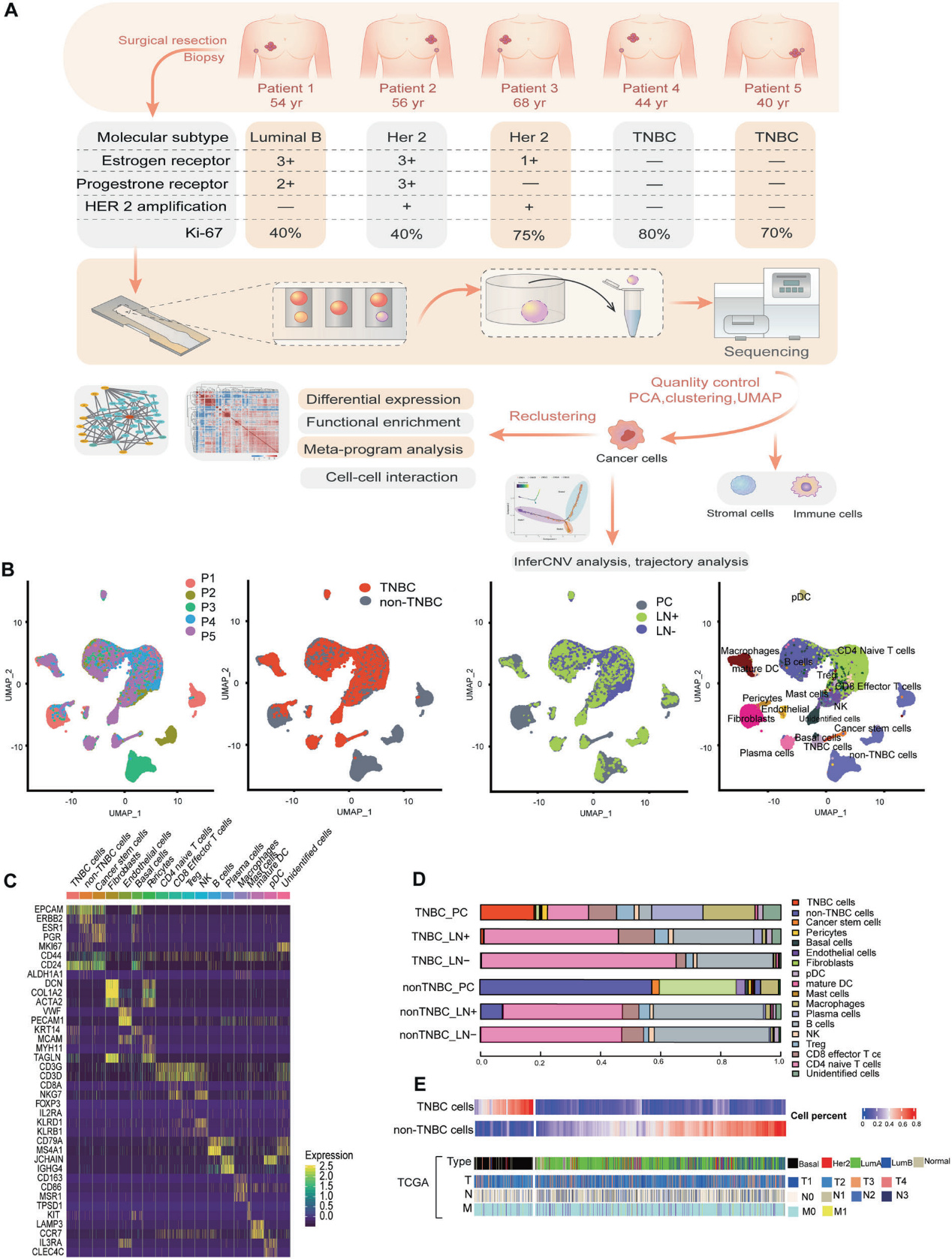
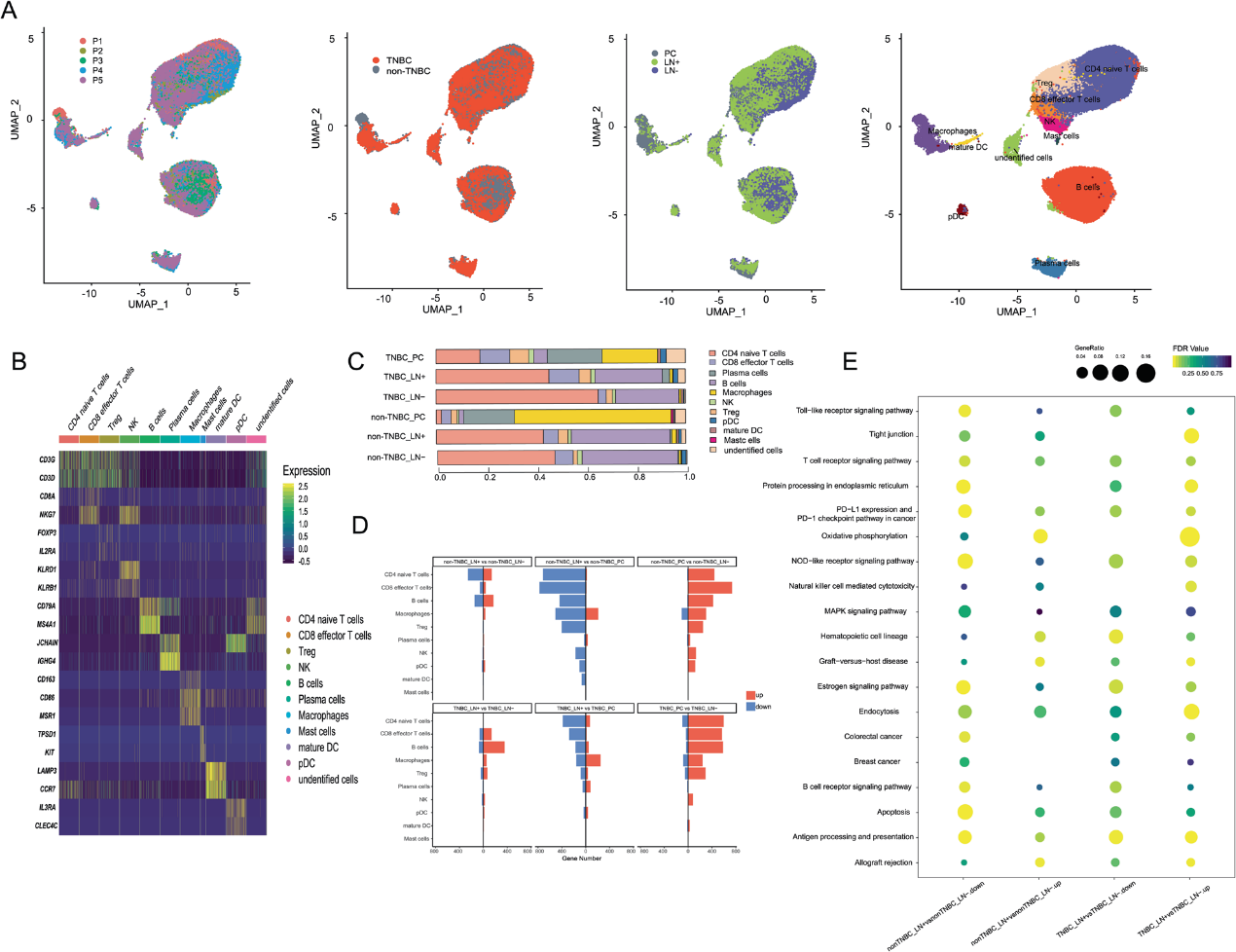
我们从南京医科大学第一附属医院的5例患者中,在治疗性手术和腋窝淋巴结清除后立即获得组织。这些组织样本代表来自患者同侧腋窝的成对原发肿瘤和淋巴结(图1A)。为了获得乳腺癌的全面转录组情况,我们对15个样本进行了单细胞RNA测序(scRNA-seq)。我们共获得来自原发癌组织的27,028个单细胞和来自腋窝淋巴结的69,768个单细胞(图1B)。我们将所有合格的细胞分为18种细胞类型(图1B, C)。CD44 + / ALDH2 + /ALDH6A1 +组被定义为乳腺癌干细胞(BCSCs)。接着,我们分析样品内部组成(图1D)。我们还采用反褶积分析对TCGA数据库中样本的细胞组成进行分析,结果显示样本中TNBC和非TNBC细胞的比例(图1E)。该scRNA-seq数据具有TNBC细胞的特征,这些特征后来被用于定义TCGA样本的分子亚型。
2)乳腺癌细胞的克隆分析
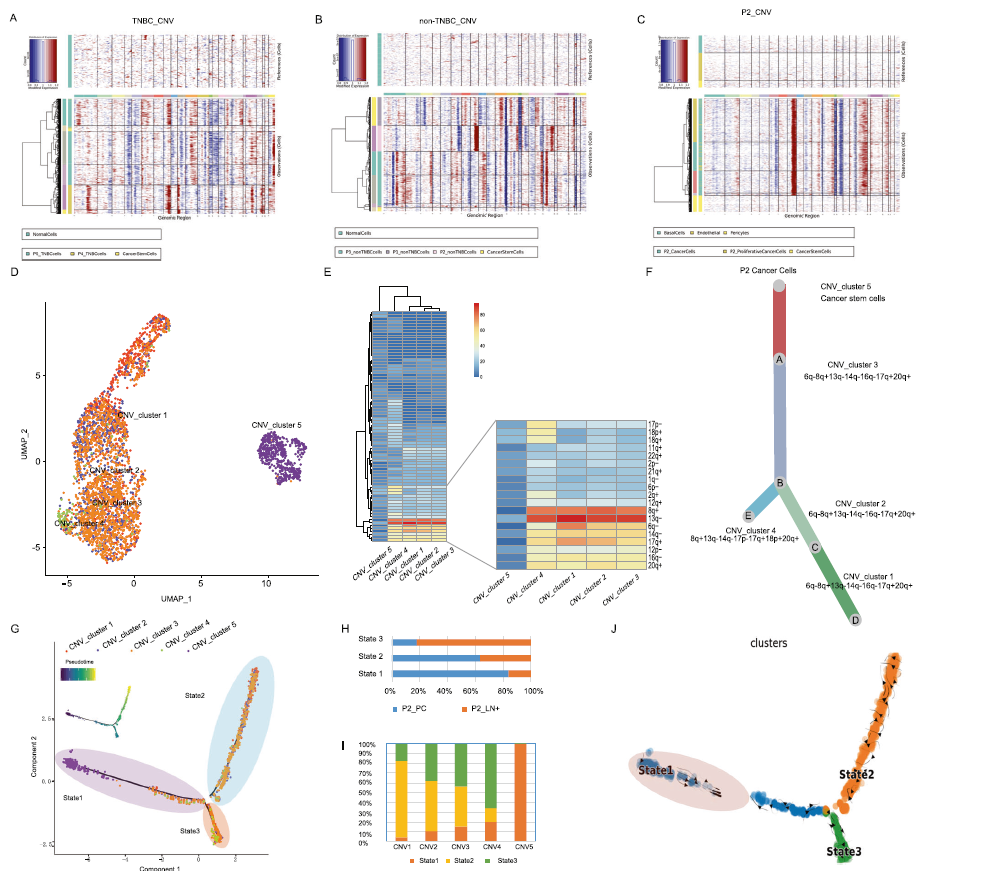
为了探索乳腺癌的基因组谱,我们应用inferCNV算法分析单个细胞的拷贝数变异(CNVs)。我们发现,与其他癌细胞亚群相比,BCSCs突变较少(图2A, B)。根据BCSCs的细胞计数,选择患者2进行进一步研究。5个CNV_cluster被分类,CNV_cluster 5与BCSC一致(图2C),随后通过UMAP可视化证实了BCSC独特的CNV特征,并将BCSC与其他恶性细胞分离(图2D)。根据inferCNV分析,Heatmap显示了5个CNV_簇的突变概况(图2E)。基于CNV突变图谱进行进化研究和轨迹分析,推断乳腺癌的发展过程(图2F, G)。BCSCs在轨迹过程的早期就被识别出来,并演化为两个癌细胞分支(图2F)。我们的CNV图谱在转录组水平上进一步证实了这一结论,CNV_cluster1和CNV_cluster4与淋巴结转移相关的突变最多。内部组成揭示了肿瘤部位与进化状态的关系,转移性肿瘤细胞处于晚期,而原发病变细胞较多,处于早期(图2H, I)。RNA-velocity证实BCSC具有干性,并根据mRNA成熟度沿其演化轨迹进行演化(图2J)。
3)转移性乳腺癌细胞的转录组特征
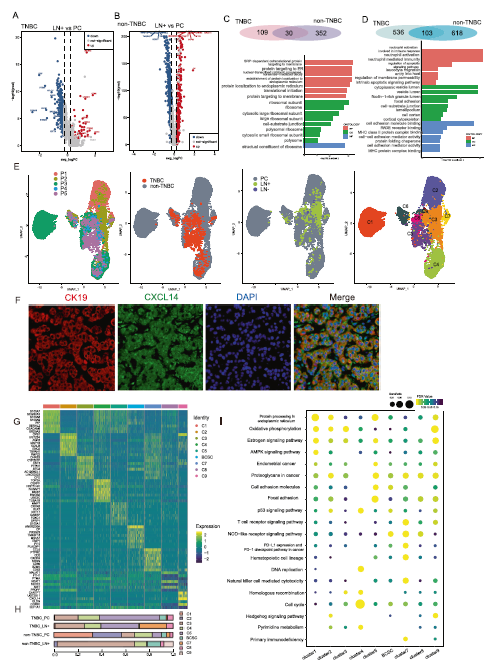
通过放大癌细胞,我们解剖了DEGs,分别鉴定了原发肿瘤和转移性肿瘤细胞富集的功能和途径。对于转移性TNBC细胞,B2M、CD52、PTMA和GZMK是最显著的DEGs,功能富集显示了免疫治疗的潜在敏感性,在转移性TNBC细胞中免疫相关项目高度富集(图3A)。对于转移性非TNBC细胞,CXCL14、STC2、CST3和RAMP3过表达(图3B)。我们的转录组图谱显示了肿瘤位点和分子亚型之间的异质性,Venn图显示了肿瘤间的相似性,在两个TNBC和三个非TNBC样本的转移中发现了30个基因上调,而在两个分子亚型中发现了103个基因下调(图3C,D)。免疫荧光染色检测淋巴结非TNBC细胞过表达基因(图3F)。不同肿瘤位点和分子亚型的转录组差异揭示了乳腺癌肿瘤间和肿瘤内的异质性,而一些发现与其他实体肿瘤的研究结果相呼应,表明乳腺癌细胞的侵袭行为来源于淋巴结转移。
UMAP根据乳腺癌亚型、患者、样本来源和细胞簇对所有合格的癌细胞进行可视化(图3E)。所有15个样本的癌细胞被过滤后分为9个簇,其中C6具有BCSC生物标志物,包括CD44和ALDH(图3G)。我们发现,在TNBC淋巴结转移中,C7细胞最多,而在非TNBC淋巴结转移中,C3细胞占多数(图3H)。参照C7的转录组图谱,CCL5、PTPRC、CD2、CXCR4和SRGN的表达靠前。富集分析旨在更好地了解肿瘤细胞簇的生物学功能,展示其在合成、代谢、细胞周期、免疫应答、信号转导等方面的生物活性(图3I)。据我们所知,这是第一个基于整合单细胞RNA测序从转录组角度揭示转移性乳腺癌差异的研究。
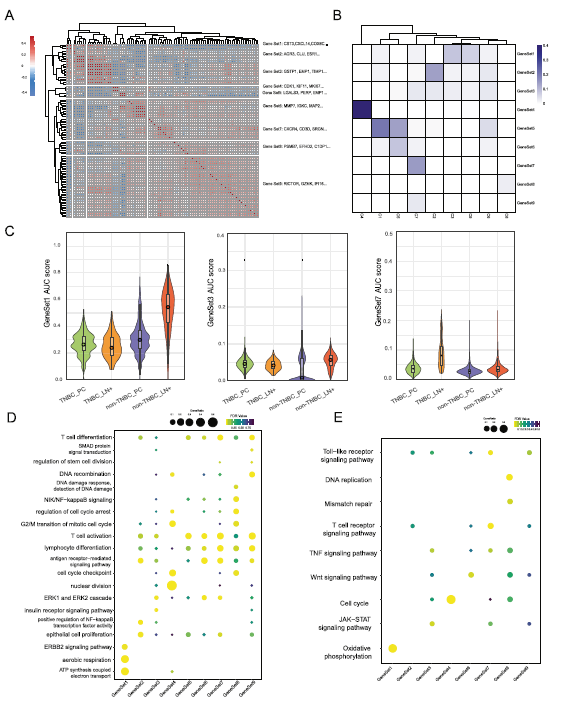
4)鉴别乳腺癌的恶性程序
我们应用cNMF将肿瘤异质性的完整转录组谱划分为9个元程序,并根据这些元程序的特征基因对每个肿瘤细胞簇进行评分(图4A-C)。每个元程序对应一个具有内在相似性或相关性的基因集,包括能量代谢(元程序1;CST3, CXCL14, COX6C),癌症相关信号通路(元程序3;GSTP1, EMP1, TIMP-1),免疫激活和应答(元程序5;LGALS3, PERP和EMP1),淋巴细胞活化(元程序7; CXCR4、CD3D SRGN),t细胞活化和DNA修复(元程序9;RICTOR、GZMK IFI16)。元程序可以更好地理解乳腺癌分子亚型和肿瘤部位的异质性和相似性。C3在元程序 1中得分较高。根据功能富集(图4D, E),该元程序富集了物质代谢和ERBB2信号通路。与转移性非TNBC细胞不同,该元程序分析表明,转移性TNBC细胞在激活淋巴结的免疫识别中具有活性(图4B、D和E)。元程序 7中富集的最重要功能包括淋巴细胞分化和激活,以及淋巴细胞介导的免疫。元程序9富集的主要功能包括t细胞受体信号通路、肿瘤中PD-L1表达和PD-1 checkpoint通路、PI3K-Akt信号通路。
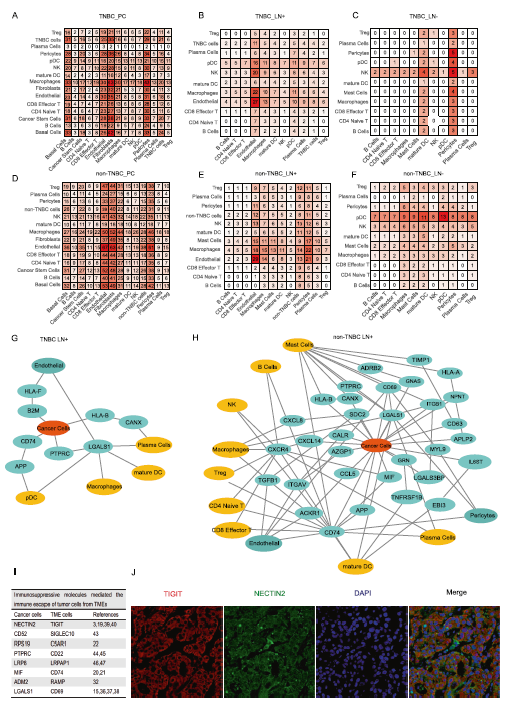
为了证明TME中免疫细胞的转录组特征,我们使用UMAP可视化来自5个原发病灶和10对腋窝淋巴结的所有免疫细胞(图5A,B)。TME在肿瘤部位的细胞组成和基因表达上都存在差异(图5C,D)。与淋巴结内的免疫细胞相比,免疫细胞浸润到原发肿瘤细胞时的生物活性更强烈,并且在不同分子亚型的转移中免疫反应的功能差异显著(图5D,E)。基于多细胞相互作用网络分析工具包(NATMI)分析,我们识别了不同样品来源的细胞类型之间的细胞间通信(图6A-F)。转移性癌细胞和免疫细胞之间的配体-受体对是淋巴结TME的重要组成部分。CD52-SIGLEC10和PTPRC-CD22参与了TNBC和非TNBC淋巴结转移(图6G, H)。基于淋巴结内癌细胞与免疫细胞间的DEGs和通信,我们的研究结果不仅解释了乳腺癌细胞在不同部位的免疫逃逸机制,也为未来针对淋巴结转移恶性过程的治疗提供了靶点(图6I)。通过与TIGIT相互作用,我们发现NECTIN2在转移性非TNBC细胞中高表达,通过与TIGIT相互作用,发现转移性癌细胞抑制T细胞激活,抑制免疫应答。我们进一步进行免疫荧光染色,证实了癌细胞与CD8+效应T细胞之间的细胞-细胞相互作用(图6J)。
结论:我们描述了乳腺癌干细胞的特征,描述了肿瘤发生的进化过程,确定了乳腺癌转移特异性亚簇的转录组谱,并详细地证明了肿瘤内部和肿瘤间的异质性。本研究为乳腺癌淋巴结转移的进一步研究提供了新的思路,为乳腺癌患者的个体化治疗奠定了基础。
参考文献:Xu K, Wang R, Xie H, Hu L, Wang C, Xu J, Zhu C, Liu Y, Gao F, Li X, Wang C, Huang J, Zhou W, Zhou G, Shu Y, Guan X. Single-cell RNA sequencing reveals cell heterogeneity and transcriptome profile of breast cancer lymph node metastasis. Oncogenesis. 2021 Oct 5;10(10):66. doi: 10.1038/s41389-021-00355-6. PMID: 34611125; PMCID: PMC8492772.






