piRNAclusterDB 2.0:一个piRNA簇数据库
PIWI 相互作用的 RNA (piRNA) 及其伙伴 PIWI 蛋白保护动物种系免受转座因子的侵害,并在生育能力中发挥关键作用。过去的大量研究揭示了 piRNA 通路的许多附加功能,主要用于调节一系列靶序列,包括转座因子、蛋白质编码基因和长链非编码 RNA。这些靶标可以在转录水平和转录后进行调节,并且通过 piRNAs 的序列互补性来识别,piRNAs 将其相关的 PIWI 蛋白引导至其目的地。虽然在脊椎动物中 PIWI/piRNA 途径主要局限于生殖系,但在节肢动物和软体动物等无脊椎动物群中,piRNA 还普遍存在于体细胞组织中。
产生 piRNA 的基因座,称为 piRNA 簇,被认为位于 PIWI 通路的中心。这些基因座从一条或两条 DNA 链转录成大的前体 RNA,当它们加载到 PIWI 蛋白上时,它们又被加工成 23-31 nt的成熟 piRNA。在迄今为止研究的所有物种中,piRNA 簇进化迅速,在进化时间尺度上出现和消失的速度相当快,在序列水平上呈中性进化。因此,每个物种都有一组独特的 piRNA 簇,在谱系之间共享不同数量的同源簇。piRNA 簇在物种内高度分化,在体细胞piRNA 簇在不同组织中表现出不同的表达水平。
近期,德国研究团队建了一个piRNA 簇数据库:piRNAclusterDB 2.0(https://www.smallrnagroup.uni-mainz.de/piRNAclusterDB),该数据库相关文章刊登在Nucleic Acids Research期刊(IF=16.971),题名为piRNAclusterDB 2.0: update and expansion of the piRNA cluster database。该数据库收集了51 个物种的 >350 个SRA数据集,总共包含 >15 000 个 piRNA 簇。其中包括软体动物、节肢动物、鱼类、两栖动物、爬行动物、鸟类和哺乳动物。
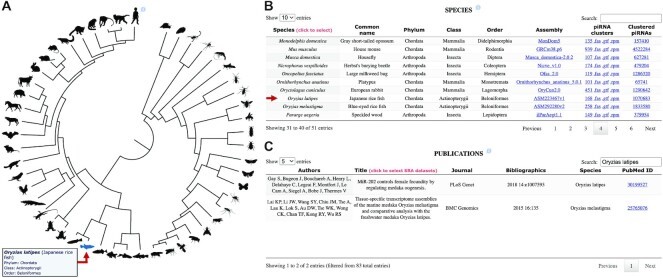
界面的主要入口点是物种选择器,它表示为一个交互式系统发育树,包含有关分类学、piRNA 簇数和簇 piRNA 序列总量的附加信息。还提供了指向我们分析中使用的相应基因组组装数据的链接,包括基因组、基因集 (GFF) 和重复掩码文件。染色体上 piRNA 簇位置的图形表示与每个物种中 piRNA 簇的数量相关联。此外,我们还提供了与数据库的小 RNA (sRNA) 数据集相关的所有出版物的完整列表,包括 PubMed ID 和直接链接。
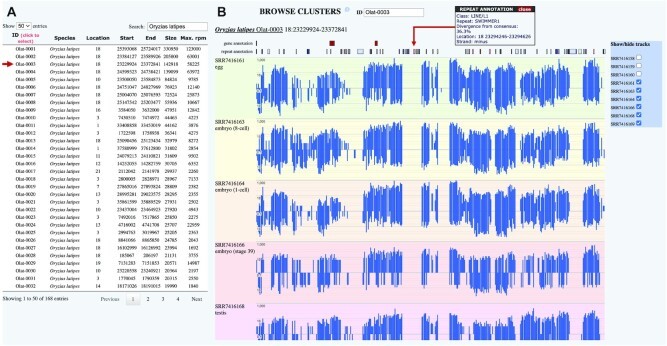
选择物种后,用户在浏览器中沿着所选物种的所有数据集浏览产生 piRNA 的基因座。可用基因座在列表中提供,并带有可选择的 piRNA 簇 ID,其中包含有关位置、大小。最后,可以单独选择和取消选择每个 SRA 数据集,以生成跨不同数据集、组织或发育阶段的 piRNA 簇表达的自定义视图。
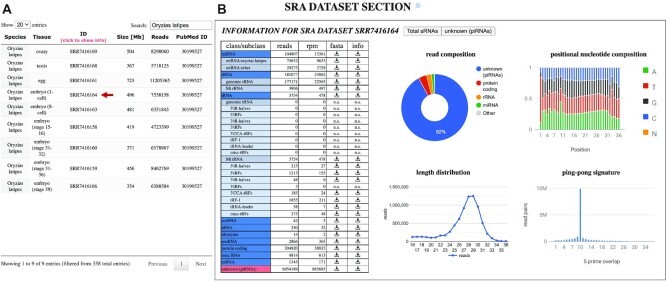
数据库的第三部分详细分析了来自选定物种的每个 SRA 数据集的处理、过滤、映射和注释的 sRNA 读数,这些数据由 unitas生成。SRA 数据集可从一个列表中选择,该列表包括有关来源组织、读取次数和相关出版物的 PubMed ID 的信息。对于每种带注释的 RNA 类型,例如 miRNA、rRNA、tRNA 衍生的 sRNA、lncRNA、mRNA 等。此外,还为总 sRNA 读数和 piRNA 读数提供了关于读数组成、长度分布、位置核苷酸组成和乒乓签名的图形输出,从而可以深入了解每个 SRA 数据集的 sRNA 构成以及 piRNA 对总读取池。