AlphaFold蛋白质结构数据库
蛋白质是具有重要生物学功能的重要大分子,因此广泛参与多项研究活动以及医学和生物技术应用,从抗击传染病到应对环境污染 都发挥重要作用。了解蛋白质原子的三维排列可为理解蛋白质功能的作用和机制提供重要线索。然而,虽然通用蛋白质资源 (UniProt) 存档了近 2.2 亿个独特的蛋白质序列,但蛋白质数据库 (PDB) 仅保存了超过55000种不同蛋白质的180000多个3D结构,因此蛋白质3D结构解析严重限制了序列空间的覆盖范围支持全球生物分子研究。
用实验确定的高分辨率结构实现对序列空间的更高覆盖率是非常劳动密集型的。它通常需要大量的试验和错误,例如,找到合适的构建体或蛋白质适合结晶的条件。尽管电子冷冻显微镜和用于结构确定的混合和综合方法 (I/HM) 领域的最新进展加快了结构确定的步伐,但已知蛋白质序列与实验蛋白质结构之间的差距仍在继续扩大。缩小这一差距的一种方法是预测数百万种蛋白质的结构。越来越多的研究人员部署人工智能 (AI) 技术,仅根据氨基酸序列计算预测蛋白质的结构。
AlphaFold 是由 DeepMind 开发的 AI 系统,可根据氨基酸序列对蛋白质结构进行最先进的预测。AlphaFold的准确性和速度允许创建一个大规模的结构预测数据库。它将使生物学家能够获得几乎任何蛋白质序列的结构模型,从而改变他们解决研究问题的方式并加速他们的项目。AlphaFold DB(https://alphafold.ebi.ac.uk)是基于AlphaFold算法构建的蛋白质3D机构预测数据库。AlphaFold DB 的初始版本包含超过360000个预测结构、相应的元信息和置信度指标。预测目前涵盖UniProt参考蛋白质组中16-2700 个氨基酸长度范围内的大多数序列(以及覆盖更长人类蛋白质的 1400 个残基片段)。
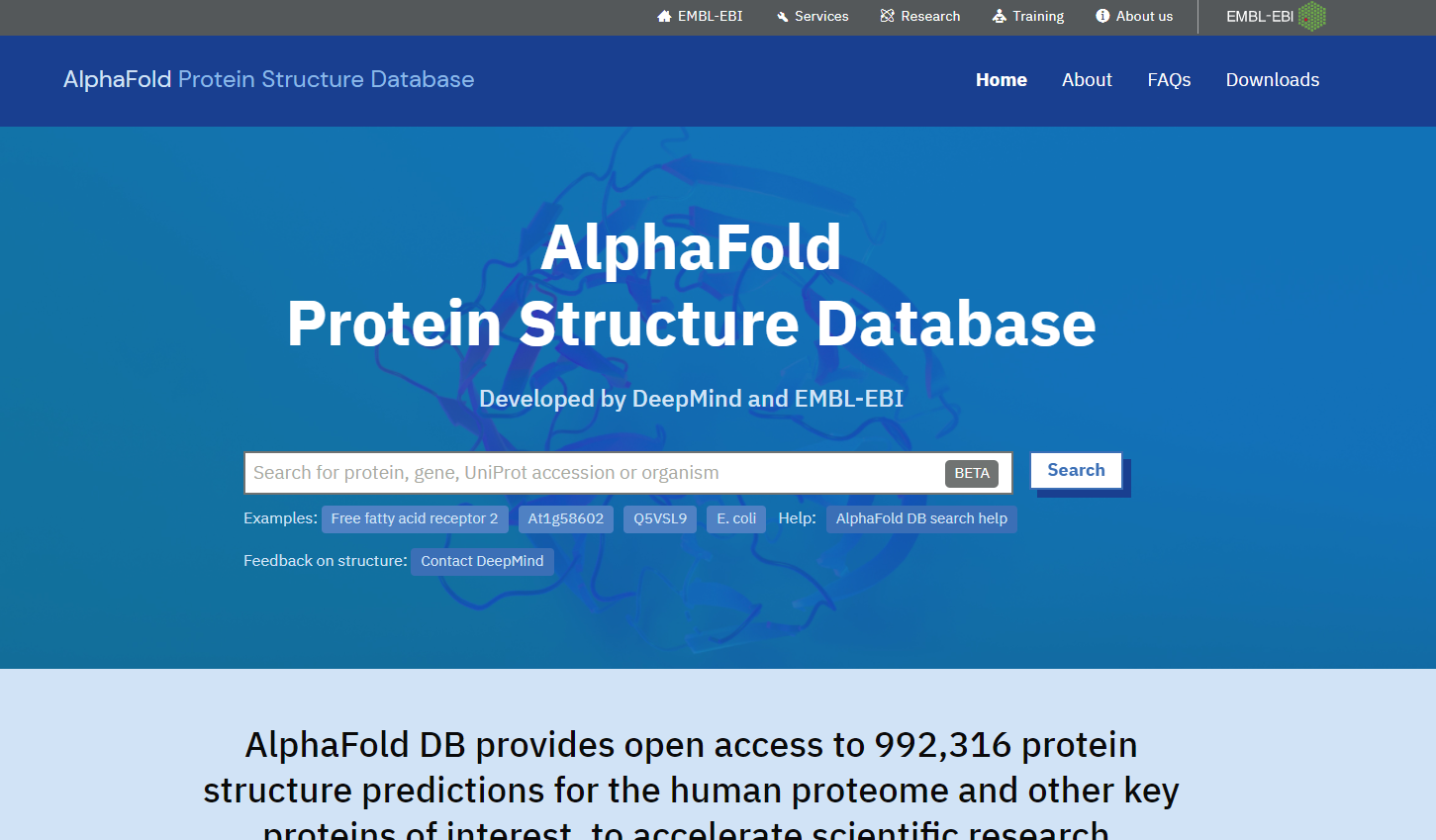
AlphaFold DB 通过网页提供对其预测的便捷访问。这些页面包含对 AlphaFold 系统的介绍,解决最常见的问题,允许批量下载完整的蛋白质组,并提供搜索引擎以查找特定于感兴趣蛋白质的页面。用户可以通过基因名称、蛋白质名称、UniProt 登录或生物名称进行搜索。
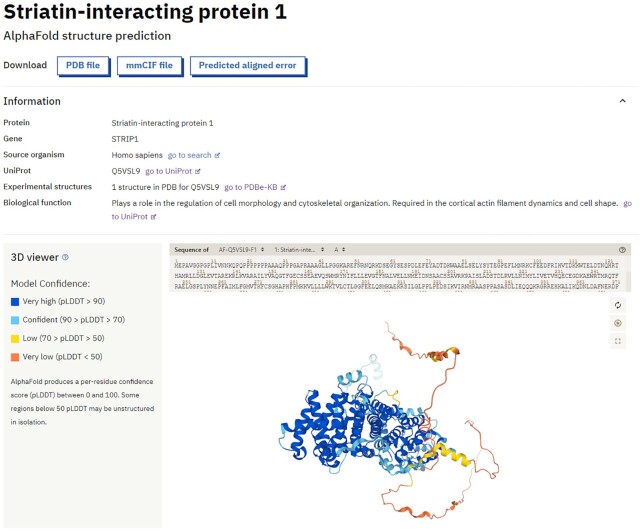
每个蛋白质都有一个专门的结构页面,显示基本信息(来自 UniProt和 PDBe)和 AlphaFold 模型的三个独立输出。前两个输出是3D坐标和每个残基置信度度量 pLDDT,用于在集成的3D分子查看器 Mol中对模型的残基进行着色。模型置信度可能会沿着一条链发生显着变化,因此在解释结构特征之前分析置信度度量是必不可少的。
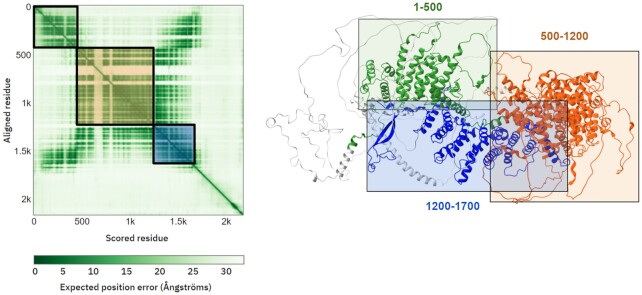
第三个输出是成对置信度预测,它有助于评估相对域位置和方向的可靠性以及蛋白质的全局拓扑结构。该图由成对的 PAE 值着色,它可以帮助用户识别哪些域具有可靠地预测的相对于彼此的位置和方向,其中深绿色表示高置信度。在绘图中选择一个区域也会在 3D 查看器中突出显示序列的相应部分。