基因组和转录组学分析揭示甲状旁腺癌的分子特征
基因组和转录组学分析增强了许多癌症的诊断和治疗选择。然而,甲状旁腺癌的分子特征在很大程度上仍未被探索,从而限制了新的治疗干预措施的发展。在此,作者对50例甲状旁腺组织(肿瘤12例,腺瘤28例,正常组织10例)进行了基因组和转录组测序,以研究甲状旁腺癌的内在和比较分子特征。作者在细胞分裂周期73(cell division cycle 73,CDC73)中证实了多个2-hit突变模式,并收敛于双等位基因失活,从而质疑其他基因是否存在第二个hit。此外,在具有生殖系突变的拷贝中,CDC73的等位基因特异性抑制表明在肿瘤发生之前存在选择性压力。转录组学分析发现,与腺瘤和正常组织相比,癌组织中E2F靶点、KRAS和TNF-α信号通路以及上皮-间质转化通路的表达上调。基于肿瘤特异性基因的分子分类模型将肿瘤与腺瘤和正常组织明显分开,在2例恶性潜能不确定的患者中证明了该模型的临床实用性。更深层次的基因表达分析和功能预测提示Wilms瘤1(WT1)是CDC73突变甲状旁腺癌的潜在生物标志物,并通过免疫组化进一步验证。总的来说,作者的研究揭示了甲状旁腺癌的基因组和转录组图谱,并可能有助于指导未来的精准诊断和治疗改进。本研究于2023年6月发表于《Experimental & Molecular Medicine》期刊上,IF:12.8,二区。
技术路线
主要研究结果
1、甲状旁腺癌的临床及生化特征
总共收集了三组共50例甲状腺组织,其中甲状旁腺癌12例,甲状旁腺腺瘤28例,正常甲状旁腺组织10例,用于基因组和转录组分析(图1)。对于基因组图谱,作者对匹配血液样本的癌组织进行了WES分析。对于转录组分析,对所有3组进行RNA测序,所得数据用于基因和基因集水平的分析。
作者首先分析了甲状旁腺肿瘤的临床及生化特征(表1)。而正常组个体年龄最小(平均年龄=38.4),癌组个体年龄小于腺瘤组个体(平均年龄=61.3岁),代表了普遍存在的遗传危险因素(即CDC73基因座杂合性丢失)。作者发现在所有三组中女性占优势(67-89%),没有统计学意义上的组特异性差异。甲状旁腺腺瘤和癌变患者术前甲状旁腺激素(PTH)和血钙水平均高于正常人群,且均以癌变患者最高。同样,3组患者的血磷水平也存在差异,但肿瘤患者的血磷水平最低。临床遗传学检测(血液的靶向测序)在12例肿瘤患者中发现6例CDC73基因胚系突变。12例甲状旁腺癌患者中,3例出现远处转移,2例随访期间出现局部复发。这些发现与已知的甲状旁腺癌的临床特征和预后是一致的。
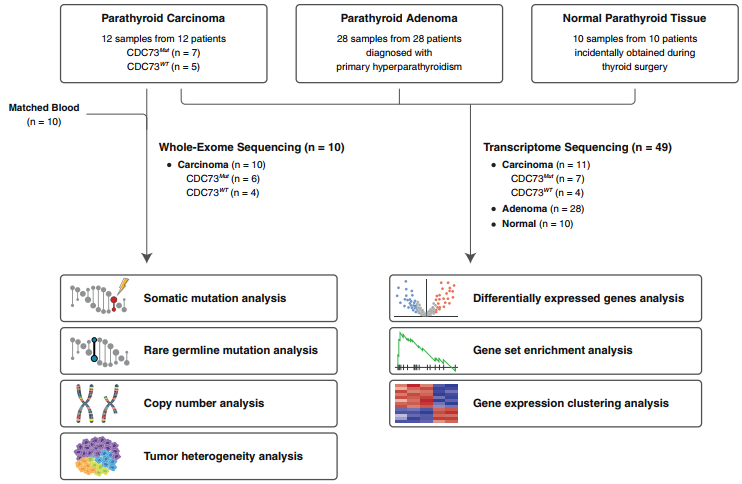
图1 研究工作流程示意图概述

2、甲状旁腺癌的基因组图谱
为研究甲状旁腺癌的基因组变异情况,作者在10例甲状旁腺癌样本中发现了体细胞突变,其中有匹配的血液样本(图2a)。非同义单核苷酸变异(SNVs)和插入-缺失(indels)的数目在18-848之间,中位数为59个,每兆碱基对应1.18个突变。除1例具有异常高突变计数(P5)的样本外,所有癌症的突变计数均低于其他癌症。
首先,作者检测CDC73的突变模式。6例(P5、P6、P11、P22、P32、P75)患者中存在(4个无义SNVs和7个移码缺失)的胚系或体细胞截短突变,考虑为CDC73-突变型(CDC73Mut)。作者发现胚系突变和体细胞突变的基因组位点明显分开(图2b);胚系突变位于Ras-like结构域的上游,Ras-like结构域对于与PAF1(聚合酶相关因子1)和染色质的相互作用至关重要,而体细胞突变集中在外显子1-2,表明转录本完全丢失。6例(P5、P6、P11和P22)患者中有4例出现明显的2次打击突变,导致CDC73双等位基因失活,称为Knudson’s two-hit假说(即1例胚系易感性和1例获得性体细胞突变)。1例患者(P32)仅有1个体细胞移码突变(CDC73p . R52Ifs * 9),但由于杂合性丢失(LOH),伴随着野生型等位基因的完全丢失,导致双等位基因失活(图2c)。1例患者(P75)仅存在种系截短突变(CDC73p . E130Gfs * 11),表现出与早期病理分类的交界性,预示着一个亚克隆体细胞突变的机会。总体而言,所有6例患者的突变模式均表现为截短CDC73导致的双等位基因失活,反对CDC73以外的基因中存在二次打击。
接下来,作者评估了4例携带野生型CDC73(CDC73WT)的(P65、P66、P68、P77)患者的突变谱。作者发现CDC73WT(中位数=29.0)患者的变异体数量低于CDC73Mut(中位数=152.5;p=0.0095,Mann-Whitney检验)患者(图2d),表明其基因组完整性较高。尽管样本量有限,作者在CDC73WT患者中观察到一些复发突变。在2例CDC73WT患者(P68为TP53 p . C3F , P77为TP53 p . H61R)中观察到TP53的体细胞错义突变,这两种突变在其他癌症中都曾被报道过,并且预期是有害的。尽管(p = 0.1333,Fisher精确检验)不具有统计学意义,但这可能意味着TP53突变在CDC73非依赖性甲状旁腺癌中的局限性。同样,在CCDC40(卷曲螺旋结构域包含40个)的3个体细胞突变中,在CDC73WT患者(P65和P77)中发现2个截短移码突变(CCDC40 p.K970Nfs * 51和CCDC40 p.G987Rfs * 96)。CCDC40突变是已知的原发性纤毛运动障碍的主要原因,但其与癌症的相关性尚未见报道。KMT2D、KRAS(in-frame delete)和FRAT2(WNT信号通路的FRAT调节因子2)的其他突变可能与甲状旁腺癌的发生有关;然而,证据仍然有限。
突变标签分析显示每个癌症组有两个不同的标签:CDC73Mut的SBS13(单碱基替换签名13)和CDC73WT的SBS6(图2e)。在CDC73Mut组中还发现了SBS1,后来被证实是P5仅此一个的签名,并且在每个CDC73Mut样本中都不存在。SBS13是CDC73Mut组发现的主要标志,已知其与活化的APOBEC胞苷脱氨酶相关,已被提议作为免疫治疗和靶向治疗的标志物。进一步的APOBEC富集分析也证实了CDC73Mut组中APOBEC的显著高相关性(图2f),这可能是CDC73Mut和CDC73WT肿瘤进展机制不同的另一条线索。
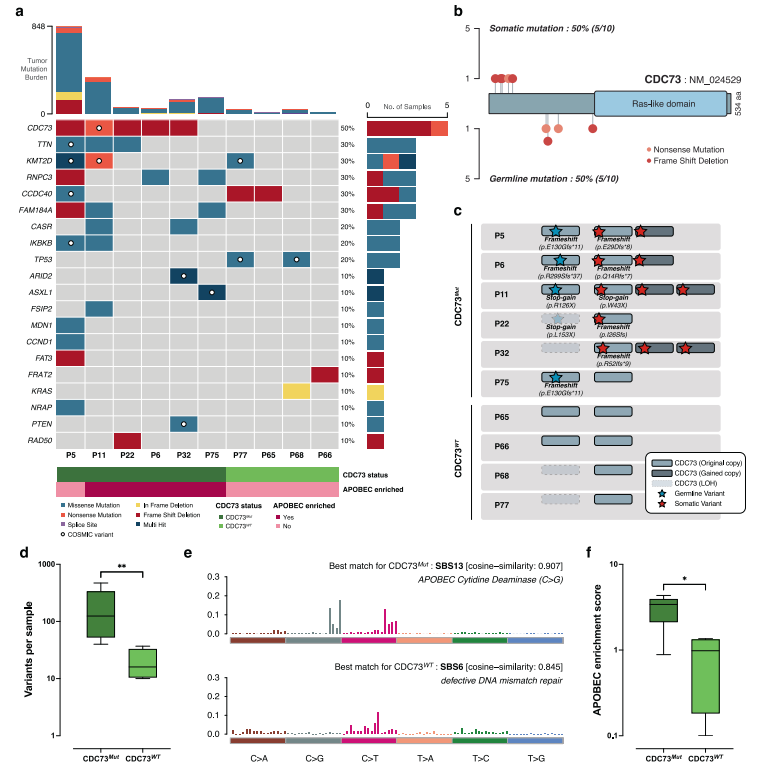
图2 甲状旁腺癌的基因组图谱
3、甲状旁腺癌的转录组学分析
使用49个组织的RNA测序数据(11个癌,7个CDC73Mut和4个CDC73WT;28个腺瘤;10例正常甲状旁腺组织),DEGs分析两种情况:癌vs正常和腺瘤vs正常(图3a)。作者发现整体基因表达谱在腺瘤中高度保守,表现出与正常甲状旁腺(Pearson相关系数r=0.982)基因表达谱的强相关性。相比之下,(Pearson相关系数r=0.943)在癌组织中的基因表达发生了明显的偏移。因此,在癌组织(n=1 136)中DEGs的数量多于腺瘤组织(n=33),其中26个DEGs在两者中差异表达。因此,鉴定出1110个癌症和7个腺瘤特异性DEGs。
通路水平分析发现,癌中富集了许多癌症标志性通路(图3b)。特别是,与正常样本(校正p值< 0.01和FDR < 0.25)相比,E2F靶点、G2M检查点、糖酵解、Myc靶点和上皮间质转化(EMT)相关的通路上调。在腺瘤中也观察到G2M和Myc靶通路的轻度上调,但KRAS信号和TNF-α信号下调,与癌相反(p< 0.003)。在腺瘤DEGs的GO富集结果中,包括即使是微小的1.5倍变化到正常的DEGs,也观察到属于细胞生长或神经元发育的基因上调。
进一步评估发现CDC73Mut和CDC73WT在通路激活方面存在差异。CDC73Mut中E2F靶基因的上调更为显著,这可能暗示针对较高突变负荷的DNA损伤应答(图3c)。而E2F和Myc靶点的激活,mTORC1和Hedgehog信号通路被普遍观察到,CDC73WT甲状旁腺癌表现出更强的EMT和氧化磷酸化的激活(图3d)。众所周知,EMT在肿瘤进展中的激活与肿瘤发生和血管生成密切相关。此外,上调的氧化磷酸化的组合可以有力地证明转移,更具体地说,混合E/M表型。事实上,CDC73WT队列中的其中一名患者在样本制备后出现了多处转移。尽管在其他CDC73WT患者中未发现转移的迹象,但考虑到这一结果,不能排除转移的可能性。
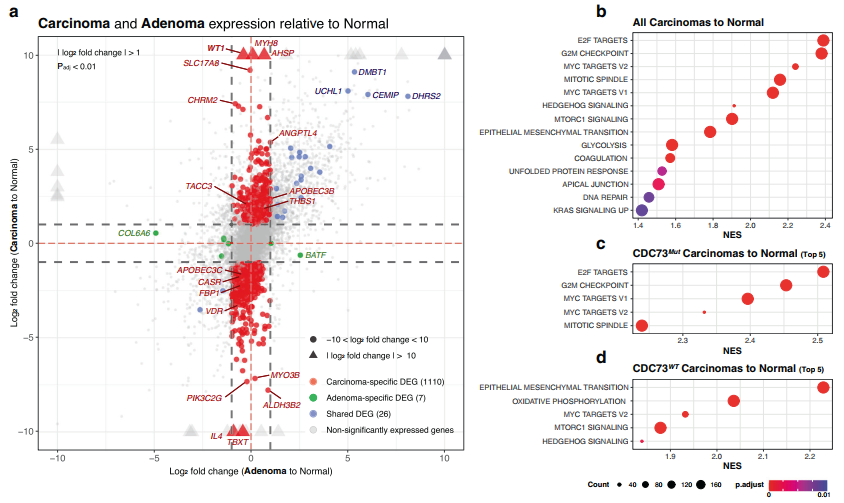
图3 甲状旁腺癌和腺瘤与正常组织的转录组学分析
4、CDC73等位基因失衡及等位基因特异性表达
如前所述,CDC73基因的2次点突变导致2个可分离的等位基因:一个携带胚系变异体(简称CDC73Germ),另一个获得体细胞突变(CDC73Som)。基因组和转录组图谱的联合分析可以检测DNA和RNA水平的不平衡,包括等位基因特异性拷贝数变化(CNAs)和两个等位基因之间的表达偏差。作者在1q31.2检测到频繁的(70%)等位基因特异性CNAs,包括CDC73的遗传性病变。值得注意的是,4个具有CDC73双打击突变(P5、P6、P11和P22)的样本均显示体细胞突变的瘤内变异等位基因频率(VAF tumor)升高,而生殖细胞突变的VAF tumor在肿瘤DNA中降低,提示CDC73Som中拷贝数增加和/或CDC73Germ中拷贝数丢失(图4a)。同样,如前所述(图2c),CDC73 LOH(P32)的患者被保留CDC73仅限于Som。因此,5例CDC73体细胞突变患者在CDC73Som中均表现为相对获益。
进一步分析发现,在转录水平上存在额外的偏向性。等位基因特异性RNA-seq分析显示,在4例CDC73双打击突变患者中,CDC73Som的基因表达比预期的(从DNA水平的等位基因频率来看)高2.3~7.9倍(图4a)。这些结果表明,CDC73Som中的等位基因增益不仅保留,而且由于等位基因特异性表达,在转录水平上进一步增强。同样,作者发现仅存在CDC73Germ突变的P75中CDC73Germ的基因表达显著低于预期,也支持在所有6例CDC73Mut患者中CDC73Som具有更高的等位基因特异性表达。
基于以上结果,作者提出了一个合理的模型来解释CDC73Som对CDC73Germ的双工偏好(基因组和转录组)(图4b)。另一个完整的等位基因(CDC73Wt)与一个失活的等位基因(CDC73Germ)一起出生,单独承担该基因的指定作用,如细胞稳态和肿瘤抑制。这导致在肿瘤发生之前更积极地使用CDC73Wt,这可以通过恶化CDC73Germ(例如,拷贝丢失或转录抑制)或积极选择CDC73Wt(例如,复制增益或转录激活)来实现。在肿瘤发生时,第二个打靶基因(体细胞截短突变)将CDC73Wt转化为CDC73Som,保持了CDC73Germ在基因组和转录组水平上的优势,并在肿瘤进展过程中获得了进一步的选择性优势。总的来说,作者的模型解释了CDC73中的等位基因不平衡和等位基因特异性表达是基于对由种系截短突变引起的CDC73单倍型不足的功能补偿,正如在其他研究中多次报道的那样。

图4 CDC73等位基因失衡及等位基因特异性表达
5、甲状旁腺癌和腺瘤的分子分型
基于RNA-seq鉴定的转录谱,作者构建了甲状旁腺癌与腺瘤和正常甲状旁腺的分子分类模型。应用最严格的过滤方法从DEGs集中筛选出597个癌症特异性强的基因,并将其用于49个样本(11例癌,28例腺瘤,10例正常甲状旁腺组织)的层次聚类。在未经任何训练或优化的初始聚类中,作者发现所有的癌症样本与非癌症样本明显分开(图5),表明基因集中存在的分子特征存在内在差异。
在2例患者中显示了分子分型的临床实用性:P27和P67。两例患者最初均被诊断为恶性潜能不确定的非典型甲状旁腺肿瘤,但均为单发;P27与癌聚集,P67与腺瘤(图5红色箭头)聚集。进一步的前瞻性随访发现P27出现临床复发,而P67未观察到病理进展的迹象,包括典型的包膜或血管侵犯。需要进一步的队列研究来验证分子分型在恶性潜能不确定的病例中的实用性,这些病例发生在0.5-5%的甲状旁腺肿瘤中。
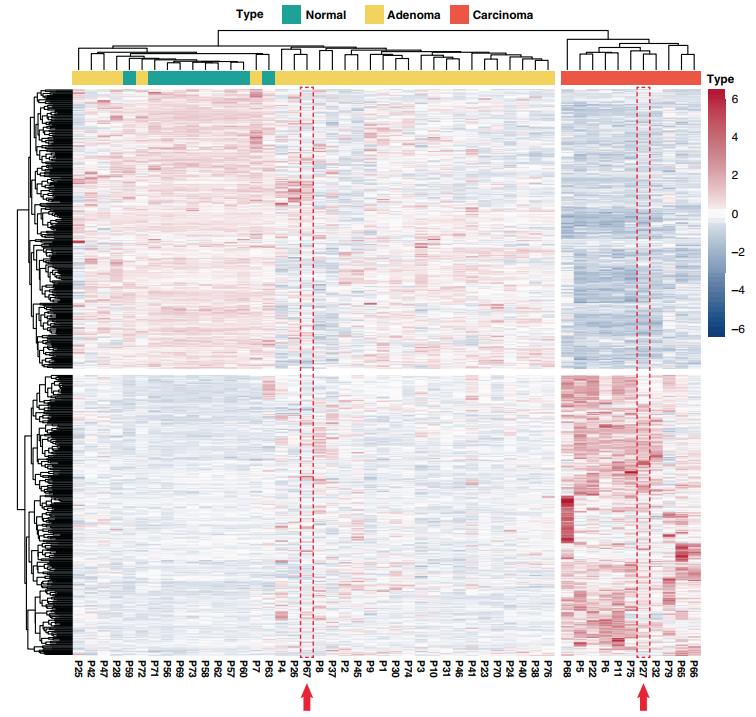
图5 甲状旁腺癌和腺瘤的分子分型
6、WT1作为CDC73突变型甲状旁腺癌的潜在标志物
利用全转录组测序数据,作者寻找可能的甲状旁腺癌单基因标志物。在肿瘤特异性表达(图4a红色圆点)的基因中,基于肾母细胞瘤1(WT1)与CDC73的功能相关性,作者重点关注肾母细胞瘤1(WT1);已知WT1直接抑制CDC73,诱导MYC和BCL-2促进细胞增殖和肿瘤发生。此外,由于WT1在肿瘤中的持续上调,WT1已被认为是多种癌症的单一分子生物标志物。
作者检验了WT1作为CDC73突变型甲状旁腺癌单基因生物标志物的可行性。作者发现WT1在肿瘤中的过表达是CDC73Mut患者特有的,而在CDC73WT患者中不存在(图6a)。此外,对38例甲状旁腺组织(28例腺瘤,4例CDC73WT癌,6例CDC73Mut癌)进行WT1的免疫组织化学(IHC)染色,证实甲状旁腺癌组织中存在CDC73Mut特异性WT1(图6b)。即无论是腺瘤(图6c)还是CDC73WT癌均未被WT1抗体染色(图6d),但5例CDC73Mut癌均显示染色阳性(图6e)。由于已知WT1的特定剪接体与某些疾病相关,作者进一步用DEXSeq检查了WT1的转录本类型,作者证实CDC73Mut组中过表达的WT1是一个典型的转录本。作者预期这些结果将为将来开发一种更快、更便宜、更准确的诊断甲状旁腺癌及其突变状态的临床测试提供依据。
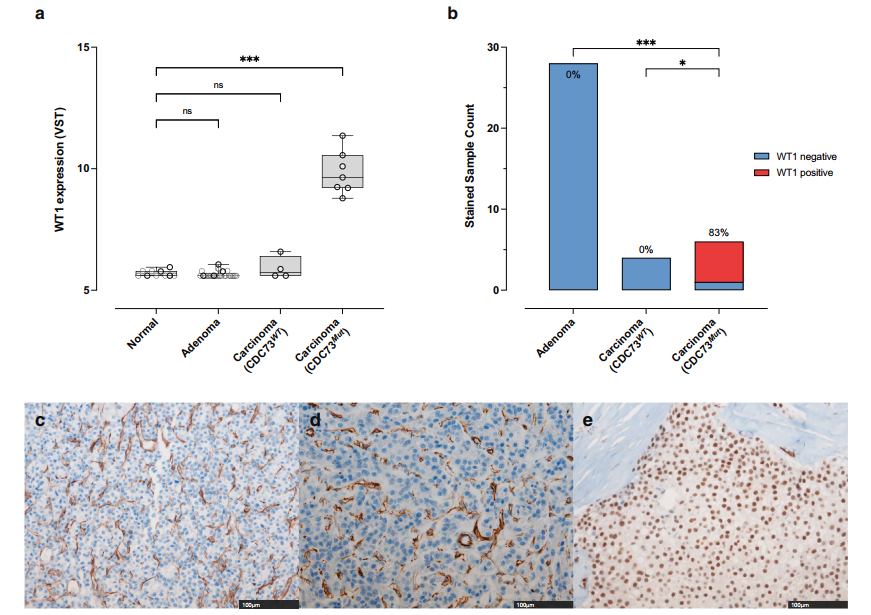
图6 WT1在CDC73Mut癌中的差异表达
结论
作者通过比较甲状旁腺癌、腺瘤和正常甲状旁腺样本的转录组学分析,确定甲状旁腺癌的特征性分子特征。作者预计,突变和转录谱、具有表型特异性表达的基因、等位基因特异性偏倚和潜在的单基因标记将为未来的研究提供新的见解,以改善诊断和个性化的治疗策略,这些已经在许多其他癌症中进行了研究。
实验方法
全外显子组测序,转录组测序,突变特征分析,等位基因特异性拷贝数分析,计算肿瘤内VAF,鉴别癌和腺瘤特异性DEGs,GSEA分析,基于DEGs的分层聚类,免疫组织化学(IHC),WT1转录物的鉴定
参考文献
Jo SY, Hong N, Lee S, Jeong JJ, Won J, Park J, Kim GJ, Kim SK, Kim S, Rhee Y. Genomic and transcriptomic profiling reveal molecular characteristics of parathyroid carcinoma. Exp Mol Med. 2023 May;55(5):886-897. doi: 10.1038/s12276-023-00968-4. Epub 2023 May 1. PMID: 37121965; PMCID: PMC10238422.






