粘蛋白-微生物组特征塑造胃癌的肿瘤微环境

胃癌(GC)是全球第五大常见癌症类型和第四大癌症相关死亡原因。由于胃癌早期缺乏症状,导致诊断延迟,预后较差。目前普遍认为胃癌是一种涉及宿主遗传易感性和环境因素的疾病,但最常见的病因是幽门螺杆菌感染。人类胃微生物群特征的研究表明,胃内生态失调是一个与癌症进展相关的动态过程,胃腺癌的特征是螺杆菌丰度降低和代表肠道共生菌属或口腔微生物组的富集。此外,胃癌组织中黏蛋白(MUC)的表达和分布也存在较大差异,其中胃和肠黏蛋白均广泛表达。根据有无黏蛋白,可分为胃、肠道、混合性或未分类/无黏蛋白表型。早期胃癌主要表现为胃黏蛋白表型,而晚期胃癌更多表现为肠黏蛋白表型。然而,在临床病理因素的背景下,胃癌中黏蛋白表达的临床重要性仍有争议,尚不清楚哪种黏蛋白表型与更好或更差的预后相关。黏蛋白是覆盖胃上皮的黏液屏障的守门人,以分泌型或跨膜糖蛋白的形式表达。此外还作为结合位点或代谢底物作为细菌的专门龛位,是细菌定点定植的重要决定因素。有研究表明,由肿瘤变化引起的异常黏蛋白改变可导致促进肿瘤进展的新微生物群的建立。因此,新类群丰度的差异可能归因于肿瘤的黏蛋白表型,但需要进一步研究。该研究发表在《Microbiome》,IF:15.5。
技术路线

主要研究结果
1. 基于黏蛋白表达的胃腺癌表型分类
在本研究中,作者首先分析了胃癌患者队列的肿瘤和癌旁组织以及功能性消化不良(FD)患者的活检组织,以检测胃(MUC1,MUC5AC,MUC6)和肠(MUC2,MUC4,MUC13)黏蛋白的mRNA相对表达量。总体而言,与肿瘤和FD组织相比,配对的癌旁非肿瘤组织中MUC1、MUC5AC和MUC6的mRNA表达显著较高(图1A)。对于肠黏蛋白,与邻近的非肿瘤组织和FD组织相比,配对肿瘤组织中的MUC13 mRNA表达显著增加,而在不同样本类型中,MUC2和MUC4 mRNA的表达未见显著变化(图1A)。在邻近非肿瘤组织中观察到的MUC2(即肠上皮化生的标志物)和MUC4(即表达痉挛性多肽的化生(SPEM)的标志物)的可变表达模式提示这些肿瘤周围部位存在化生性改变。根据黏蛋白mRNA的相对表达水平将胃腺癌细分为胃型、肠型、混合型或空黏蛋白表型组。在108个肿瘤样本中,13个(12%)在胃中,19个(17.6%)在肠中,17个(15.7%)在空白组中,47个(43.5%)在混合黏蛋白表型组中(图1B)。12个样本(11.1%)由于RNA质量不足无法确定黏蛋白表型(图1B)。随后,基于黏蛋白mRNA表达数据进行主成分分析,以验证不同表型组的肿瘤分类。值得注意的是,MUC2和MUC13 mRNA表达是具有肠黏蛋白表型的肿瘤的主要决定因素,而MUC1、MUC5AC和MUC6 mRNA表达是识别具有胃黏蛋白表型的肿瘤的最佳因素(图1C)。最后,作者还通过IHC在蛋白水平评估了黏蛋白的表达,进一步证实了mRNA表达数据(图1D)。
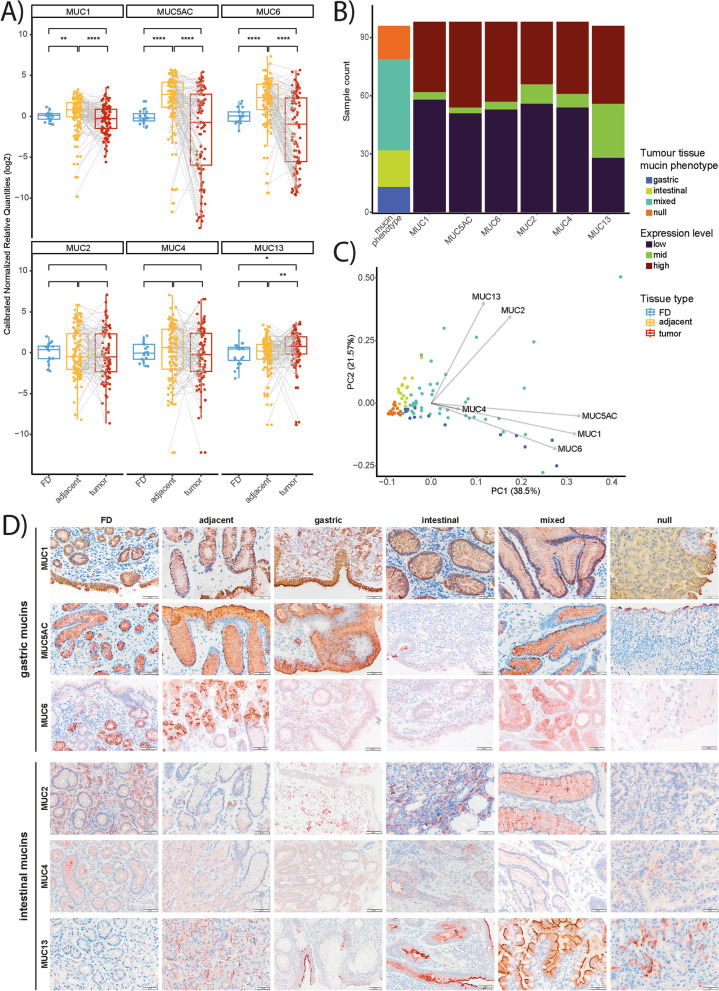
图1 与FD患者相比,GC患者胃中的异常黏蛋白特征
2. 肠黏蛋白表型和异常MUC13 mRNA表达与较差的生存率相关
作者使用Kaplan-Meier和Cox比例风险模型研究了黏蛋白mRNA表达、黏蛋白表型和5年生存率之间的关系(图2)。对于后者,临床患者数据(年龄、性别、肿瘤分期和位置)也被考虑在内。两种方法均显示肠黏蛋白表型与较差的生存率相比胃、混合和无效黏蛋白表型(log-rank检验,P=0.01;Wald检验,P=0.016,图2A)。作者还确定了个体黏蛋白表达水平与生存率之间的显著相关性。Kaplan-Meier生存曲线显示,胃癌组织中MUC5AC和MUC6 mRNA低表达与较差的生存期相关(P≤0.027;图2B)。MUC1 mRNA的表达也有类似的趋势,但差异无统计学意义(P=0.069;图2B)。相反,MUC2 mRNA中表达与较差的预后相关(P=0.038),而MUC13 mRNA低表达和高表达与较差的预后相关(P=0.068;图2B)。然而,仅Cox比例风险模型定义了MUC13高水平表达和较差的生存期之间的显著关联(P=0.016;图2B)。此外,贲门(P=0.02)和3期(P=0.001)也与较差的结局相关(图2B)。
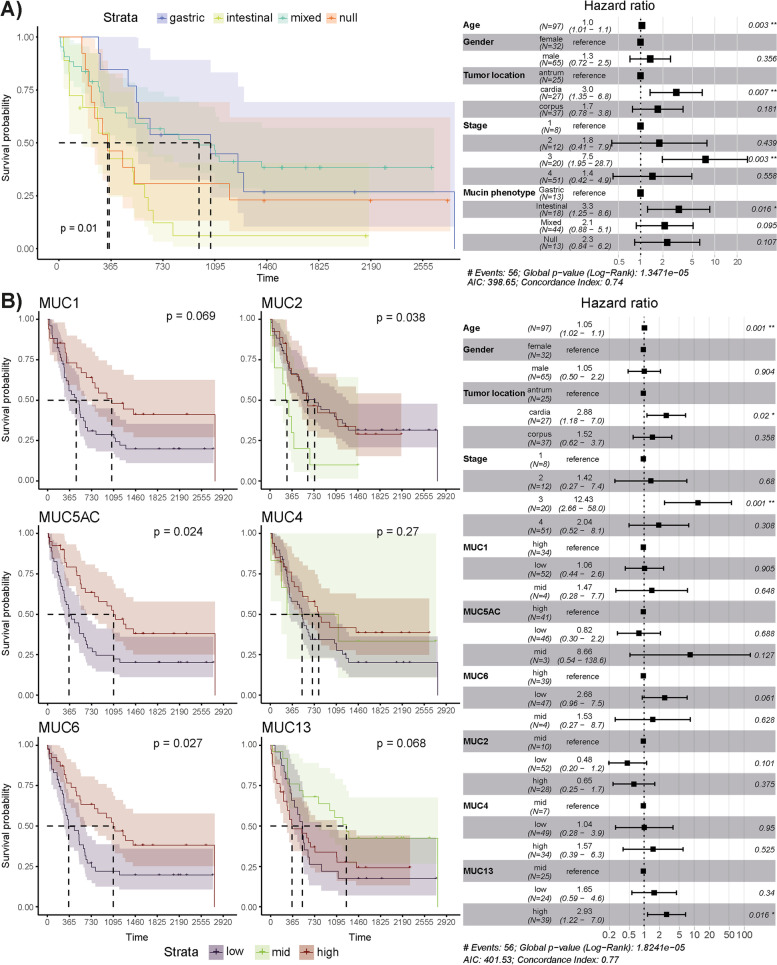
图2 肠道黏蛋白表型和异常MUC13表达与GC患者生存率降低相关
3. 不同黏蛋白表型组之间的微生物群失调发生改变
为了确定与不同GC黏蛋白表型相关的生态失调,作者首先利用每种组织类型(肿瘤、邻近非肿瘤组织和FD组织)和每种黏蛋白表型的相对细菌丰度评估了微生物组结构的变化(图3A)。总体而言,Patescibacteria的丰度在FD和癌旁组织(P=0.024)、FD和肿瘤组织(P=0.014)、FD和具有肠道(P=0.057)、混合(P=0.068)和无(0.0191)黏蛋白表型的肿瘤之间存在显著差异(图3A)。无黏蛋白表型的肿瘤与胃(P=0.0126)或肠(P=0.0295)黏蛋白表型之间的弯曲菌属丰度也存在显著差异,这可以归因于不同表型之间的螺杆菌属丰度差异。此外,在无黏蛋白表型的肿瘤与具有胃(P=0.0067)、肠(P=0.0032)或混合(P=0.0249)黏蛋白表型的肿瘤之间,拟杆菌科的丰度有显著差异(图3A)。后者在FD病例和无黏蛋白表型的肿瘤之间也有显著改变(P=0.0079;图3A)。
为了支持上述观点,作者使用α(即样本内)和β(即样本间)多样性指标进一步研究了群落组成的变化(图3B,C)。与基于调查的所有四项指标的肠道黏蛋白表型,以及基于Chao1和观察到的丰富度的混合黏蛋白表型的肿瘤样本相比,具有无黏蛋白表型的肿瘤样本显示出显著较低的α多样性(图3B)。考虑到观察到的丰富度和Shannon指数,作者还观察到与有胃黏蛋白表型的肿瘤相比,有无效黏蛋白表型的肿瘤的α多样性显著降低(图3B)。β多样性使用Bray-Curtis和加权UniFrac系统发育距离指标进行分析,并在主坐标分析(PCoA)图中可视化(图3C)。前两个轴获得了Bray-Curtis距离的24.1%和16.5%,与加权UniFrac距离的结果(24.5%和15%)相似。有趣的是,具有肠黏蛋白表型的肿瘤样本更多地分布在轴2,而具有胃表型的肿瘤仍然强烈地聚集在FD样本周围。相反,当使用ANOSIM测试群落组成的差异时,没有发现显著差异(图3C)。
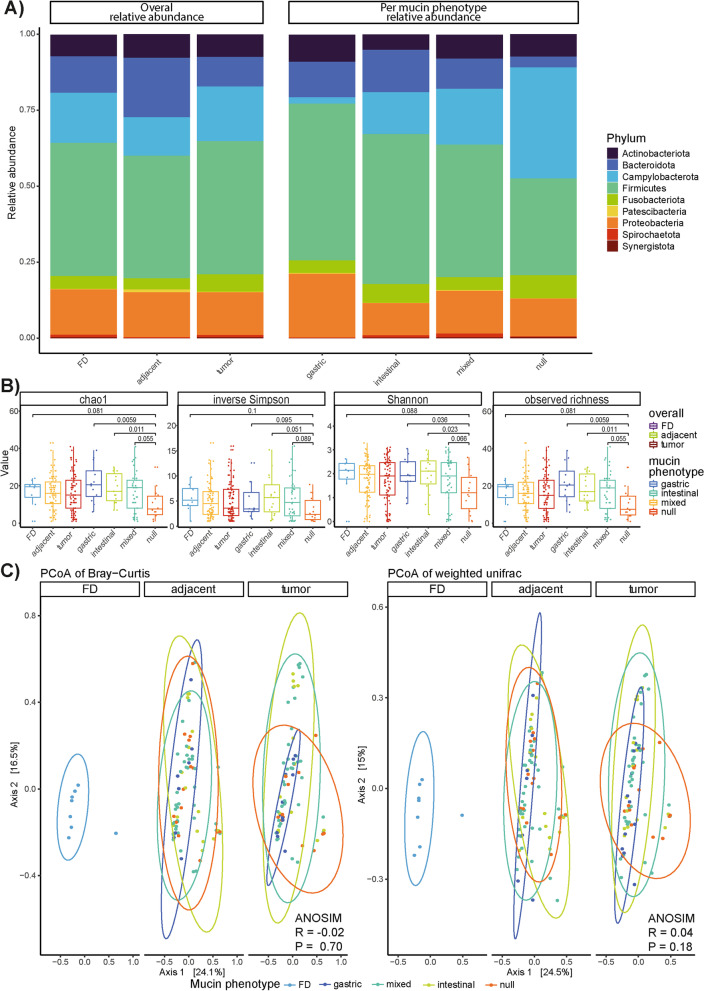
图3 FD、肿瘤和邻近非肿瘤组织之间,以及具有胃、肠、混合和无效黏蛋白表型的肿瘤之间的微生物群组成差异
为了进一步研究胃肿瘤中异常黏蛋白表达对细菌富集或缺失的影响,作者采用差异丰度分析和Spearman相关检验评估细菌属与单个黏蛋白或黏蛋白表型的相关性。与其他表型相比,螺杆菌属在MUC5AC低的肿瘤样本中富集(图4)表达。此外,Megasphaera在MUC5AC低表达的样本中富集(图4)。在MUC1 mRNA水平低的胃腺癌中,观察到大量的卟啉单胞菌(图4)。关于细菌属和肠黏蛋白表达之间的关联,作者在MUC2 mRNA水平高的胃腺癌中发现了链球菌的缺失(图4),而在MUC4 mRNA表达处于中等水平的肿瘤中,乳杆菌的数量显著较多(图4)。有趣的是,乳杆菌属、奈瑟菌属、普雷沃菌属和韦荣菌属在MUC13高表达的胃肿瘤样本中富集(图4)。

图4 与异常黏蛋白表达相关的GC富集和GC去除细菌的系统发育树
4. 不同黏蛋白表型的胃腺癌中不同的群落复杂性
采用物种共现概率模型分析GC中细菌属之间的正相关和负相关。图5显示了在不同黏蛋白表型组和MUC13低表达和高表达的胃肿瘤中鉴定出的细菌网络。在不同黏蛋白表型组之间,总体交互作用数量有显著差异,大多数交互作用见于混合黏蛋白表型和肠黏蛋白表型的肿瘤(图5B,C)。此外,不同黏蛋白表型之间的共排除交互作用与共现交互作用的比值不同,在肠型、混合型和无黏蛋白表型的肿瘤中,相对较多的共现交互作用(阴性与阳性比值分别为0.23、0.30和0.25;图5B-D),而胃黏蛋白表型患者的共排除交互作用多于共发生交互作用(1.4;图5)。此外,在属水平进行Pearsonχ2检验,以检验不同黏蛋白表型的胃癌对不同属的网络贡献的差异。残差被用作相关性的度量,仅考虑大于2的残差。在胃黏蛋白表型的腺癌中,与有另一种黏蛋白表型的肿瘤相比,毛管厌气带、Gemella和Reyranella的相互作用在比例上较多(图5A)。月形单胞菌属(Selenomonas)和密螺旋体(Treponema)对肠黏蛋白表型的样本细菌群落的影响较大,而罗氏菌属(Rothia)和普雷沃菌属(Prevotella)对无黏蛋白表型的肿瘤样本的贡献较大(图5)。在肠黏蛋白表型的胃腺癌中,大多数相互作用见于细小单胞菌属、Sediminibacterium、螺杆菌属、月形单胞菌属、梭杆菌属、雷氏菌属、密螺旋体、纤毛菌属、嗜血杆菌属、奈瑟菌属、韦荣球菌属、普雷沃菌属和链球菌属(图5B)。在混合黏蛋白表型的肿瘤样本中,相互作用最多的细菌属为别普雷沃菌属(Alloprevotella)、梭杆菌属(Fusobacterium)、Sediminibacterium、奈瑟菌属(Neisseria)、韦荣球菌属(Veillonella)、雷氏菌属(Reyranella)、罗氏菌属(Rothia)、链球菌属(Streptococcus)、嗜血杆菌属(Haemophilus)、细小单胞菌属(Parvimonas)、普雷沃菌属(Prevotella)和螺杆菌属(Helicobacter)(图5C),而罗氏菌属(Rothia)、普雷沃菌属(Prevotella)和螺杆菌在无黏蛋白表型的样本中贡献较多(图5D)。
最后,作者发现韦荣球菌属、奈瑟菌属和普雷沃菌属在MUC13高表达的肿瘤中发挥塑造群落结构的作用(图5E、F)。
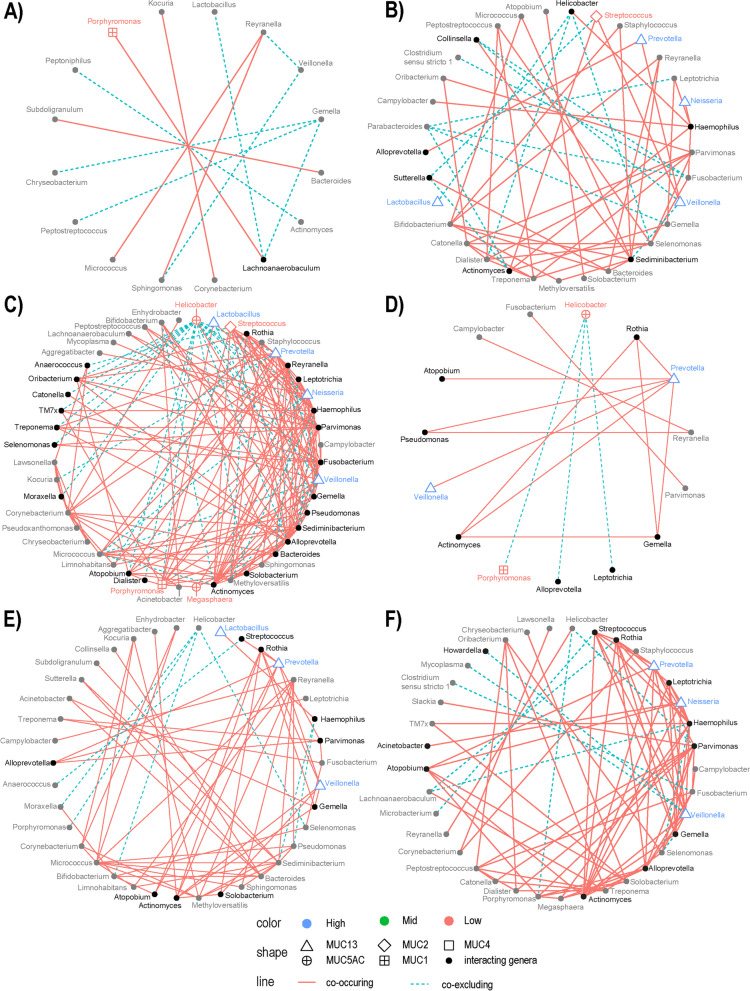
图5 与黏蛋白表型或异常MUC13表达相关的GC富集和GC缺失属的相关性
5. 胃癌中微生物组的功能能力由黏蛋白表型定义
通过PICRUSt2宏基因组推断,评估每个样本类型(FD、肿瘤和邻近非肿瘤组织)和黏蛋白表型或胃肿瘤中异常表达的黏蛋白的黏膜相关微生物组的功能。使用ALDEx2检验相对通路丰度的差异。当考虑FD、肿瘤和邻近的非肿瘤样本时,作者发现三条通路(嘌呤、鸟苷和腺苷核苷酸降解)在GC队列中存在差异丰度和富集(图6A)。此外,在被分配到不同黏蛋白表型的肿瘤之间,作者发现了5条预测的KEGG通路的差异表达,除了焦点降解通路外,大多数通路在无黏蛋白表型的肿瘤中被缺失(图6B)。
作者对每种黏蛋白重复了上述评估。对于MUC1和MUC2的mRNA表达水平,没有发现差异丰富的通路。有趣的是,在MUC6和MUC5AC低表达的样本中,嘌呤核糖核苷降解途径被耗尽。相反,在MUC13高表达的样本中,作者观察到糖降解途径的富集。此外,异乳酸发酵也富集在MUC13高表达的样本中,而丙酮酸发酵至CH2=CHCOOR I在MUC13正常表达的样本中与MUC13高表达和低表达的样本相比被耗尽。在MUC2高表达的样本中也观察到蔗糖降解IV途径的富集。胃癌微生物组代谢潜能的上述变化提示其高度依赖黏蛋白作为食物来源。
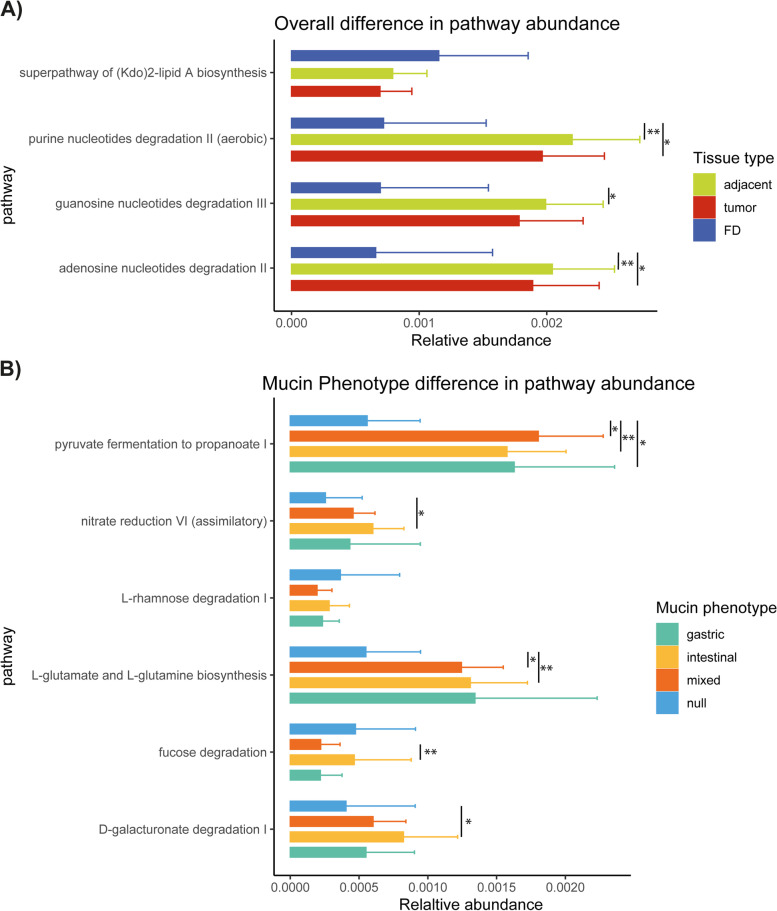
图6 宏基因组通路的相对丰度由胃腺癌中的黏蛋白表型确定
结论
综上所述,该研究确定了不同的黏蛋白-微生物组特征,这些特征塑造了胃癌的肿瘤微环境,肠道或异常的MUC13黏蛋白环境与不良结局相关。作者还发现,奈瑟菌属、普雷沃菌属和韦荣球菌属等口腔致病性菌属成员是胃癌中MUC13介导的信号传导的潜在驱动因子,这些信号传导可能是预测疾病结局的有用生物标志物。由于并非所有的胃癌前疾病最终都会演变为癌症,因此癌前状态患者的这种口腔分类群-MUC13特征也可能有助于预测进一步演变为癌症的可能性。此外,具有肠黏蛋白表型的腺癌有利于促炎口腔细菌的建立,形成强大的共现网络。最终,理解这些在胃癌发生中的黏蛋白-微生物组特征可能会影响胃癌的预防和治疗策略,因此建议在其他胃癌队列中进行充分的独立外部验证。
实验方法
RT-qPCR,免疫组化,生存分析,16S rRNA基因测序,差异丰度分析,微生物网络分析
参考文献
Oosterlinck B, Ceuleers H, Arras W, De Man JG, Geboes K, De Schepper H, et al. Mucin-microbiome signatures shape the tumor microenvironment in gastric cancer. Microbiome. 2023 Apr 21;11(1):86.






