OrganoidDB:类器官转录组及单细胞测序数据库
类器官是来源于多能干细胞(胚胎干细胞或诱导干细胞)或成体干细胞,甚至是患者特异性组织样本的三维体外组织培养物。类器官可以被引导分化成具有器官特征的各种细胞类型,并且具有与真实器官相似的组织学组织,有时与真实器官的组织学组织相似,有时无法区分。此外,它们的形成概括了器官生长的自组织过程。因此,类器官是比二维细胞系更有前途的模型,用于人体过程和结构的体外研究、疾病发作和临床前药物开发。此外,与动物模型相比,类器官可以以更高的效率和速度生成,并提供更准确的人体组织表示。
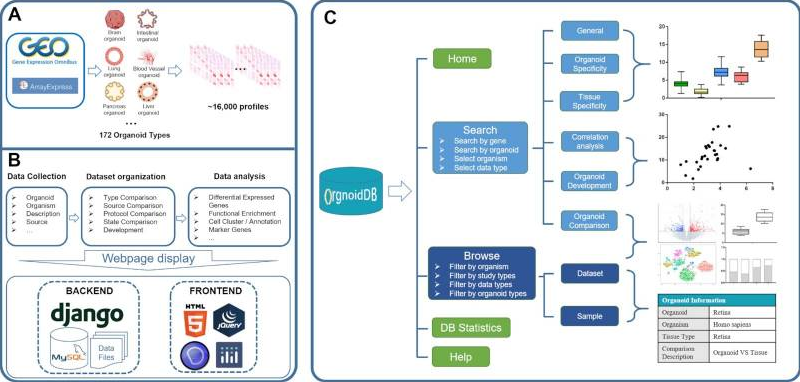
为了充分发挥类器官技术在疾病研究和药物开发领域的潜力,亟待解决的关键问题是验证类器官能否忠实地反映人体的生物过程,这涉及细胞组成、分化和状态以及对外界刺激反应的标准定义并描述这些模式。此外,开发一种能够更准确地模拟体内环境的可靠类器官培养方案也将成为未来的研究方向。因此,使用高通量测序技术(尤其是单细胞测序)将类器官与相应的人体组织进行比较,将为评估类器官的质量提供一种有效的方法。正在进行的类器官细胞图谱项目于 2021 年提出,致力于探索类器官的单细胞表征。此外,用于类器官研究的转录组学数据也越来越多。然而,目前还没有公开的类器官相关数据库或来自公共类器官研究的转录组图谱的集中数据门户。
今天我们介绍OrganoidDB 数据库(http://www.inbirg.com/organoid_db/),这是一种用于类器官的综合转录组数据(芯片、RNA-seq和单细胞 RNA-seq)资源。
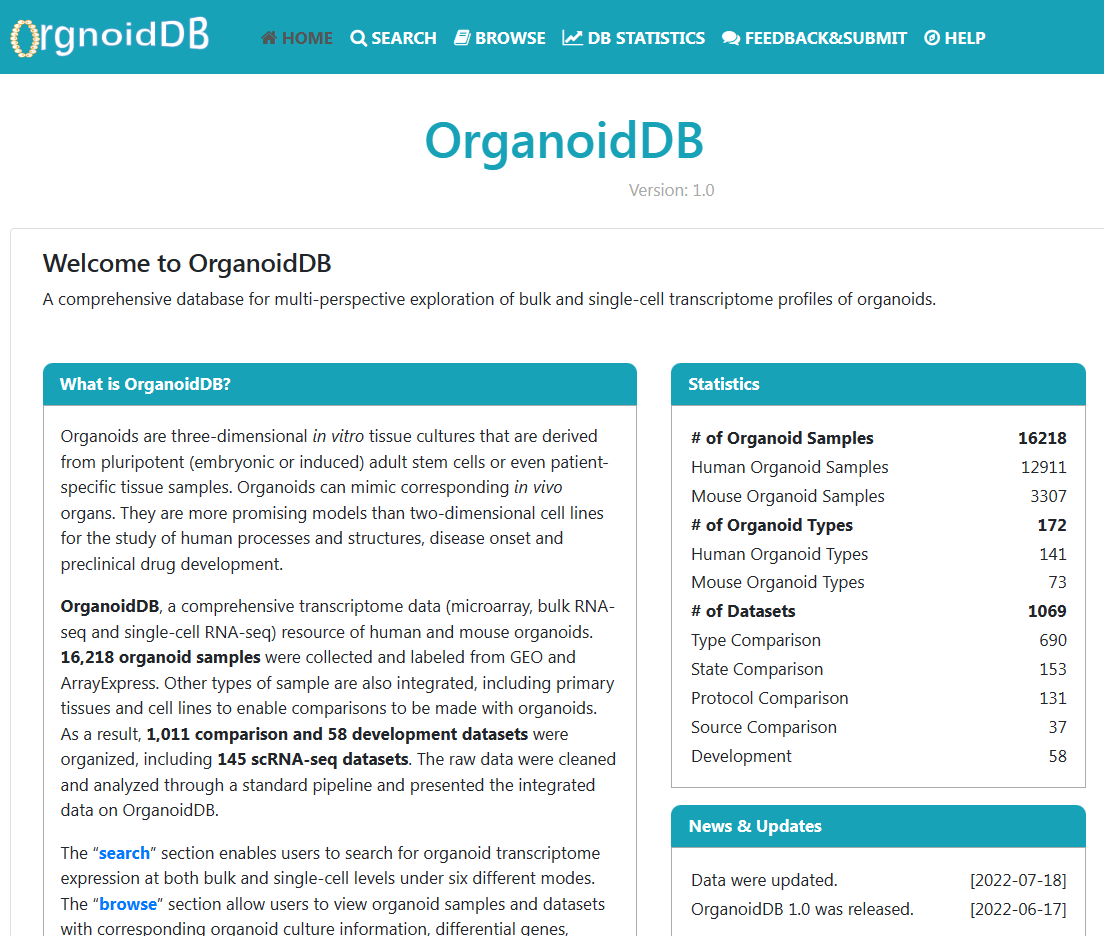
目前,OrganoidDB提供了16218个类器官样本的转录组数据,包括12911个人类器官样本和3307个小鼠类器官样本。这些样本涵盖了172种不同的类器官类型,包括肠道类器官、脑类器官、肺类器官等。这些样本被组织成 1069 个数据集,包括145个单细胞 RNA-seq 数据集,分为五大类——“类型比较”、“状态比较”、“协议比较”、“来源比较”和“类器官开发”。这五个类别对生物学背景的描述如下:“类型比较”将类器官样本与其他样本类型(如组织、原代细胞和异种移植物)进行比较;“状态比较”比较不同状态的类器官,例如疾病与正常状态、增殖与静止状态、成人与胎儿状态;“方案比较”比较了在不同生长条件下生长的类器官,例如分化培养基与扩增培养基、共培养物与单一培养基、基质胶与胶原蛋白,“来源比较”比较了从不同来源生长的类器官,例如成体干细胞 (ASC) 与多能干细胞 (PSC)、诱导多能干细胞 (iPSC) 与胚胎干细胞 (ESC) 以及患者来源的类器官 (PDO) 与患者来源的异种移植类器官 (PDXO)。 最后,“类器官发育”计算每个基因在基因表达值和类器官发育时间点之间的相关性。
在搜索页面,OrganoidDB允许用户在人或小鼠的体积和单细胞RNA水平下,以六种模式对类器官和其他样本类型的基因表达进行多视角探索(图(图2A)。2安培).总则。允许用户搜索不同类器官与原代组织、细胞系培养物、PSC 培养物和球状体相比的基因表达概览。
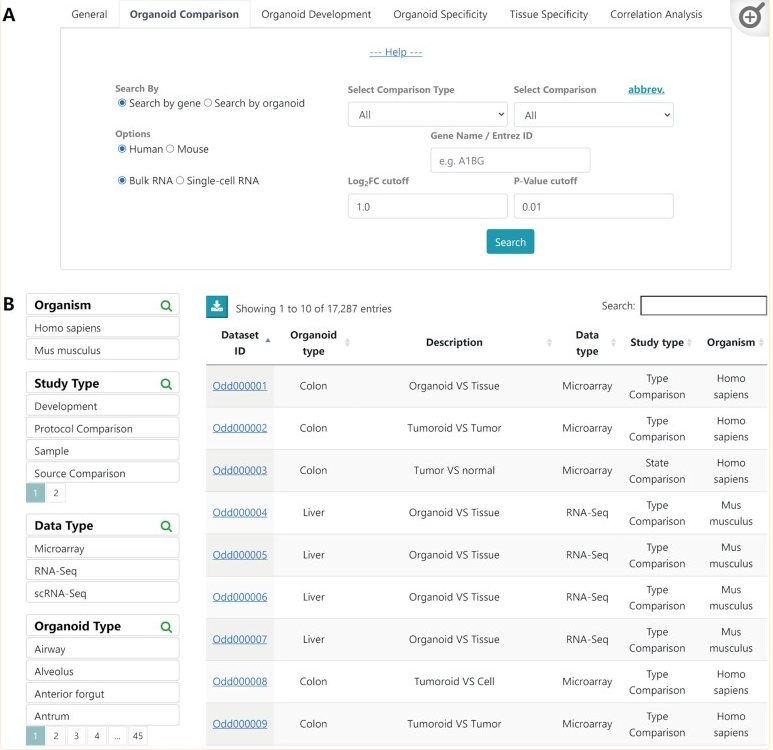
详细信息页面,包含样本和数据集的可用类器官培养信息。提供了样本特征,例如样本捕获时间和派生样本的患者人口统计信息。对于批量RNA数据集,提供了整体分析结果,包括DEGs、富集功能/通路和差异细胞类型标志物。对于单细胞RNA-seq数据集,除了整体分析外,单细胞聚类分析还将生成细胞聚类图,其中包含细胞类型注释(如果可用)、聚类标记基因和聚类中的DEGs。其他交互式图,包括箱形图、火山图和热图,也用于详细信息页面中的数据可视化。







