METTL1介导的tRNA m7G修饰通过tRNA调控的翻译控制促进AML的白血病生成
RNA修饰已被证实在调节细胞生物学过程中发挥着重要作用。最近,N7-甲基鸟苷(m7G)修饰及其修饰因子METTL1/WDR4已被证实在多种癌症中具有致癌基作用。本文旨在确定METTL1/WDR4在急性髓系白血病(AML)中的功能和分子机制,更好地了解AML的白血病发生机制,为AML治疗提供了有希望的候选靶点,提高AML的临床治愈率。通过qRT-PCR、急性髓细胞性白血病临床样本的免疫印迹分析和公开急性髓细胞性白血病数据集的生物信息学分析,对METTL1/WDR4的表达水平进行量化。采用CCK-8检测和细胞计数检测确定细胞增殖情况。使用流式细胞仪检测评估细胞周期和凋亡率。体外试验机制研究采用了多种技术,如Northern印迹分析、液相色谱-串联质谱(LC-MS/MS)、tRNA稳定性分析、转录组测序、小非编码RNA测序、定量蛋白质组学和蛋白质合成测量。结果表明METTL1和WDR4在AML患者中显著上调,其表达水平与AML患者预后不良密切相关。且敲除METTL1可有效抑制AML细胞增殖,促进细胞凋亡,并在G1期阻止细胞周期,从而抑制体外AML细胞进展。METTL1可通过特异性介导tRNA上m7G修饰状态,调节细胞内携带m7G修饰tRNA的稳定性和生物发生,影响胞内涉及翻译调节过程和酶活性等相关蛋白翻译效率。该研究证实了METTL1介导的m7G-tRNA修饰在AML进展中的重要作用,为未来有效AML治疗策略的开发提供了理论基础。本文于2024年1月发表在《Experimental Hematology & Oncology》,IF:10.9。
技术路线
主要实验结果:
1. METTL1和WDR4在AML中上调,与预后不良相关
为了评估METTL1/WDR4在AML中的表达,从NCBI GEO数据库中收集了涉及健康个体和新诊断AML患者的公开数据集。骨髓细胞转录组分析显示,与健康个体相比,AML患者METTL1和WDR4的表达水平显著升高(图1A)。新桥医院对新诊断的AML患者和健康供体的公开数据集和qRT-PCR验证的进一步比较分析表明,METTL1和WDR4在骨髓和外周血样本中均显著上调(图1B、C)。在亚型分析中,METTL1和WDR4在几乎所有FAB亚型中都表现出上调,尤其是在M1、M2、M4、M5和M6亚型中(图1D、E)的AML。此外,根据第五次WHO分类标准重新分析了具有易位和突变亚型的AML患者中METTL1/WDR4复合物的丰度。在具有完整临床数据的样本中,与健康供体相比,大多数WHO亚型中METTL1和WDR4的表达上调。此外还分析了公共数据集以证实在患者中的观察结果,结果与实验数据相似。Western blot分析也证实,METTL1和WDR4在AML患者的骨髓单核细胞中表达更高(图1F)。此外,使用来自癌症基因组图谱(TCGA)的急性髓系白血病(LAML)数据集发现METTL1或WDR4表达水平显著较高的AML患者生存率较差(图1G),这表明METTL1和WDR4在调节AML进展中具有潜在功能。
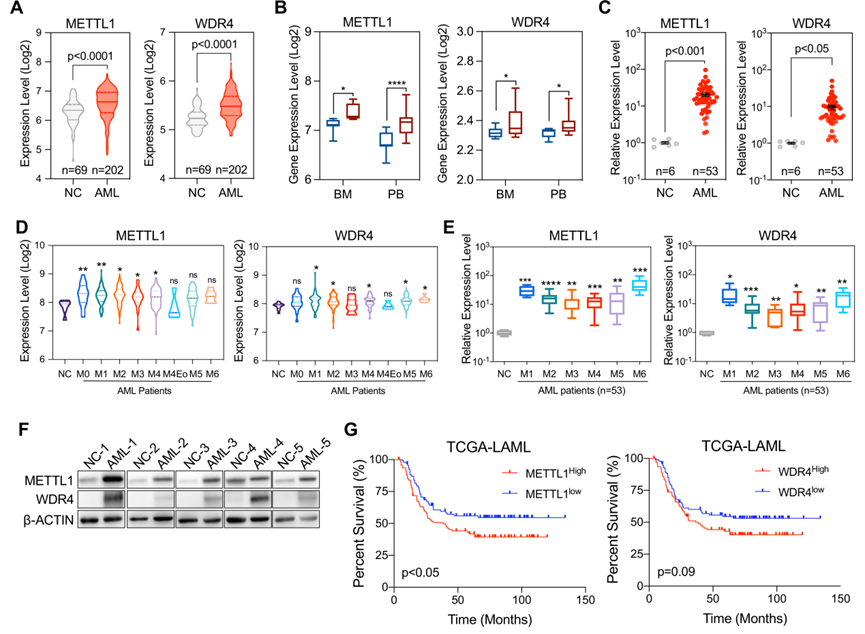
图一:METTL1/WDR4上调与AML患者预后不良有关
2.METTL1敲除可抑制人AML细胞的存活和生长
为了研究METTL1在AML中的作用,我们使用shRNA系统来敲除METTL1的表达水平,其中包括一个阴性对照(shNC)和两个独立的shMETTL1(shMETTL1-1:shM1-1和shMETTL1-2:shM1-2),它们在THP-1和MOLM-13 AML细胞系中显示出有效的METTL1蛋白水平敲除(图2A)。结果显示敲除METTL1抑制了上述AML细胞的细胞生长速率(图2B、C)。细胞周期分析显示,在AML细胞中敲除METTL1后,G1期细胞显著增加,S期细胞减少(图2D、E),表明敲除METTL1诱导细胞周期停滞在G1期。此外,膜联蛋白V/7-AAD染色显示,敲除METTL1诱导AML细胞凋亡(图2F-H)。蛋白质印迹分析显示,METTL1敲除导致BCL2下调,促进Caspase3的裂解,从而提高了AML细胞的凋亡率(图2I)。相反,METTL1在HL60AML细胞中的过表达促进了细胞增殖(图2J,K)。当接受阿糖胞苷(CYT)治疗时,METTL1敲除的AML细胞表现出显著降低的增殖能力和对阿糖胞苷诱导的细胞死亡的易感性增加(图2L、M)。相反,用阿糖胞苷处理的过表达HL60细胞的METTL1表现出显著加速的生长速率和减少的细胞凋亡(图2N-P)。
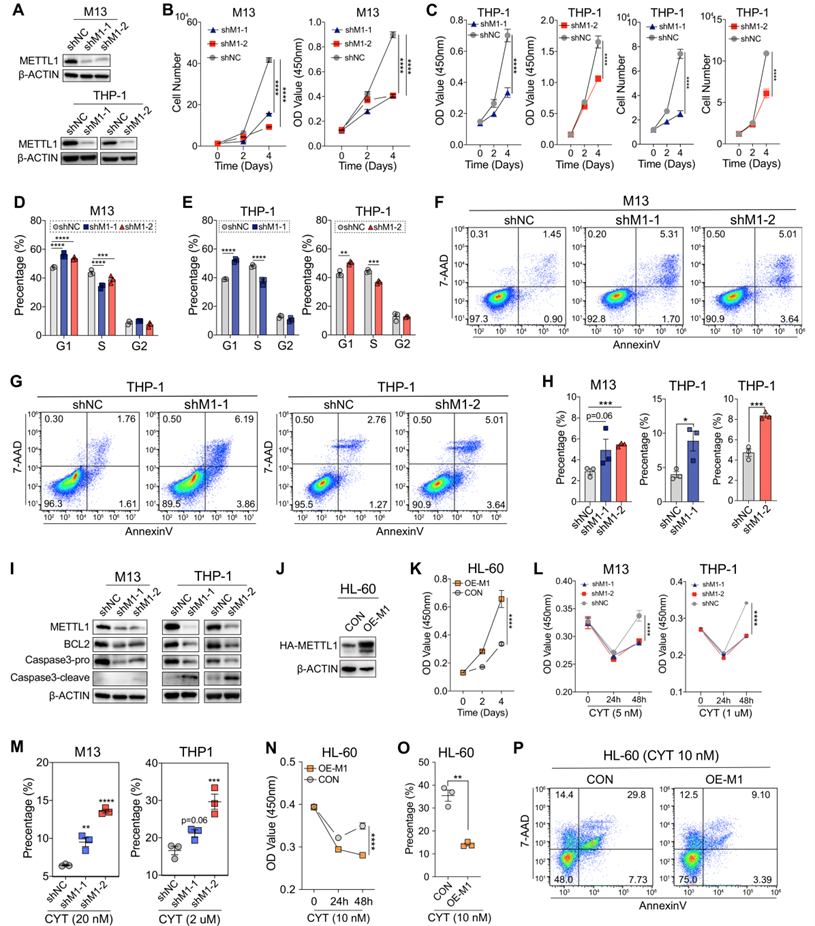
图二:METTL1敲除抑制人AML细胞的存活和生长。
3.AML中METTL1的表达受DNA甲基化的调控
之前的数据表明,METTL1的表达可能受到DNA甲基化的调控。为了研究METTL1在AML细胞中保持高表达状态的原因,从数据库DiseaseMeth和TCGA中收集AML的DNA甲基化阵列数据(450k)发现AML患者中METTL1的DNA甲基化水平(chr12:58165414–58167914)低于健康对照组(DiseaseMeth数据,图3A)。根据美国国家生物技术信息中心(NCBI)的基因信息,METTL1位于12号染色体的第一外显子58162254-58165888(-)位点上,该DNA甲基化区域大约位于距离转录起始位点(TSS)2千碱基(kb)以内,因此可以归类为基因启动子区域。此外,METTL1的DNA甲基化水平与METTL1 mRNA表达呈负相关(TCGA数据,图3B)。通过应用靶向整体DNA去甲基化水平的基于胞嘧啶的TET酶抑制剂(bobcat339盐酸)处理AML细胞,发现使用bobcat339盐酸处理24h和48h后,AML细胞系中METTL1的转录水平显着降低,同时METTL1蛋白下调(图3C、D),表明Tet家族调控的METTL1基因启动子区域的DNA去甲基化可能在调控AML细胞中METTL1激活中发挥主要作用。
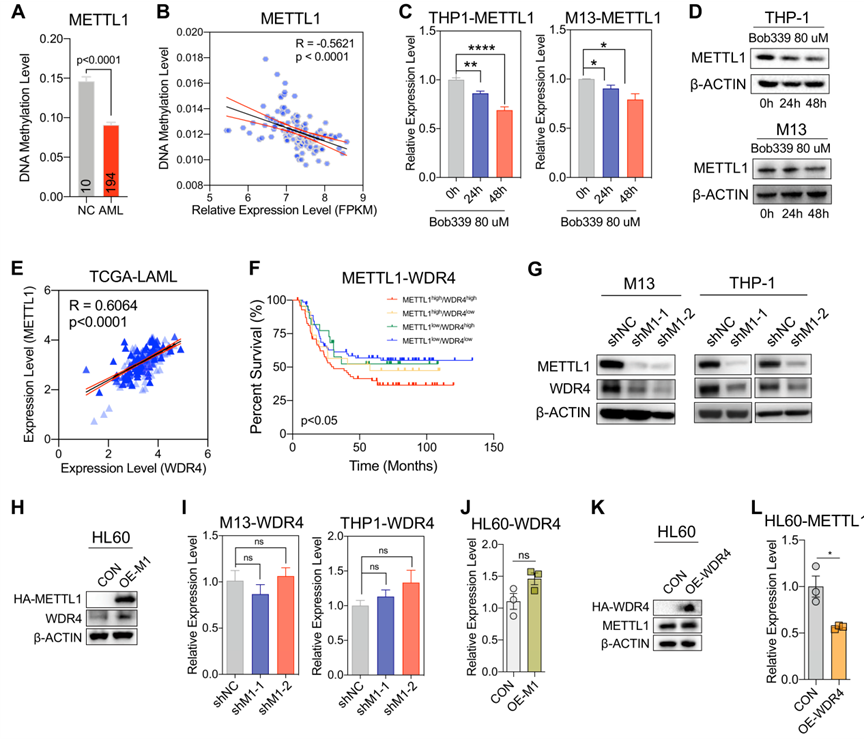
图三:DNA甲基化调节AML细胞中METTL1的表达以及METTL1和WDR4的相关性。
4.METTL1和WDR4在AML细胞中的协同表达由METTL1引导
之前的研究表明,METTL1和WDR4共同作用以增加RNA中的m7G修饰,并在包含AML在内的各种癌症中显示出协同表达模式。为了进一步研究AML中METTL1和WDR4协同表达的调控机制,首先分析了癌症基因组图谱(TCGA)中的急性髓系白血病(LAML)转录组数据集,并确认METTL1的转录水平与WDR4呈正相关(r=0.6064,p<0.0001,图3E)。此外,同时表达高METTL1和WDR4的AML患者预后最差(图3F),表明METTL1和WDR4之间存在正协同表达模式。进一步分析表明,WDR4的蛋白水平在METTL1敲除后显著降低,并随着METTL1过表达而增加(图3G,H)。然而,WDR4的mRNA表达水平在AML细胞中METTL1敲除或过表达后没有明显变化(图3I,J),表明WDR4通过METTL1的调控可能发生在转录后水平。值得注意的是,在AML细胞中,WDR4表达的改变对METTL1的表达水平无明显影响,无论是在mRNA水平还是在蛋白水平(图3K,L)。这些结果表明,METTL1和WDR4在AML细胞中的协同表达是由METTL1通过转录后调控机制引导的。
5.敲除METTL1会导致细胞中tRNA和mRNA上的m7G修饰丰度降低
为了证实METTL1是否能决定AML细胞中tRNA和mRNA上m7G修饰的丰度,应用基于LC-MS/MS的高通量RNA修饰定量检测平台检测METTL1敲除AML细胞中m7G RNA修饰的状态。首先,从有METTL1或没有METTL1的AML细胞中分离出富含tRNA的片段。通过LC-MS/MS平台,对tRNA和mRNA上的18种RNA修饰进行量化(图4A).与对照组相比,METTL1基因敲除的AML细胞中tRNA上的m7G水平明显下降,而其他类型的RNA修饰的丰度几乎不受影响(图4B-D),表明METTL1会特异性地影响AML细胞中tRNA上的m7G丰度。最近的研究表明,哺乳动物细胞中的mRNA也含有相当丰富的内部m7G修饰,因此我们接下来要检测富含poly(A)的mRNA上的m7G水平。由于mRNA仅占总RNA的一小部分(约3–7%),而mRNA通常在每个分子的5'末端都含有三个m7G帽,因而进行了两轮poly(A)富集实验以去除其他RNA的污染,并进行了两次去帽实验以在很大程度上去除mRNA 5'末端的m7G帽,从而聚焦于mRNA的丰度变化。结果表明,在METTL1敲除的AML细胞中,poly(A)富集的mRNA上的m7G水平明显下降,,而m2G,m1G,m22G,m227G不受影响(图4E-G)。有趣的是,与tRNA上的RNA修饰丰度不同,在METTL1敲除后,在METTL1敲除的AML细胞中,poly(A)富集的mRNA上的m7G水平明显下降,表明在METTL1敲除的AML细胞中,poly(A)富集的mRNA上的m7G水平明显下降。
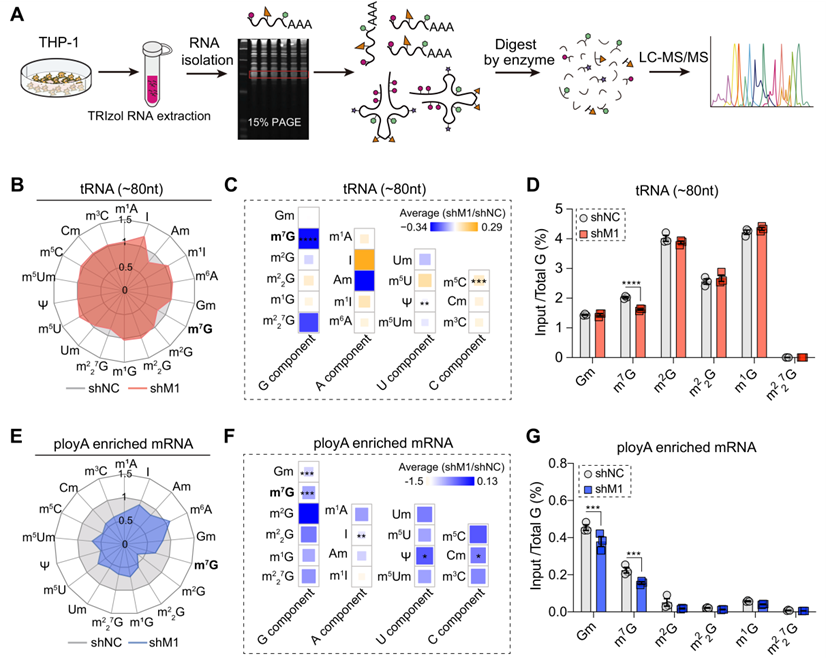
图四:敲除METTL1会导致THP-1细胞中tRNA和mRNA上的细胞m7G修饰丰度降低
6.METTL1敲除通过tRNA表观转录组失调减少AML细胞中新生蛋白质合成
最近的研究表明,缺失METTL1可降低哺乳动物细胞中m7G修饰转录本的翻译效率,并强调mRNA内部m7G是一种新型表转录组标记,在翻译中具有调控作用。此外,研究还表明,METTL1/WDR4介导的tRNA甲基组上的m7G也是mESC中正常mRNA翻译所必需的,并调控ESC的自我更新和分化。由于数据显示METTL1的敲除可降低AML细胞中富含poly(A)的mRNA和总tRNA上的m7G水平,接下来要研究的是METTL1敲除引起的AML细胞中m7G丰度的降低是否会影响细胞翻译效率。由此进行了OPP标记实验,以检测AML细胞中新生蛋白质的整体生物发生情况。结果显示,通过siRNA敲除METTL1确实降低了AML细胞中OPP平均荧光强度(MFI)的水平(图5A,B),表明在METTL1敲除的AML细胞中,新生蛋白合成和全局mRNA翻译受到了抑制。然而,转录组测序显示METTL1敲除的AML细胞中几乎没有基因发生改变(图5C),这一结果表明,在METTL1敲除的AML细胞中观察到的新生蛋白合成抑制可能并不是主要受METTL1敲除的AML细胞中的基因改变影响(图5D)。此外,定量蛋白质组学实验发现,METTL1敲除诱导下调的蛋白富集于蛋白翻译过程和一系列酶活性(图5D、E),这与上述结果一致,表明METTL1调控AML细胞的全局翻译。
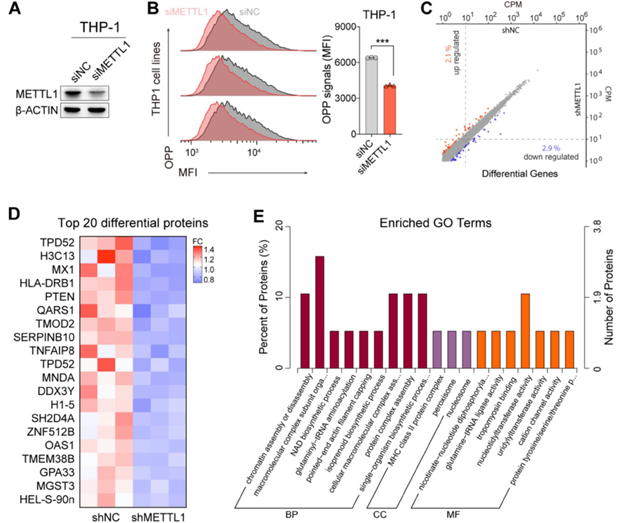
图五:敲除METTL1可抑制THP-1细胞中新生蛋白质的合成
为了进一步证实METTL1敲除AML细胞的翻译抑制是否是由m7G失调的tRNA甲基组诱导的,首先对AML细胞中一些选定的tRNA进行Northern印迹检测,数据显示与METTL1对照AML细胞相比,METTL1敲除AML细胞中tRNAAla、tRNAVal和tRNALeu的水平显著下降(图6A-D)。表明METTL1介导的tRNA m7G缺失可能会影响AML细胞中的细胞tRNA谱。进一步的OPP标记实验显示,与METTL1对照细胞相比,转染了从METTL1敲除细胞中分离的tRNA的AML细胞的OPP平均荧光强度(MFI)水平降低(图6E-G),表明m7G失调的tRNA甲基化组也影响了细胞tRNA库的翻译功能。综上所述,上述结果表明,METTL1介导的tRNA m7G修饰可调控细胞tRNA库和tRNA表转录组,从而进一步导致AML细胞全局mRNA翻译失调。
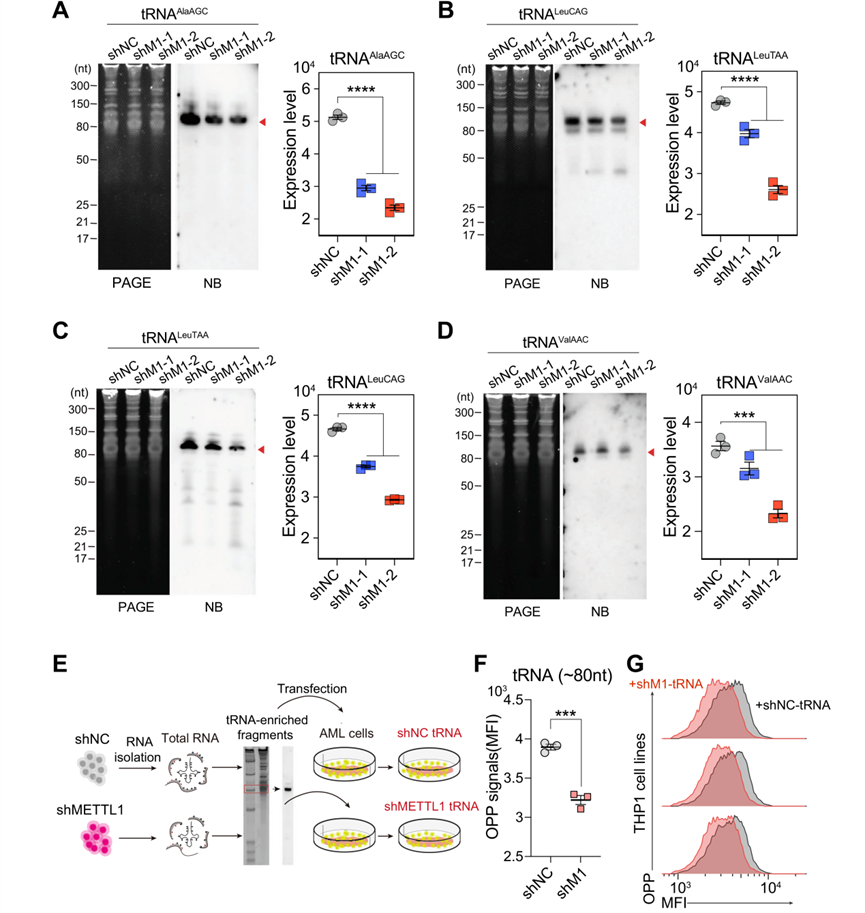 图
图
六:敲除METTL1会导致tRNA失调,从而抑制新生蛋白质的合成
7.METTL1促进AML细胞中tRNA衍生小RNA的生物发生
为了研究METTL1基因的敲除如何影响tRNA谱,用RNase A/T1处理从METTL1敲除的AML细胞和METTL1对照AML细胞中分离的总tRNA。结果表明,METTL1的敲除增加了tRNA对Rnase A/T1降解的敏感性(图7A),从而解释了在AML细胞中观察到的由METTL1敲除引起的tRNA改变。作为一种新型的调节性小非编码RNA,tRNA衍生的小RNA—tsRNA在各种基本生物学条件下以及调节多种癌症的肿瘤发生中发挥重要作用。tsRNA的生物发生对各种细胞应激敏感,由其前体tRNA的二级结构和甲基化组决定,可将切割位点暴露于多个Rnase,促进tsRNA的生物发生。在这项研究中发现METTL1的敲除降低了总tRNA中的m7G水平,促进了Rnase A/T1消化下的tRNA降解,这可能会促进tsRNA在AML细胞中的生物发生。为了探究敲除METTL1是否能提高AML细胞中tsRNA的整体水平进行了METTL1敲除和METTL1对照AML细胞之间的高通量小非编码RNA测序实验,结果表明,在METTL1敲除AML细胞中,包括5'tsRNA、内在tsRNA和3'CCA-tsRNA在内的一些tsRNA的水平显著升高(图7B),通过Northern印迹和RT-PCR进一步证实了这一点(图7C-E)。相反,高通量小非编码RNA测序和RT-PCR实验表明,METTL1过表达的AML细胞中大多数tsRNA相应减少(图7F、G)。然而,当将来自METTL1敲除细胞的分离的tsRNA片段转染到AML细胞中时,AML细胞中的整体翻译效率并未受到影响,这表明tsRNA的分子调控机制可能不像AML细胞中的tRNA那样参与细胞新生蛋白质合成,应进行更多研究以揭示tsRNA在AML白血病发生中的作用机制。总之,上述结果表明,METTL1介导的tRNA上的m7G修饰在调控AML细胞中tRNA稳定性和tsRNA生物发生中起着重要作用,可能会进一步影响AML的细胞活力和白血病发生。
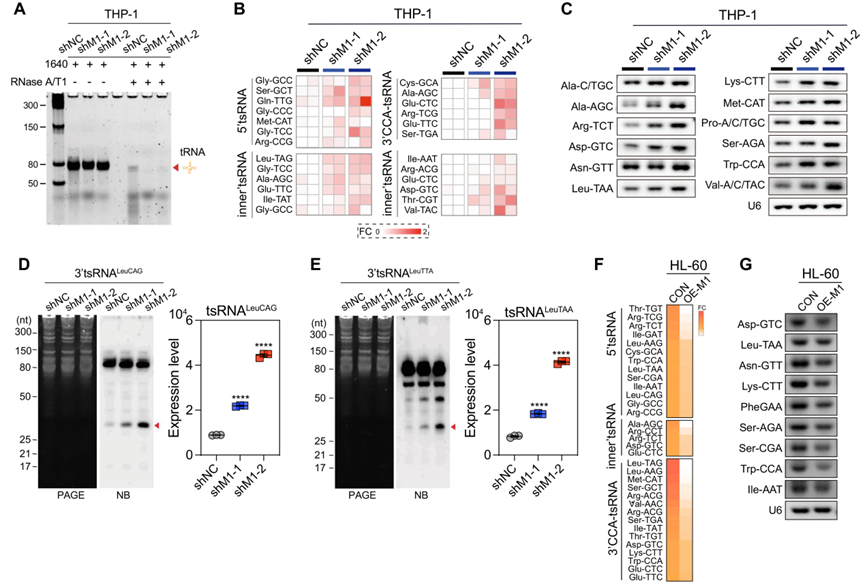
图七:METTL1敲除促进tsRNA生物发生。
实验方法:
qRT-PCR,WB,生物信息学分析,CCK-8法和细胞计数法,northern印迹法、液相色谱-串联质谱(LC-MS /MS)、tRNA稳定性分析、转录组测序、小非编码RNA测序、定量蛋白质组学和蛋白质合成测定。
参考文献:
Zhao P, Xia L, Chen D, Xu W, Guo H, Xu Y, Yan B, Wu X, Li Y, Zhang Y, Zhang X. METTL1 mediated tRNA m7G modification promotes leukaemogenesis of AML via tRNA regulated translational control. Exp Hematol Oncol. 2024 Jan 24;13(1):8. doi: 10.1186/s40164-024-00477-8. PMID: 38268051; PMCID: PMC10807064.






