与原发性癌细胞组成相似但免疫细胞表型不同的远处转移乳腺癌细胞
乳腺癌是女性最常诊断的癌症,远处转移是乳腺癌相关死亡的主要原因。阐明与转移性疾病相关的肿瘤和免疫生态系统的变化对于提高对转移的理解和最终治疗至关重要。在这里,我们开发了一个深入的、空间分辨率的单细胞图谱,研究了原始人类乳腺肿瘤和匹配的远处转移瘤中肿瘤和免疫细胞的表型多样性,使用成像质量细胞计数术来分析总共75个独特的抗体靶点。尽管相同的肿瘤细胞表型通常存在于原始肿瘤和转移部位,这表明原始肿瘤具有强大的创始人效应,但它们的比例在匹配样本之间有所不同。值得注意的是,除了大脑之外,转移部位不影响肿瘤表型组成。转移部位总体上表现出较低的免疫细胞数量,但骨髓细胞以及耗尽的细胞毒性T细胞比例较高。骨髓细胞表现出不同的组织特异性组成特征,并且转移部位潜在的矩阵重塑表型的存在增加。这项对转移性乳腺癌肿瘤和免疫细胞表型组成的分析强调了该疾病在患者体内和远处转移部位的不均匀性,表明骨髓细胞是可能针对这些部位的主要免疫调节剂。匹配的原始和转移性乳腺肿瘤的多重成像分析提供了肿瘤微环境的表型和空间地图,揭示了匹配样本之间相似的癌细胞组成和不同的免疫学特征。本文于2025年1月发表于《Cancer Research》,IF:13.3。
研究技术路线:
研究结果:
1. 原始乳腺肿瘤和匹配转移瘤的高度多重单细胞图谱
为了确定远处转移的独特特征,我们在87名乳腺癌患者队列中比较了FFPE 原发性肿瘤和匹配的骨、脑、肝脏和软组织转移(图1A)。该队列之前对B细胞和T细胞渗透以及LAG 3和PD-1的表达进行了特征。原发性肿瘤之前被归类为腔室A(激素受体阳性、HER 2阴性和低Ki 67)、腔室B(激素受体阳性、HER 2可变和高Ki 67)、HER 2富集(HER 2阳性和激素受体阴性)和TN(激素受体阴性和HER 2阴性)亚型。
我们使用高度多重的MHC来描述这些样本中的肿瘤和免疫细胞表型。我们用三个抗体组对每个样本的三个连续切片进行了染色,总共针对75种蛋白质和蛋白质磷酸化位点,重点关注肿瘤细胞、T细胞或骨髓细胞(图1B)。我们的肿瘤细胞小组包括关键临床标志物(ER、HER 2和Ki 67)、生长相关信号途径标志物(p-mTOR、pRB和pS 6)、与免疫相互作用相关的标志物(HLA-ABC和PD-L1)和关键谱系标志物(CK 5、CK 14和GATA 3)。我们的两个免疫小组包括能够对CD 8+和CD 4 + T细胞亚群进行表型特征的标记物,包括T细胞的调节状态(FOXP 3)和耗尽状态(例如,LAG 3、PD-1和Tim 3),并推断骨髓细胞的功能状态(例如,MP 9、STING和CD 38)。许多测量的标志物已经可以作为治疗靶向,或者药物很快就会上市。
根据指导方针,基于苏木素和伊红染色,病理学家将原发性肿瘤切片注释为肿瘤内、肿瘤内富含间质和侵袭性边缘区域。该数据集平均包括来自原发性肿瘤的六张图像和来自转移部位的两张图像,每个患者、每个小组。我们总共记录了2,043张图像(每个面板681张图像),并检测到每个面板超过100万个单细胞。我们使用数据处理和单细胞分割管道分析了这些图像,比较了由于单个图像和部位之间绝对细胞数量的变化而导致的细胞类型的比例(而不是绝对)数量。为了确保防止组织特异性染色伪影,我们评估了降维图上图像和单细胞的聚集,并检查了预期的染色模式。我们强调对骨样本进行QC的需要,这对于使用MHC的制备和成像来说尤其具有挑战性。
我们使用无监督的基于图形的集群来识别肿瘤、免疫或间质细胞,然后根据平均标记物表达进行集群注释。肿瘤细胞是组织中最常见的细胞类型(>50%的细胞),正如预期的那样,免疫细胞是最不常见的(<15%的细胞;图1C,代表性比例,肿瘤组)。转移部位的免疫细胞比原发性肿瘤部位少。差异最大的是间质细胞,肝脏样本的比例最低(8%),骨骼样本的比例最高(47%)。原发性肿瘤样本的代表性图像如图1D所示。
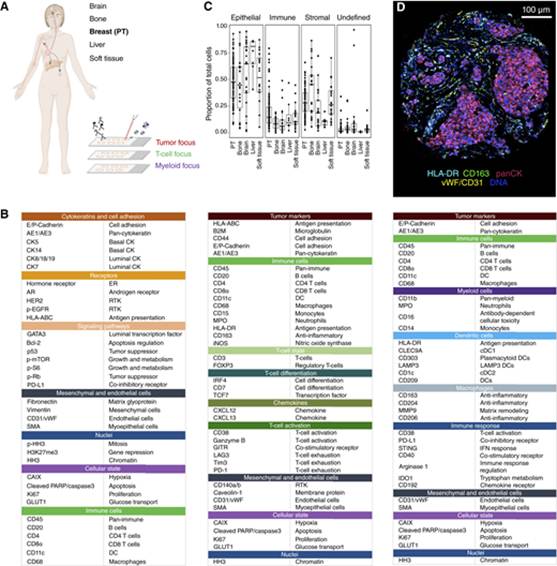
Figure 1原始乳腺肿瘤和匹配转移瘤的高度多重单细胞图谱
2. 转移瘤与配对原发性肿瘤具有相同的肿瘤细胞表型,但组成不同
为了比较匹配的原发性肿瘤和转移瘤的肿瘤细胞表型和组成,我们首先将所有肿瘤细胞分为20个表型簇(图2A,左)。与我们之前在不同乳腺癌队列中的工作一致,我们确定了预期的表型,例如CK 5+和CK 14+基础细胞、死亡细胞、Ki 67+增生细胞、HER 2高细胞以及以GATA 3为特征的各种腔细胞表型、不同水平的E-钙粘蛋白、细胞角蛋白(CK 7、CK 8/18/19)和激素受体表达。有趣的是,转移性疾病诊断相对较早的患者的原发性肿瘤富含缺氧和EGFR+肿瘤表型。在所有患者中,所有20种表型均在原发部位和转移部位发现(图2A,右)。对于大多数患者来说,在初次和转移部位存在的肿瘤细胞表型方面存在高度重叠,但不同部位之间的比例差异很大(图2B)。尽管匹配样本之间的一些表型存在差异,其中增生性Ki 67 + pRB+表型富含转移,基础表型以及腔型和抗细胞死亡表型富含原发性肿瘤(图2C),但这些表型仅代表两种样本类型的一小部分。因此,匹配的原代和转移样本显示存在相似的肿瘤细胞表型,但不同部位的表型比例有所不同。
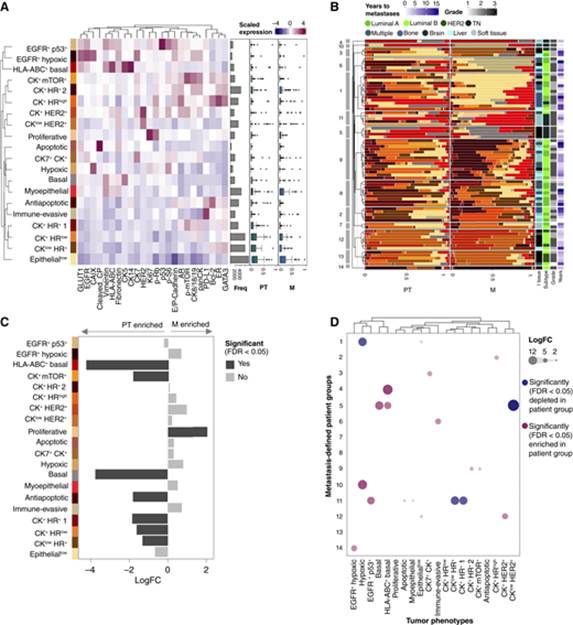
Figure 2转移瘤与配对原发性肿瘤具有相同的肿瘤细胞表型,但组成不同
我们观察到通过对转移样本进行聚集可以识别出14个不同的患者组。这些组中的大多数是由几名具有相似肿瘤表型组成的患者组成的,但也有一些是单身患者,代表表现出独特特征的患者。大多数患者群体以特定的肿瘤细胞表型为主,这与之前的发现一致。这些患者组跨越分子亚型和转移组织部位,差异丰度测试确定了各组中富集或耗尽的肿瘤表型(图2D)。匹配的原代和转移样本之间最丰富的肿瘤细胞表型经常不同(图3A)。为了进一步研究这一点,我们定量比较了原发部位和转移部位的肿瘤表型组成。除了HER 2阳性脑样本外,原发样本和转移样本的总体表型组成相似(图3B)。匹配样本中的成分比较还表明,转移样本与匹配而不是随机原发性肿瘤更相似,尽管可以观察到相当大的扩散(图3C,左)。这进一步与匹配样本肿瘤细胞中平均标志物表达的弱正相关(R范围为0.3至0.6)一致。这些分析表明,患者转移部位的表型组成与匹配原发性肿瘤表型组成的相似程度有所不同,但匹配对的组成总体上比随机非匹配对更相似。匹配的转移性和原发性肿瘤样本的组成相似性并不显着取决于转移部位(Kruskal-Wallis检验; P ½ 0.141;图3C,右),我们也通过其他相似性指标证实了这一点。肿瘤分子亚型确实显示出较小的影响;然而,HER 2阳性和TN亚型在匹配样本之间表现出比腔亚型更高的成对相似性。与此一致,在原发性肿瘤或转移中单独分析的肿瘤细胞组成在相同分子亚型的样本中再次相似,但在具有相同转移部位的样本中则不同。此外,肿瘤细胞的平均单细胞标志物表达不取决于转移部位(大脑除外)。原发性肿瘤在表型上并不比转移样本更不均匀,并且更不均匀的原发性肿瘤并没有引发更不均匀的转移。我们的肿瘤单细胞表型分析的结论独立于我们允许的集群数量(数据未显示),并且不会随着不同的集群方法而发生实质性变化。
总之,我们的高度多重单细胞分析表明,乳腺癌患者匹配的原代和转移样本之间通常存在肿瘤细胞表型,但匹配样本之间表型的比例差异很大。尽管匹配样本的整体肿瘤细胞组成比偶然预期的更加相似,但个体患者的组成相似性各不相同。我们发现了一种富含转移的单一表型,但不是转移特有的,还有几种富含原发性肿瘤。分子亚型(而不是转移部位)与转移样本中的肿瘤细胞组成相关。最后,匹配样本之间表型相似性的程度与转移部位无关,这表明肿瘤细胞表型不存在位点依赖性选择压力。
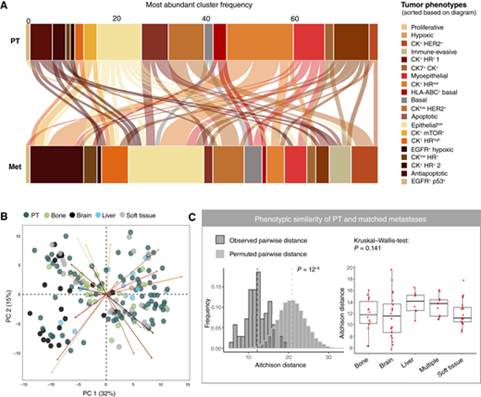
Figure 3转移瘤与配对原发性肿瘤具有相同的肿瘤细胞表型,但组成不同
3. 原发性肿瘤和转移性免疫微环境的表型和空间图
为了能够比较原发性肿瘤和转移的免疫格局,我们确定了广泛的免疫细胞类型类别(即T细胞、B细胞、NK细胞、骨髓细胞和浆细胞)使用T细胞面板数据集(图4A)。然后我们对所有T细胞进行子集化,进行聚集仅使用T细胞相关标记物来识别CD 4 + T辅助细胞和CD 8+细胞毒性T细胞,并基于相似的表达谱将它们元聚集成功能亚型(图4 B)。在CD 8+区室中,我们发现了一种耗尽的亚型(PD-1、Tim 3、LAG 3和颗粒酶B)和一种激活的亚型(CD 38、GITR和颗粒酶B)等。在CD 4+区室中,我们观察到了两种调节性T细胞(Treg)亚型,即发育具有较低FOXP 3表达的Treg和具有GITR共表达的激活Treg,以及具有高CXCL 13和PD-1表达的T毛囊辅助细胞(Tfh)亚型。CD 4+和CD 8+区室中的TCF 7高细胞可能代表自我更新的前体T细胞。尽管我们对这些亚型的识别是基于对检测到的标记物的手动注释,但当可能使用共享标记物进行评估时,它们与基于全基因组数据识别的表型良好一致。
为了研究骨髓细胞表型,我们采用了类似的方法。我们根据CD 163的表达鉴定了骨髓细胞,CD 68、HLA-DR、CD 11 c和CD 16。接下来,我们仅根据骨髓相关标志物的表达水平对骨髓细胞进行聚集(图4C和D)。我们手动注释了MMP 9+巨噬细胞、CREC 9A+经典DC 1型(cDC 1)以及具有不同水平CD 204、CD 163、CD 209和CD 206的各种巨噬细胞群,表明具有免疫调节或促炎症功能。揭示了不同口味的额外骨髓细胞,包括PD-L1+缺氧骨髓细胞亚群、免疫调节IDO+骨髓细胞和免疫抑制ARG 1+骨髓细胞。由于骨髓细胞和巨噬细胞的多样性和可塑性,我们没有元集群这些细胞类型;我们进一步指出,对标记物表达的任何功能解释都可能是不完美的。
在所有组织部位都可以检测到广泛的免疫细胞表型。有趣的是,晚期复发患者的Tfh细胞、B细胞和IDO+骨髓细胞富含原发性肿瘤,而早期复发患者的PD-L1+缺氧骨髓细胞。因此,我们的分析提供了匹配的原发和转移样本中乳腺癌肿瘤免疫微环境的高分辨率组成图。
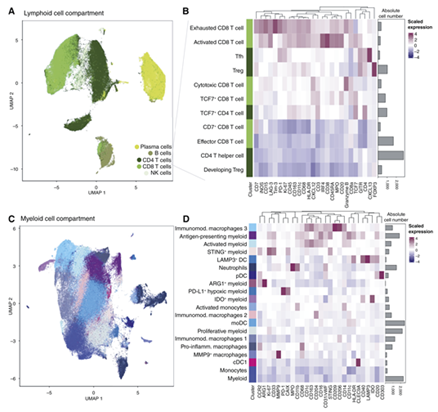
Figure 4 原发性肿瘤和转移性免疫微环境的表型和空间图
4. 转移性肿瘤的免疫环境是组织依赖性的,但受原发性肿瘤的影响
我们比较了原发性肿瘤和转移样本中免疫微环境的组成。我们仅比较了肿瘤内区域,因为转移样本主要是肿瘤内,并且原发性肿瘤的边缘和肿瘤内区域之间存在免疫组成差异。原发性肿瘤在广泛的免疫细胞类型丰度方面与转移性样本相似(图5A)。然而,配对差异丰度测试显示原发性肿瘤中B细胞和T细胞富集,转移部位骨髓细胞富集(图5 B),反映了转移处骨髓细胞密度稳定,但淋巴细胞密度相对于原发性肿瘤降低。在总体免疫细胞组成中,转移样本与匹配的原发性肿瘤并不比与随机原发性肿瘤更相似(图5C)。初发性肿瘤和转移性肿瘤的T细胞亚型丰度不同(图5D),由转移瘤中抗原经历表型(耗尽的CD 8 T细胞和Tfh细胞)和细胞毒性CD 8 + T细胞相对于匹配的原发性肿瘤更高比例驱动,以及CD 7 + CD 8 T细胞和发育中的Treg表型比例较低(图5E),也与细胞密度分析一致。
与总体免疫组成相反,匹配样本的T细胞亚型比随机比较更相似(图5 F)。同样,根据骨髓细胞表型簇丰度将原发部位和转移部位分别分组(图5G)。这是由于转移性样本中ARG 1+骨髓细胞、MP 9+巨噬细胞和中性粒细胞等比例富集而导致的,而原发性肿瘤富含LAMP 3 + DC、CD 204高/CD 163低免疫调节巨噬细胞和cDC 1(图5 H);再次,骨髓细胞亚型的细胞密度显示出类似的趋势。至于T细胞亚型,匹配样本的骨髓细胞表型簇也比随机预期的更加相似(图5 I)。
我们进一步询问转移性免疫微环境是否存在组织依赖性特征。原始肿瘤和转移性肿瘤之间的差异丰度测试显示,位点之间T细胞亚型几乎没有差异(图6A)。耗尽的CD 8 T细胞在大脑、肝脏和软组织样本中富集,而骨骼样本显示CD 7 + CD 8 + T细胞耗尽。此外,脑和软组织样本中细胞毒性CD 8 + T细胞比例较高,转移至肝脏和脑的原发性肿瘤中发生TMF的比例较高。尽管这些样本中T细胞密度总体降低,但脑转移瘤中细胞毒性和耗尽的CD 8 T细胞仍按比例增加,这与脑转移瘤中大多数其他T细胞亚型密度较低的趋势一致。相对于原发性肿瘤,骨转移瘤中大多数T细胞类型的密度也大幅降低。骨髓表型簇在不同的转移部位显示出许多不同的表型簇的富集(图6 B),例如,骨和脑样本中的MMP9+巨噬细胞、脑样本中的中性粒细胞和PD-L1+缺氧骨髓细胞的富集,而相应的原发性肿瘤富含LAMP 3 + DC、单核细胞来源的DC和cDC 1。唯一的例外是软组织样本,其中匹配的转移性和原发性肿瘤样本之间没有骨髓簇显示出显着差异。这些比例差异与不同部位骨髓细胞亚型密度的趋势一致。无论是一般免疫细胞类型、T细胞亚型还是骨髓簇,原发性肿瘤和转移样本之间的组成相似性都不取决于转移组织部位(图6C)。
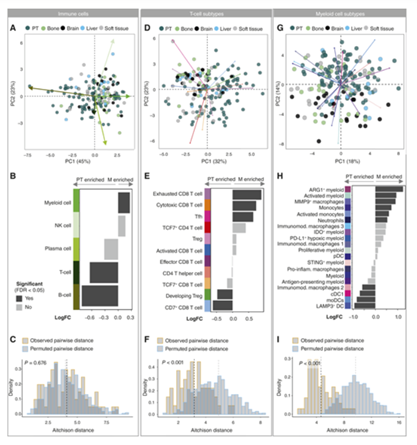
Figure 5 转移性肿瘤的免疫环境是组织依赖性的,但受原发性肿瘤的影响
最后,我们专门研究了转移性肿瘤中的免疫特征。组织部位之间的T细胞组成显着差异,但组中心位置很接近(图6D,左)。这些差异是由软组织与脑或骨样本之间的差异驱动的,主要由TCF 7 + CD 4 T细胞和TMF解释(图6D,右)。相比之下,骨髓细胞表现出强烈的组织依赖性组成特征(图6 E),所有组织部位之间存在显着差异。免疫细胞组成在骨髓细胞的分子亚型之间也显着差异,但在T细胞区室中则没有。此外,在对每个分子亚型转移瘤之间的免疫细胞表型丰度和所有转移瘤的平均丰度的成对比较中(图6 F),腔A转移瘤显示Tfh、单核细胞、STING+ DC和中性粒细胞的存在与其他亚型相比显着减少。相比之下,Her 2亚型与MMP 9+巨噬细胞相关。尽管由于每个分子亚型组内的患者数量较少,因此应谨慎解释这些结果,但它们与先前的原发性肿瘤研究一致。我们还观察到转移性免疫微环境的差异,这取决于我们定义的患者组(图2B)的肿瘤表型组成(图6 G)。特别有趣的是,第6组患者富含免疫逃避性肿瘤表型,PD-L1缺氧和IDO+骨髓细胞也显着富集,并且TSYS耗尽。
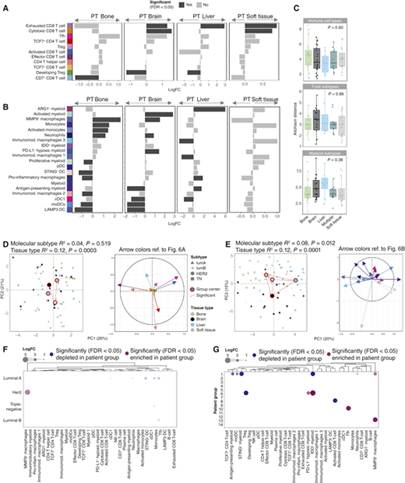
Figure 6 转移性肿瘤的免疫环境是组织依赖性的,但受原发性肿瘤的影响
总之,与匹配的原发性肿瘤相比,转移部位的T细胞丰度降低,但Tfh和耗尽的CD 8 + T细胞的相对比例增加。骨髓细胞隔室显示出强烈的组织特异性组成特征,在不同的转移组织部位富含几种不同的巨噬细胞表型。同样,分子亚型与DC表型、Tfh、中性粒细胞和MMP9+巨噬细胞的不同丰度相关。
5. 原发性肿瘤和转移瘤共享空间肿瘤和免疫细胞邻域
最后,我们在空间上对原发性和转移性肿瘤中的免疫细胞亚型进行了描述。首先,我们计算了肿瘤和间质内T细胞和骨髓细胞亚型的相对富集度整个队列中的每张图像中的面具。肿瘤面具中唯一富含的T细胞亚型是CD 8+耗尽的T细胞,正如预期的那样,因为慢性T细胞受体参与导致T细胞耗尽(图7A,左)。只有少数骨髓细胞团在肿瘤面具中富集,包括PD-L1+缺氧骨髓细胞和STING+骨髓细胞(图7A,右)。在间质掩模中富集最高的骨髓细胞亚型也在边缘图像中富集。
我们对所有图像中的所有细胞类型进行了空间分析(即,使用所有抗体组生成)。我们使用肿瘤组中的少数标志物来测试原发性肿瘤和转移之间相互作用的变化,这些标志物也在免疫和间质区室中识别关键细胞类型。我们观察到细微的差异,例如,与转移瘤相比,T细胞在原发性肿瘤中表现出明显更大的避免抗细胞死亡肿瘤细胞,但总体而言,变化有限。接下来,我们测试了每个感兴趣区域的细胞类型丰度显着更高或更低的相关性。在这里,我们询问每个小组中识别的细胞类型是否与用其他两个小组染色的连续图像中的其他细胞类型相关。由于分析的数据来自连续的组织切片,因此相关性报告了与图像维度(500 μm & 500 μm)相对应的长度尺度上的空间关系。这些分析表明,原发性肿瘤和转移性肿瘤相关性之间几乎没有显着差异,并且我们主要检测到不同免疫细胞类型之间的变化。值得注意的是,p53+ EGFR+肿瘤细胞表现出高度相关性(即,在原发性肿瘤中与不同的T细胞亚型占据了共同区域,但在转移部位不占据,而许多T细胞亚群与转移部位的ARG 1+骨髓细胞占据了共同区域,但在原发性肿瘤中不占据)。
我们询问转移部位是否包含空间小生境或细胞类型的局部富集,也称为CN,以及这些在转移与原发性肿瘤方面是否不同。因为肿瘤细胞、T细胞和通过对连续切片染色来分析骨髓细胞,我们分别对每一个进行CN分析。我们鉴定了12种肿瘤CN,例如,CN 3具有增生性、EGFR+缺氧和CK 7+肿瘤细胞,CN 7由缺氧细胞组成,CN 8包括免疫逃避和低内皮表型(图7 B,左。激素受体高CN 10在相对于原发性肿瘤的多个转移部位富集,但大脑和肝脏除外。脑转移瘤中的CN 10被耗尽,而富含缺氧CN 7。除了大脑之外,我们在表皮CNs中观察到了很少有特定部位的模式。
我们的分析检测到14种T细胞CN(图7 B,中间),每个都包含T细胞亚型的独特组合,例如,CN 1中产生CD 8和GSK的CD 8 + T细胞耗尽,CN 3中产生TCF 7-高CD 4+和CD 8 + T细胞,CN 4中产生CD 8 + T细胞激活。比较原发部位和转移部位之间每个T细胞CN的丰度显示几乎没有差异(图7 B,中间)。例外是CN 6(主要含有发育中的TSYS)和CN 9(CD 8+效应细胞和CD 8 +/CD 7 + T细胞),它们的骨转移相对于原发性肿瘤被耗尽(图7 C,中间)。总体而言,T细胞的空间结构在原发性肿瘤和转移性肿瘤之间相似。
我们鉴定了9种骨髓细胞CN(图7 B,右),其中脑转移瘤中的CN 7被耗尽,含有DC,并富含CN 8,主要含有激活的骨髓细胞(图7 B,右)。这些结果与脑转移瘤中这些表型与原发性肿瘤的差异丰度一致(图6 B),表明这些表型不仅改变了丰度,而且形成了这些样本之间丰度也不同的空间CN。我们还观察到,与原始组织相比,骨转移瘤富含CN 1,含有中性粒细胞和ARG 1+骨髓细胞。总而言之,我们对原发性和远处转移性肿瘤的空间分析显示,两个肿瘤部位的空间组织总体相似,但特定局部社区的丰富程度存在差异。
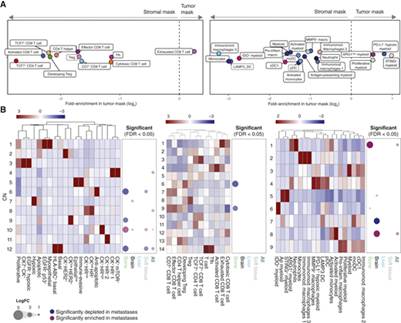
Figure 7 原发性肿瘤和转移瘤共享空间肿瘤和免疫细胞邻域
研究结论:
总而言之,我们以前所未有的详细程度和单细胞分辨率检查了匹配的原始和远处转移性乳腺肿瘤样本中的肿瘤和免疫细胞表型组成。我们证明了患者中转移性疾病的多样性,并确定骨髓细胞是转移部位的主要免疫调节剂,这表明它们可能是转移特异性治疗的潜在目标。我们的数据集提供了一个独特的资源来比较乳腺癌的原始肿瘤和转移性肿瘤。
研究方法:
组装组织芯片,抗体面板,抗体染色,使用 IMC 采集数据,数据预处理和细胞分割,肿瘤-瘤体掩膜,R 中的数据分析,肿瘤、髓系和 T 细胞聚类方法,数据可视化,丰度差异分析,成对距离和表型相似性计算,用于评估组织位点相似性的 PERMANOVA,跨面板细胞类型的相关性分析,细胞邻域分析,统计设计。
参考文献:
Laura Kuett, Alina Bollhagen, Sandra Tietscher, Bettina Sobottka, Nils Eling, Zsuzsanna Varga, Holger Moch, Natalie de Souza, Bernd Bodenmiller; Distant Metastases of Breast Cancer Resemble Primary Tumors in Cancer Cell Composition but Differ in Immune Cell Phenotypes. Cancer Res 1 January 2025; 85 (1): 15-31. https://doi.org/10.1158/0008-5472.CAN-24-1211






